1.概述
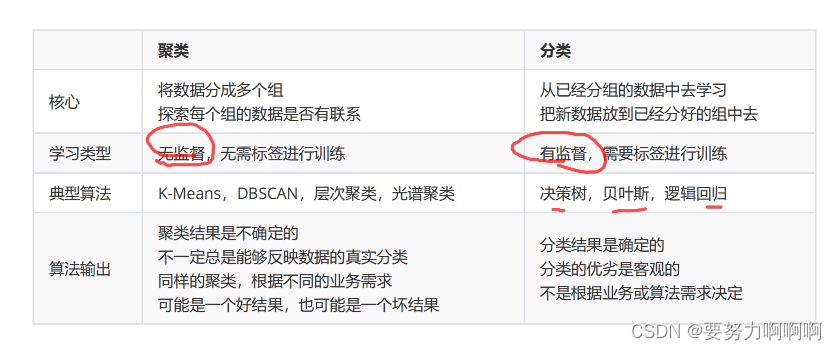
聚类算法又叫做‘无监督学习’,其目的是将数据划分成有意义或有用的组(或簇)。
2.KMeans
关键概念:簇与质心
KMeans算法将一组N个样本的特征矩阵X划分为K个无交集的簇,直观上来看是簇是一组一组聚集在一起的数据,在一个簇中的数据就认为是同一类。簇就是聚类的结果表现。
簇中所有数据的均值 通常被称为这个簇的“质心”(centroids)。在一个二维平面中,一簇数据点的质心的横坐标就是这一簇数据点的横坐标的均值,质心的纵坐标就是这一簇数据点的纵坐标的均值。同理可推广至高
维空间。
3.簇内误差平方和的定义和解惑
我们认为,被分在同一个簇中的数据是有相似性的,而不同簇中的数据是不同的.因此我们追求“组内差异小,组间差异大”。聚类算法也是同样的目的,我们追求“簇内差异小,簇外差异大”。而这个“差异“,由样本点到其所在簇的质心的距离来衡量。对于一个簇来说,所有样本点到质心的距离之和越小,我们就认为这个簇中的样本越相似,簇内差异就越小。
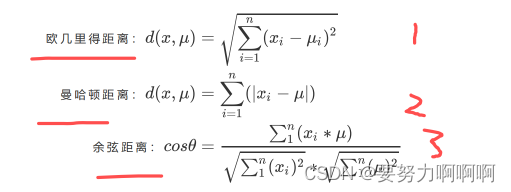
n代表每一个样本的特征个数,x代表样本,miu代表簇中的质心。

其中,m为一个簇中样本的个数,j是每个样本的编号。这个公式被称为簇内平方和(cluster Sum of Square),又叫做Inertia。而将一个数据集中的所有簇的簇内平方和相加,就得到了整体平方和(Total Cluster Sum of
Square),又叫做total inertia。Total Inertia越小,代表着每个簇内样本越相似,聚类的效果就越好。因此KMeans追求的是,求解能够让Inertia最小化的质心。
4.sklearn.cluster.KMeans
4.1重要参数n_clusters
n_clusters是KMeans中的k,表示着我们告诉模型我们要分几类。这是KMeans当中唯一一个必填的参数,默认为8类,但通常我们的聚类结果会是一个小于8的结果。
简单聚类
from sklearn.datasets import make_blobs
import matplotlib.pyplot as plt
#首先简单的进行一次聚类
'''
步骤如下:
1。首先使用sklearn中的方法来自定义自己的数据
2。画出散点图,顺带画出已经分好簇的散点图,使用plot,同时也对原始的数据已经分好簇的数据进行画图描述使用四种颜色
3。基于这个分布,使用kmeans来进行聚类,需要导入聚类中的kmeans
设置要分好的类别,然后对其进行拟合,预测的结果使用label可以看到预测出来的结果.
使用sluster_centers_可以输出每一个簇的分类点的位置
使用inertia_可以查看总的欧氏距离是多少
4。分别猜测其中有3,4,5,6四个簇,通过簇内平方和来判断哪一个数目是最好的。
'''
#自己创建数据集,生成的数据大小是500*2,500个数据,每一个数据有两个特征,分别对应的是x1,x2
X,Y = make_blobs(n_samples=500,n_features=2,centers=4,random_state=1)
fig,ax1 = plt.subplots(1)
ax1.scatter(
X[:,0],
X[:,1],
marker = 'o',
s=8
)
color = ['red','pink','orange','gray']
# fig,ax2 = plt.subplots(1)
# for i in range(4):
# ax2.scatter(
# X[Y==i,0],
# X[Y==i,1],
# s=8,
# c=color[i]
# )
plt.show()
from sklearn.cluster import KMeans
n_clusters = 3
cluster = KMeans(n_clusters=n_clusters,random_state=0).fit(X)
n_clusters = 4
cluster = KMeans(n_clusters=n_clusters,random_state=0).fit(X)
n_clusters = 5
cluster = KMeans(n_clusters=n_clusters,random_state=0).fit(X)
n_clusters = 6
cluster = KMeans(n_clusters=n_clusters,random_state=0).fit(X)
y_pred = cluster.labels_#聚类之后分好的类别。
print(y_pred)
pre = cluster.fit_predict(X)
#print(pre == y_pred)
inertia = cluster.inertia_
centroid = cluster.cluster_centers_
print(inertia)
color = ['red','pink','orange','gray','green','yellow']
fig , ax3 = plt.subplots()
for i in range(n_clusters):
ax3.scatter(X[y_pred==i,0],X[y_pred==i,1]
,marker='o'
,s=8
,c=color[i]
)
ax3.scatter(centroid[:,0]
,centroid[:,1]
,marker='x'
,s=15
,c='black')
plt.show()
4.2 聚类算法的模型评估指标
不同于分类模型和回归,聚类算法的模型评估不是一件简单的事。
1.在分类中,有直接结果(标签)的输出,并且分类的结果有正误之分,所以我们使用预测的准确度,混淆矩阵,ROC曲线等等指标来进行评估,但无论如何评估,都是在”模型找到正确答案“的能力。
2.回归中,由于要拟合数据,我们有SSE均方误差,有损失函数来衡量模型的拟合程度。但这些衡量指标都不能够使用于聚类。
4.3.1 当真实标签已知的时候
如果拥有真实标签,我们更倾向于使用分类算法。但不排除我们依然可能使用聚类算法的可能性。如果我们有样本真实聚类情况的数据,我们可以对于聚类算法的结果和真实结果来衡量聚类的效果。常用的有以下三种方法:
互信息分:取值范围在(0,1)之中越接近1;聚类效果越好;在随机均匀聚类下产生0分;
V-meature:取值范围在(0,1)之中越接近1;越接近1,聚类效果越好
调整兰德系数:取值范围在(0,1)之中越接近1;越接近1越好
4.3.2 当真实标签未知的时候:轮廓系数
其中轮廓系数是最常用的聚类算法的评价指标。它是对每个样本来定义的,它能够同时衡量:
1)样本与其自身所在的簇中的其他样本的相似度a,等于样本与同一簇中所有其他点之间的平均距离
2)样本与其他簇中的样本的相似度b,等于样本与下一个最近的簇中的所有点之间的平均距离
根据聚类的要求”簇内差异小,簇外差异大“,我们希望b永远大于a,并且大得越多越好。

很容易理解轮廓系数范围是(-1,1),其中值越接近1表示样本与自己所在的簇中的样本很相似,并且与其他簇中的样本不相似,当样本点与簇外的样本更相似的时候,轮廓系数就为负。当轮廓系数为0时,则代表两个簇中的样本相似度一致,两个簇本应该是一个簇。可以总结为轮廓系数越接近于1越好,负数则表示聚类效果非常差。
如果一个簇中的大多数样本具有比较高的轮廓系数,则簇会有较高的总轮廓系数,则整个数据集的平均轮廓系数越高,则聚类是合适的。如果许多样本点具有低轮廓系数甚至负值,则聚类是不合适的,聚类的超参数K可能设定得太大或者太小。
4.3.3 当真实标签未知的时候:Calinski-Harabaz Index
除了轮廓系数是最常用的,我们还有卡林斯基-哈拉巴斯指数(Calinski-Harabaz Index,简称CHI,也被称为方差比标准),戴维斯-布尔丁指数(Davies-Bouldin)以及权变矩阵(Contingency Matrix)可以使用。
在这里我们重点来了解一下卡林斯基-哈拉巴斯指数。Calinski-Harabaz指数越高越好。对于有k个簇的聚类而言,Calinski-Harabaz指数s(k)写作如下公式:
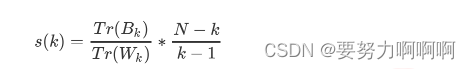
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_samples, silhouette_score
import matplotlib.pyplot as plt
import matplotlib.cm as cm
import numpy as np
from sklearn.datasets import make_blobs
n_clusters = 5
X,Y = make_blobs(n_samples=500,n_features=2,centers=4,random_state=1)
fig, (ax1, ax2) = plt.subplots(1, 2)
fig.set_size_inches(18, 7)#是用来设置生成图片大小的
ax1.set_xlim([-0.1, 1])#设置x坐标轴的
ax1.set_ylim([0, X.shape[0] + (n_clusters + 1) * 10])
clusterer = KMeans(n_clusters=n_clusters, random_state=10).fit(X)
cluster_labels = clusterer.labels_#分出来的类别
silhouette_avg = silhouette_score(X, cluster_labels)
print("For n_clusters =", n_clusters,"The average silhouette_score is :", silhouette_avg)
sample_silhouette_values = silhouette_samples(X, cluster_labels)
y_lower = 10
for i in range(n_clusters):
ith_cluster_silhouette_values = sample_silhouette_values[cluster_labels == i]
ith_cluster_silhouette_values.sort()#这里是为了能够从小到大向上排列
size_cluster_i = ith_cluster_silhouette_values.shape[0]
y_upper = y_lower + size_cluster_i#这里是留出来上下的空间
color = cm.nipy_spectral(float(i)/n_clusters)#设置颜色使用的。
'''
进行颜色的填充。只不过是填充的方向不同。
fill_between是进行y轴方向上的填充,也就是竖直方向的填充
fill_betweenx是进行x轴方向上的填充,也就是水平方向的填充
'''
# 第一个参数是范围
# 第二个参数表示填充的上界
# 第三个参数表示填充的下界,默认0
ax1.fill_betweenx(np.arange(y_lower, y_upper),ith_cluster_silhouette_values,facecolor=color,alpha=0.7)#进行颜色填充的
ax1.text(-0.05, y_lower + 0.5 * size_cluster_i, str(i))#精准的在图上的某一个点位进行写字。前面两个是坐标信息,后面是内容。
y_lower = y_upper + 10
ax1.set_title("The silhouette plot for the various clusters.")
ax1.set_xlabel("The silhouette coefficient values")
ax1.set_ylabel("Cluster label")
ax1.axvline(x=silhouette_avg, color="red", linestyle="--")#ax1.axvline设置一条中轴线。所有样本的轮廓系数。
ax1.set_yticks([])
ax1.set_xticks([-0.1, 0, 0.2, 0.4, 0.6, 0.8, 1])
colors = cm.nipy_spectral(cluster_labels.astype(float) / n_clusters)
ax2.scatter(X[:, 0], X[:, 1] ,marker='o',s=8,c=colors )
centers = clusterer.cluster_centers_
# Draw white circles at cluster centers在簇中画出来质心
ax2.scatter(centers[:, 0], centers[:, 1], marker='x',
c="red", alpha=1, s=200)
ax2.set_title("The visualization of the clustered data.")
ax2.set_xlabel("Feature space for the 1st feature")
ax2.set_ylabel("Feature space for the 2nd feature")
plt.suptitle(("Silhouette analysis for KMeans clustering on sample data ""with n_clusters = %d" % n_clusters), fontsize=14, fontweight='bold')
plt.show()
4.3 重要参数init & random_state & n_init:初始质心怎么放好?
在K-Means中有一个重要的环节,就是放置初始质心。如果有足够的时间,K-means一定会收敛,但Inertia可能收敛到局部最小值。是否能够收敛到真正的最小值很大程度上取决于质心的初始化。init就是用来帮助我们决定初始化方式的参数。
init:可输入"k-means++“,“random"或者一个n维数组。这是初始化质心的方法,默认"k-means++”。输入"kmeans++”:一种为K均值聚类选择初始聚类中心的聪明的办法,以加速收敛。
random_state:控制每次质心随机初始化的随机数种子
n_init:整数,默认10,使用不同的质心随机初始化的种子来运行k-means算法的次数。
4.4 重要参数max_iter & tol:让迭代停下来
当质心不再移动,Kmeans算法就会停下来。但在完全收敛之前,我们也可以使用max_iter,最大迭代次数,或者tol,两次迭代间Inertia下降的量,这两个参数来让迭代提前停下来。
max_iter:整数,默认300,单次运行的k-means算法的最大迭代次数
tol:浮点数,默认1e-4,两次迭代间Inertia下降的量,如果两次迭代之间Inertia下降的值小于tol所设定的值,迭
代就会停下
random = KMeans(n_clusters = 10,init="random",max_iter=10,random_state=420).fit(X)
y_pred_max10 = random.labels_
silhouette_score(X,y_pred_max10)
random = KMeans(n_clusters = 10,init="random",max_iter=20,random_state=420).fit(X)
y_pred_max20 = random.labels_
silhouette_score(X,y_pred_max20)






![[C++]string类模拟实现](https://img-blog.csdnimg.cn/c60fc66ba14e48b3837bd944fb19fccd.png)