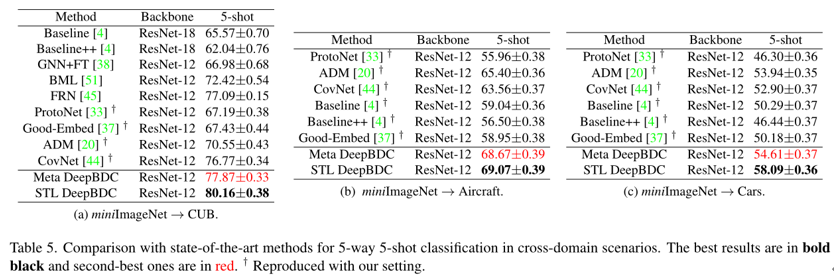
题目:AlphaFold accelerates artificial intelligence powered drug discovery: efficient discovery of a novel CDK20 small molecule inhibitor
文献来源:Chem. Sci., 2023, 14, 1443
代码:是一个很大的流程
https://www.pandaomics.com/
https://www.chemistry42.com/
https://github.com/insilicomedicine/ipanda
https://github.com/insilicomedicine/GENTRL
https://github.com/insilicomedicine/TRIP
1.背景介绍
蛋白质的三维结构与其在细胞中的功能和氨基酸导致的生物学影响密切相关。蛋白质结构是研究基因-疾病关联和作用模式(MoA)的一种通用工具,可以用于评估治疗靶点的可用药性。基于结构的药物发现(SBDD)一直是识别hit molecules和进行先导物优化的主要方法,该方法的实现需要一个靶点的三维结构。但是蛋白质的预测是一个非常困难的事。直到AlphaFold才有了转机。目前,AlphaFold模型可以用于帮助通过晶体学和低温电子显微镜确定实验结构,指导PINK1的功能研究、帮助识别致病突变,以及探索蛋白-蛋白相互作用。
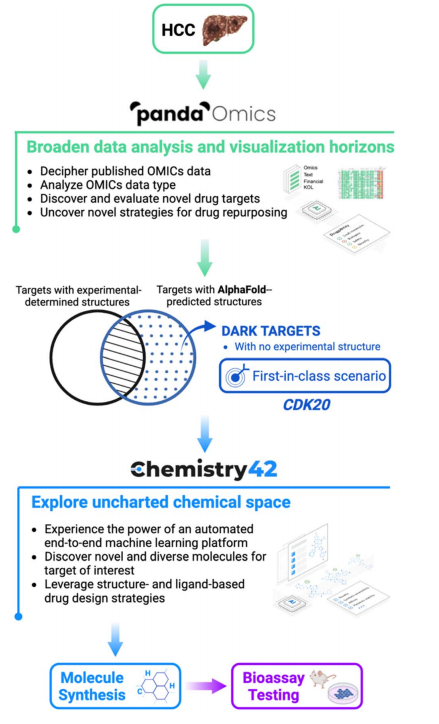
图1 AlphaFold与 Insilico Medicine端对端结合结合,以及人工智能药物发现平台 Insilico Medicine,在肝细胞癌药物发现中实现靶点选择和hit生成到hit鉴定。这个新的治疗靶点缺乏实验结构信息,是通过alphafold预测获得的。这种靶点代表了治疗HCC的一类新靶点。
在这项工作中,作者通过将AlphaFold预测的蛋白质结构与端到端人工智能药物发现平台 Insilico Medicine结合,快速识别出一个新靶点的从头分子。图1展示了整个工作流程。因为HCC(hepatocellular carcinoma)与肝癌和有效治疗的缺乏息息相关,所以HCC作为目标。总的来说,通过对HCC的10个数据集的文本和组学数据的分析,PandaOmics提供了含有20个靶点的列表。经过多种筛选之后,最后CDK20(Cyclindependent kinase 20)成为了目标单点。选择CDK20的原因是因为这个靶点与疾病有着紧密的联系,但是实验结构信息较少,而且这三年内关于该靶点的临床药物或者审批药物也比较少。利用AlphaFold预测的CDK20结构,Chemistry42平台进行基于结构的化合物生成,生成8918个分子,进行分子对接和聚类之后,7个化合物进行合成和生物测试。其中,化合物ISM042-2-001在CDK20激酶结合试验中显示其Kd值为9.2 ± 0.5 mM(n = 3)。通过Chemistry42和AlphaFold预测的蛋白质结构,作者只花了30天就完成了第一次hit。然后,预测的结合模式指导第二轮化合物生成、合成和测试,从而得到了更多具有纳摩尔抑制能力的分子ISM042-2-048。目前,这项工作是首次报道的成功地利用AlphaFold预测的蛋白质结构来识别早期药物发现的新靶点的新分子。
2.靶点的筛选和鉴别
肝细胞癌(HCC)是肝癌的主要类型,约占患者总数的75%。由于世界各地区的预后都很差,肝癌的发病率与死亡率非常接近。
PandaOmics是一个自动药物发现的人工智能引擎,旨在加速和优化药物发现早期阶段的关键步骤。该生物计算平台结合了用于数据分析、可视化和解释的生物信息学方法与高级多模态深度学习方法,可以用于靶点的鉴别。PandaOmics的治疗靶点以及生物标志系统的构建是在多重分数的基础上形成的。这些分数来自于与基因和目标疾病有关的文本/OMICs数据。文本的倾向挑出那些在文献中广泛出现的基因。而OMICs为基础的分数则相反,根据差异表达、基因变异、相互作用组拓扑结构、信号通路扰动分析算法、基因敲除/过表达实验等方法来探索基因与疾病分子上的联系。这种方法允许用户获得在常见的一般知识或简单的生物信息学分析中可能不明显的隐藏假设。人工智能工具有利于揭示一些靶点假设。总体评分方法对特定疾病的靶点候选物进行排序,然后根据其新颖性、小分子和抗体的可及性、安全性、组织特殊性、晶体结构可用性和主要生物结构进行筛选。
PandaOmics平台的另一个特性是,它能够将来自不同实验的数据合并成一个单一的元分析,并利用来自所有数据集的见解结合在一起,以实现精确的目标优先级。作者为HCC的10个数据集(1133个疾病样本和674个健康对照)组成的每种相关疾病创建了元分析。对靶点候选物进行排序之后,PandaOmics会进行筛选最后获得最后可能符合目标疾病场景特征的一个靶点集合。这些靶点没有结晶实验结构但是具有AlphaFold模型给出的结构信息。最后在人为选择的前20个靶点候选物中,CDK20作为HCC疾病条件下的最好靶点被选择。然后这个靶点信息被输入到 Chemistry42平台用于自动化地生成小分子抑制剂。
3.CDK20是一个很有前途的癌症治疗靶点
CDK20,又称 cell cycle-related kinase(CCRK),是周期蛋白依赖性激酶家族的最新新成员,因其在多种人体组织中的功能(细胞周期依赖和独立)而受到近年来的广泛关注。CDK20在许多人体组织中都具有相当的翻译水平广泛表达。而且,越来越多的临床前证据表明,CDK20在许多肿瘤细胞系中都存在过度表达。这个结论来自于多种不同类型癌症患者的肿瘤样本:结肠直肠癌、肝细胞癌(HCC)、肺癌和卵巢癌。具体的表达原理可见原文。
4.利用AlphaFold预测的结构用于CDK20的分子生成
到今天为止,尽管针对CDK家族其他成员的抑制剂研究取得了巨大的成功,但报道的CDK20抑制剂数量仍然非常有限(如图2所示)。
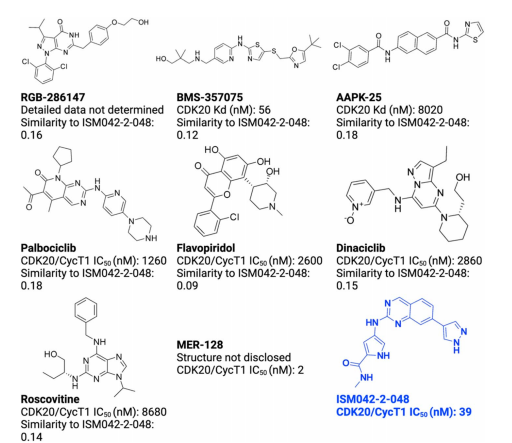
图2 从文献中报道的CDK20抑制剂和本文中发现的新型抑制剂ISM042-2-048以及利用RDKit从Morgan指纹中计算出所报告的分子与ISM042-2-2-048的谷本相似性。
图3描述了Chemisty42平台上利用SBDD方法来生成和预测CDK20抑制剂。
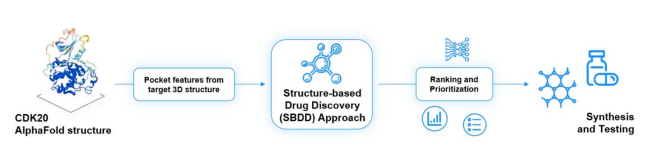
图3 Insilico Medicine公司的CDK20抑制剂生成过程。
当用户将蛋白质结构上传到Chemisty42平台时,程序会自动使用内置的能量基方法来确定假定的结合位点。蛋白质的表面被探针(甲基)均匀地覆盖,而且每个探针与受体原子的非共价相互作用的能量都会被计算出来。能量低于用户定义阈值的探针被聚集在单独的口袋中。每个深度空腔都以口袋体积、表面和深度描述符进行评分。基于这些描述符,Chemisty42提供了一个定义好的结合位点的列表。
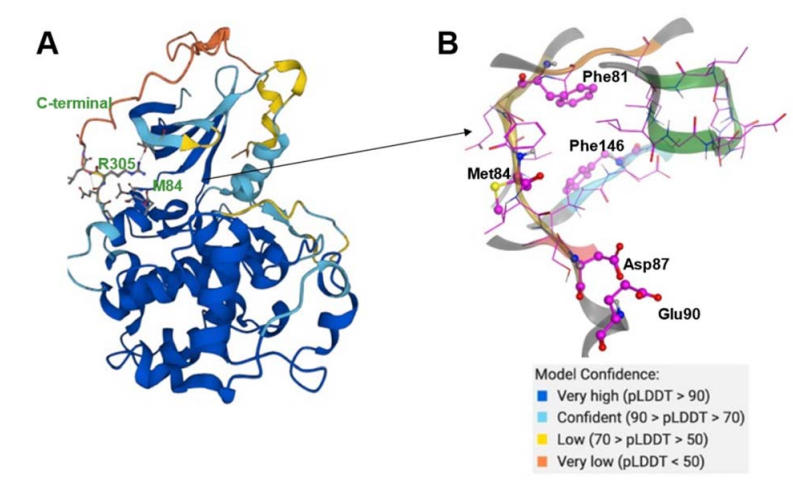
图4 (A) AlphaFold预测的CDK20结构(AF-Q8IZL9- F1-model_v1);CDK20的(B) ATP口袋,具有DFG-in(残基Phe146)构象。Met84是铰链残物。绿色部分是p型环。两个酸中心Asp87和Glu90位于该蛋白的溶剂暴露区域。
AlphaFold(AF-Q8IZL9-F1- model_v1)预测的CDK20结构除了如图4a所示的C端外,总体上具有较高的识别水平。AlphaFold预测结构的C端构象阻断了蛋白质的溶剂暴露区域,C端残基Arg305占据了ATP口袋,如图4(A)所示。C端有一个具有各种构象的灵活环。AlphaFold预测结构中的C端并不利于通过占据ATP口袋来设计抑制剂的构象。因此,C端(Pro303-Gly346)被移除,只有从残基Met1到残基Ile302的结构被用作分子生成的Chemisty42平台输入。Chemisty42平台确定了一个体积为150埃3的浅ATP结合口袋(图 4B)。在铰链残基Met84附近,残基Phe81阻止配体到达后口袋。预测的结合口袋在溶剂化区域有一个DFG-in构象和两个酸性中心(Asp87和Glu90)。平台使用一种基于口袋的生成方法进行生成创新性的分子结构。铰链残基Met84被定义为所需的结合点。来自ATP口袋的其他三维结构信息已被用于引导分子的生成更好地指向目标口袋,如口袋的三维形状、口袋体积和口袋中原子的空间排列。Chemisty42共设计了8918个分子。通过分子对接以及聚类,优先考虑54个铰链核心结构的分子,最终选择7个化合物进行合成。
5.结果和讨论
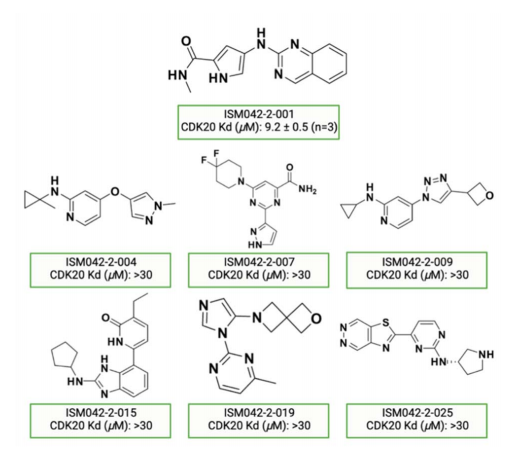
图5 从Chemisty42平台的第一轮生成结果中选择的7个分子化学结构,用于合成和CDK20结合试验中的测试。
图5展示了这7种化合物的化学结构。在所选化合物中,一个化合物ISM042-2-001在CDK20激酶结合试验中Kd值为9.2 ± 0.5 mM(n = 3,一个具有代表性的结合曲线如图6A),在CDK20激酶活性试验中半抑制浓度值为>6000 nM。作者只花了30天的时间就发现了 hit molecule。他们还提出了ISM042-2-001通过分子对接的结合模式,如图6b所示:四个氢键的相互作用用虚线表示。除了与铰链残基Met84形成的两个氢键外,ISM042-2-001还通过酰胺-NH基与残基Leu85相互作用,并通过吡咯-NH基与p-环中的残基Ile10相互作用。或者,酰胺-NH基团或吡咯-NH基团可以与溶剂化区域的两个酸中心Asp87和Glu90形成氢键。
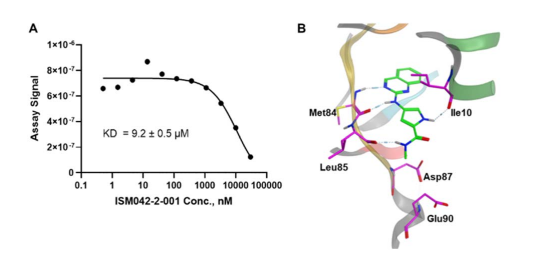
图 6 ISM042-2-001在CDK20激酶结合试验中的代表性结合亲和曲线。数据点以一次实验中重复最低点的平均值表示。在三个独立实验中也得到了相似的结果,KD是三个独立实验的平均值±SD。(B)预测了ISM042-2-001与CDK20的结合姿态。
基于预测的结合姿态和效价数据,作者利用Chemisty42平台进行了第二轮化合物生成。通过两种方法提高新分子的结合亲合力:(1)喹唑啉环上的官能团占据gatekeeper区域附近的疏水口袋;(2)修饰吡咯-2-羧酰胺阳离子进入溶剂化区域,使其与酸性残基Asp87或Glu90作用。采用上述策略,16个生成的分子中有6个被合成并测试。如图7所示,其中ISM042-2-048和ISM042-2-049的结合亲和力分别比ISM042-2-001提高了15倍和24倍,Kd值分别为566.7 ± 256.2 nM和360.0 ± 14.1 nM。ISM042-2-048与CDK20的预测结合模式如图8B所示。基于所提出的结合模式,ISM042-2-048的吡唑基团除了在铰链区域和溶剂区域的具有相互作用外,还与残基Lys33形成氢键,而这解释了其结合亲和力的显著提高。ISM042-2-048与已报道的CDK20抑制剂不同,具有一种新的支架以及低分子相似性,如图2所示。此外,ISM042-2-048对CDK20激酶活性的抑制作用,平均半抑制浓度为33.4 ± 22.6 nM(n = 3),在Huh7中表现出选择性抗增殖活性(半抑制浓度= 208.7±3.3 nM),一个过表达CDK20的HCC细胞系与反筛选细胞系HEK293(半抑制浓度= 1706.7 ± 670.0 nM)的比较如图9所示。下一轮的优化将很快开始,以进一步提高效价,ADME的特性也将对激酶的选择性进行评估。
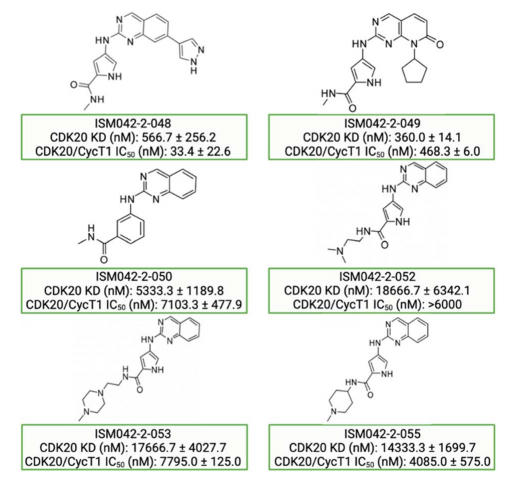
图7 Chemisty42平台第二轮合成的化学结构,这些结构进行了CDK20结合和激酶活性测定实验,对应的数据来自三个独立的实验。
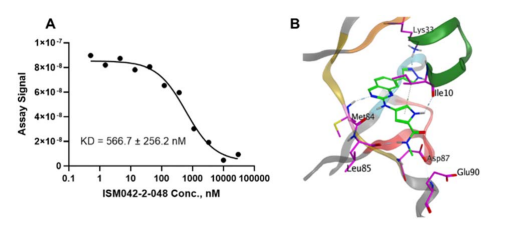
图8 (A)CDK20激酶结合试验中ISM042-2-048的代表性结合亲和曲线。数据点以一次实验中重复最低点的平均值进行表示。在三个独立实验中得到了相似的结果,KD是三个独立实验的平均± SD。(B)预测了CDK20中ISM042-2-048的结合姿态。
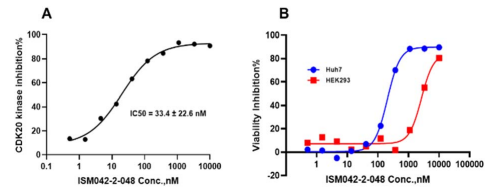
图9 (A)CDK20激酶活性测定中ISM042-2-02-048的代表性剂量-反应曲线。在三个独立的实验中也得到了相似的结果,半抑制浓度是三个独立实验的平均最低点± SD。(B)ISM042-2-048在Huh7细胞系和反筛选细胞系HEK293中的细胞活力曲线。数据点以一次实验中重复最低点的平均值表示。在三个独立的实验中也得到了类似的结果。
6.总结
基于结构的药物发现(SBDD)已经是识别hit molecules和进行先导物优化的主要方法。而预测蛋白质结构的AlphaFold是一个强大的工具,可以预测没有或具有有限的结构信息的新靶点。在此,作者通过使用AlphaFold预测信息作为平台的输入,在30天内实现目标选择、分子生成、化合物合成和生物测试,获得CDK20抑制剂。在合成的7个化合物中,ISM042-2-001在CDK20激酶结合试验中显示Kd值为9.2 ± 0.5 mM(n = 3)。基于初步的SAR,进行了第二轮AI化合物生成,并在30天内合成了6个化合物并测试,发现了首个hit ISM042-2-001。此外,作者发现了一个更好的hit molecule-ISM042-2-048,其平均Kd值为566.7 ± 256.2 nM(n = 3),平均半抑制浓度值为33.4 ± 22.6 nM(n = 3)。此外,ISM042-2-048在CDK20高表达(半抑制浓度= 208.7 ± 3.3 nM)的HCC细胞系Huh7中也表现出良好的抗增殖活性,而在反筛选细胞系HEK293(半抑制浓度= 1706.7 ± 670.0 nM)中作用较弱。这一初步结果表明,设计的CDK20抑制剂并没有诱导任意的细胞毒性,而是对过表达CDK20的HCC细胞有更强的作用,因此可以作为评估该靶点生物学功能的工具分子。该分子的进一步优化以及ADME性质的评价正在进行中。此外,这项工作代表了成功利用 AlphaFold预测蛋白结构获得新靶点hit identication的第一个例子。这种方法的进一步应用也应用于其他靶点类型,如GPCR和E3酶的相关实验正在进行中。
具体的实验细节可看Materials and methods部分。
-------------------------------------------
欢迎点赞收藏转发!
下次见!