甲基化组学全流程分析(适用于27k的甲基化数据)
前面我已经讲过450k和870k EPIC的甲基化芯片的全流程分析教程,分析功能更为完善和强大,今天讲讲甲基化位点探针较少的27k甲基化芯片的分析流程。
我开发了一款本地电脑无限使用的零代码生信数据分析作软图神器电脑软件OmicsTools,欢迎大家使用OmicsTools进行生物医学科研数据分析和作图,该软件件能让大家在不需要任何编程和代码编写的基础上,分析次数没有限制,可以无限使用,让您在自己的电脑上快速进行大量的生信分析和加速大家的科学研究。
我开发的本地电脑OmicsTools 软件在github上的zihaoxingstudy1/OmicsTools仓库中,大家可以下载安装OmicsTools进行各种生信分析和可视化作图。

具体的分析流程
分析步骤简介
- 第一步把甲基化的表达矩阵提取出来
- 第二步拿到甲基化的表达矩阵后,不用再做其它的处理,直接用Limma做差异分析就行了。
- 第三步是甲基化探针注释出基因名称,并跟甲基化差异分析结果文件合并
- 第四步做火山图热图,箱式图这些
- 第五步,go,kegg,gsea通路富集分析等。
我开发了一款本地电脑无限使用的零代码生信数据分析作软图神器电脑软件OmicsTools,欢迎大家使用OmicsTools进行生物医学科研数据分析和作图,该软件件能让大家在不需要任何编程和代码编写的基础上,分析次数没有限制,可以无限使用,让您在自己的电脑上快速进行大量的生信分析和加速大家的科学研究。
我开发的本地电脑OmicsTools 软件在github上的zihaoxingstudy1/OmicsTools仓库中,大家可以下载安装OmicsTools进行各种生信分析和可视化作图。
GEO甲基化27k芯片数据下载教程
1.1根据GEO的GSE数据集编号自动下载和处理GEO数据教程(必须要运行的模块,GEO数据下载要首先运行这个模块)

详细教程地址: https://zhuanlan.zhihu.com/p/708053447
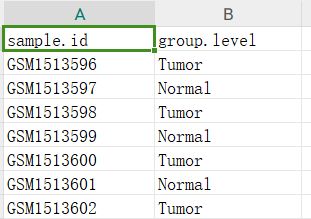
下载GEO数据一定要先运行下这个模块,部分数据集不一定能下载提取出表达矩阵,但是一般能下载到非常完整的样本的注释信息等数据文件,这在对GEO数据集临床信息分析或分组差异分析是非常重要的,当然如果少数GSE数据集遇到没有提取出GSEXXX_sample_info.csv的情况,也可以看看我b站的教学视频,根据GEO网页中的样本编号的分组情况,自建一个这样的分组文件,分组文件一般是长这个样子的,有两列,一列列名叫sample.id,是GSM编号信息,另一列是group.level,存放的是分组信息,当然sample.id这一列的样本名还是要以表达矩阵的样本名为准,这样才能让表达矩阵后面跟样本分组信息按照相同的列名整合在一起:
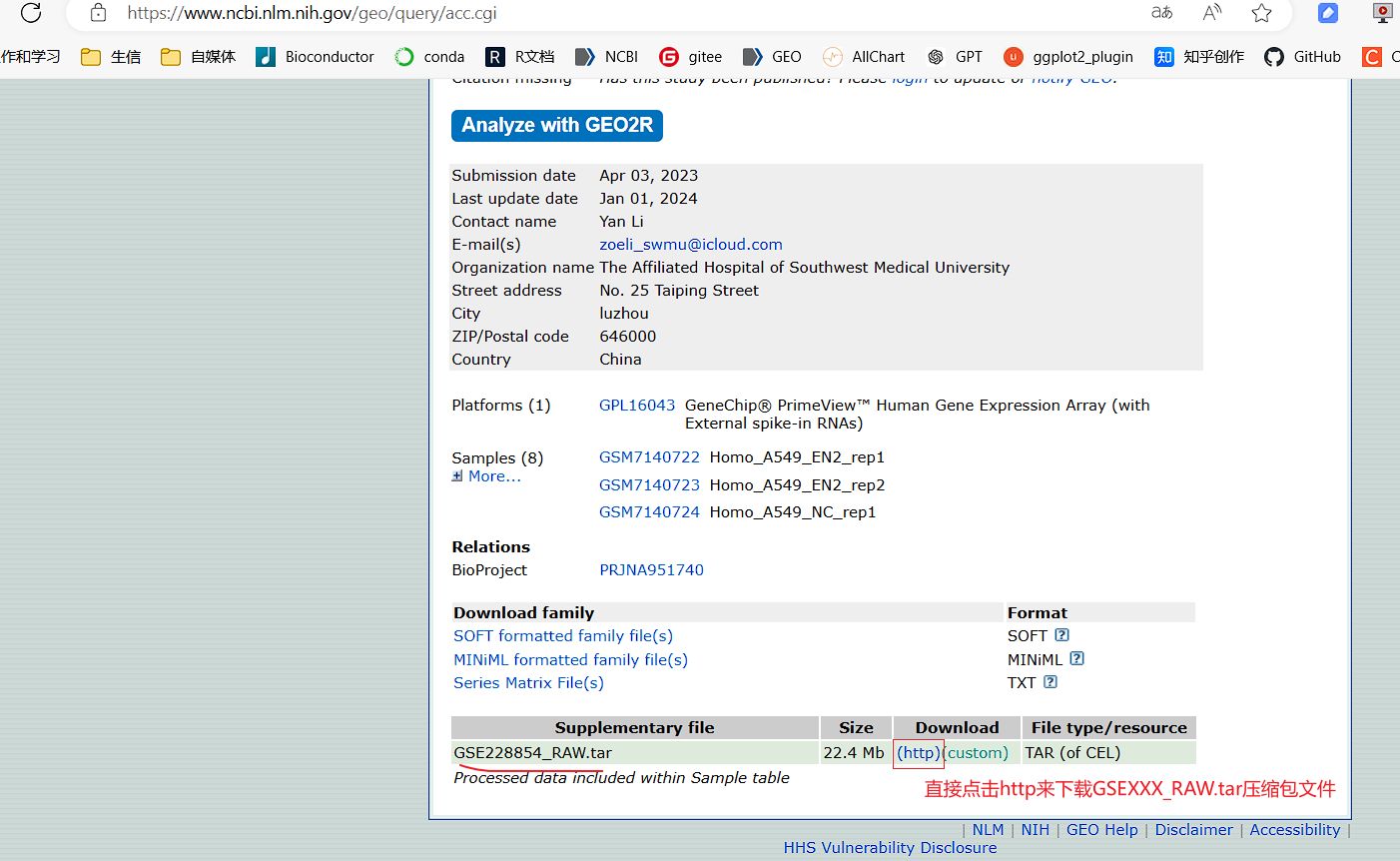
对于部分没有下载出表达矩阵的数据集,如果该数据集的GEO网页中有GSEXXX_RAW.tar格式的压缩包,可以运行下面的1.2这一步对RAW.tar的压缩包文件进行提取和处理。
1.2 甲基化芯片GSE数据集RAW.tar压缩包下载和多样本整合处理教程 (GEO网页中有RAW.tar压缩包可以用这一步下载处理)
教程地址
教程地址:转录组和基因芯片GSE数据集RAW.tar压缩包下载和多样本整合处理教程 - 邢博士谈科教的文章 : https://zhuanlan.zhihu.com/p/708244032
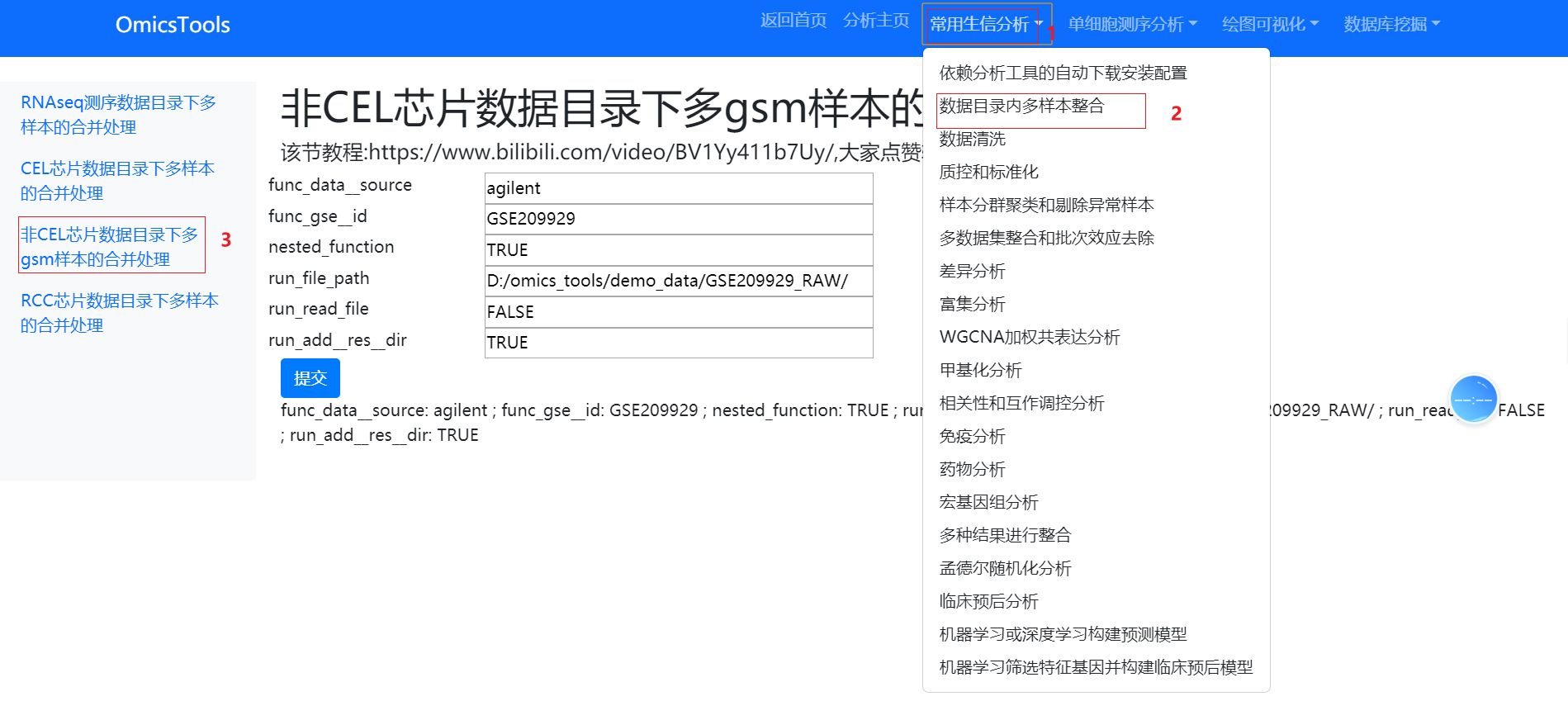
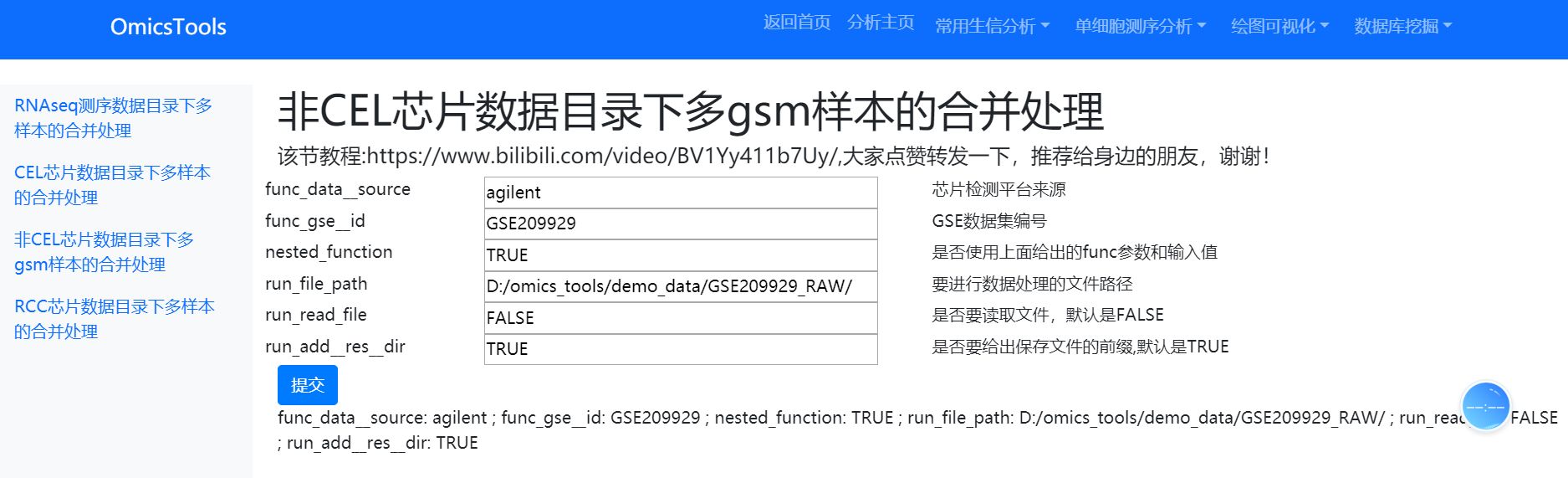
1.3 GEO网页中存在可以手动下载的表达矩阵等文件的下载处理教程
如果在1.1步没有自动下载提取出基因表达矩阵,GEO网页中也没有GSEXXX_RAW.tar压缩包文件,但是存在可以手动下载的一些表达矩阵等数据文件,可以用这一步进行下载和处理。
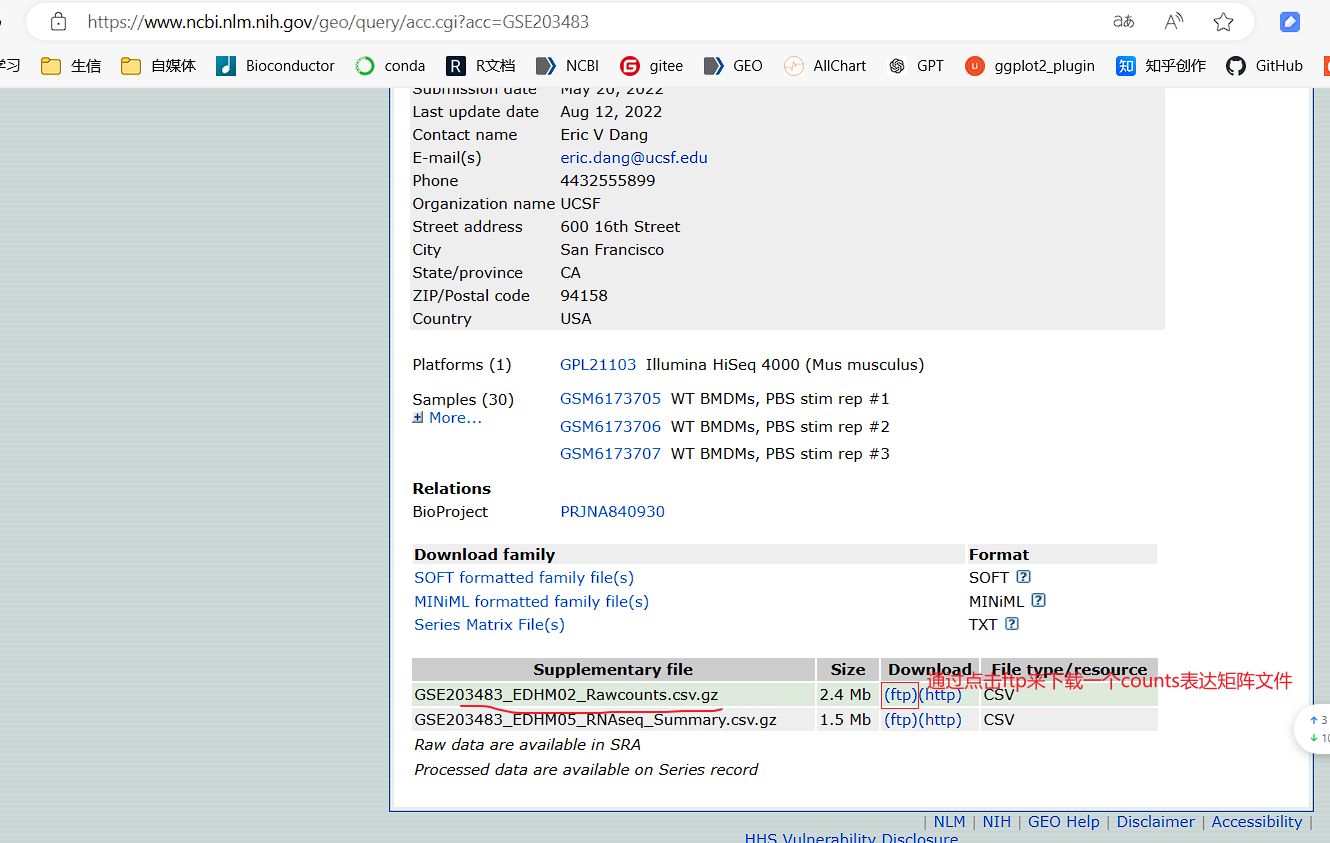

教程视频: https://www.bilibili.com/video/BV1Yf421X7Cj/
下载到的甲基化beta矩阵用limma做差异分析
这个分析很简单,刚开始就两步,第一步把甲基化的表达矩阵提取出来,第二步拿到甲基化的表达矩阵后,不用再做其它的处理,直接用Limma做差异分析就行了。
提取得到的甲基化beta矩阵示意图
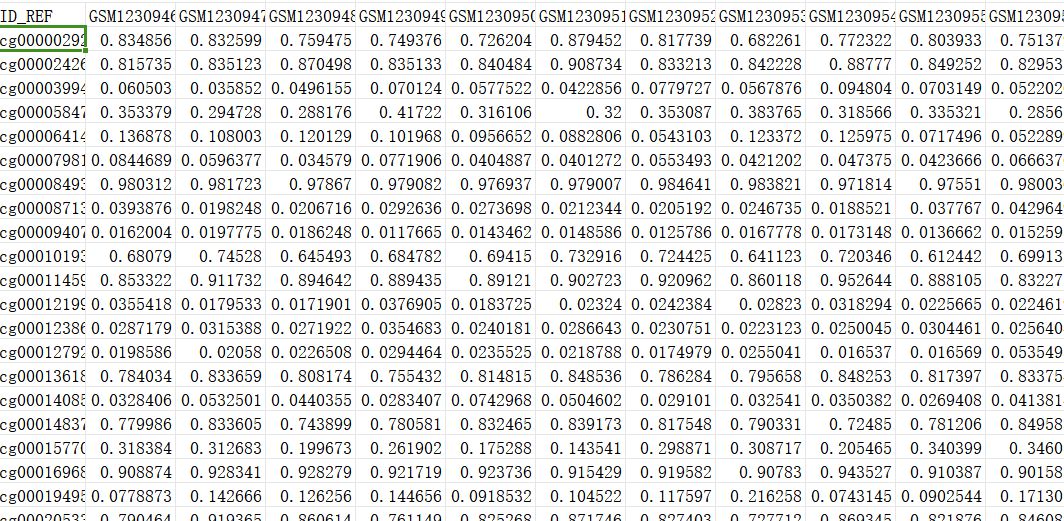
甲基化beta就是在0-1之间,0是该位点没有发生甲基化,1是该位点完全甲基化了
使用limma甲基化矩阵做差异分析

limma差异分析结果
这里limma差异分析结果的gene列是甲基化探针位点,不是甲基化基因名称,需要跟甲基化探针跟基因的注释关系文件进行合并
甲基化探针注释出基因名称,并跟甲基化差异分析结果文件合并
因为每个基因含有多个甲基化位点,所以差异分析结果必须同时保留甲基化位点列和基因名称列,不能把甲基化位点列删掉,同时尽量也包含的有甲基化位点在基因组染色体的起始位置,终止位置,染色体编号,甲基化位点的注释区域等信息,这些都可以从甲基化探针对应的注释文件中获取。
3.1 获取甲基化探针的注释文件(以GEO数据集为例)
GEO的数据集的探针注释信息一般都是存放在GPL平台网页的注释文件中。
进入到GPL平台页面后,下拉到页面末尾,如果有Download full table ,就把这个注释table下载一下
、
下载得到甲基化探针对应的注释信息
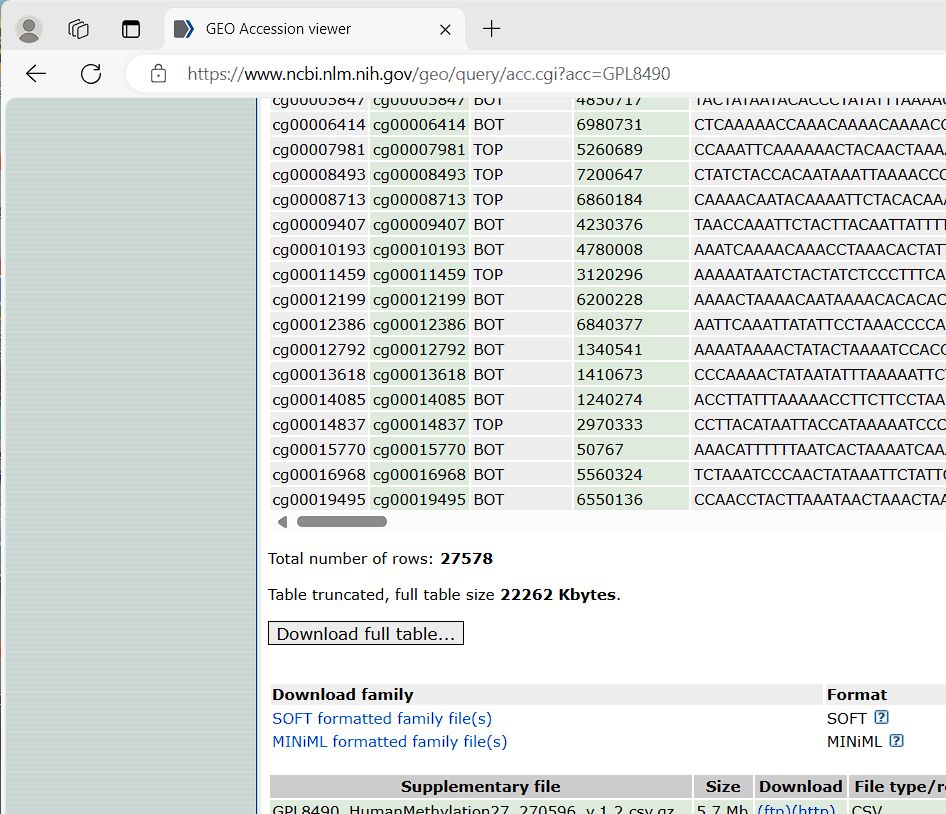

3.2把甲基化探针注释文件跟差异分析结果文件合并


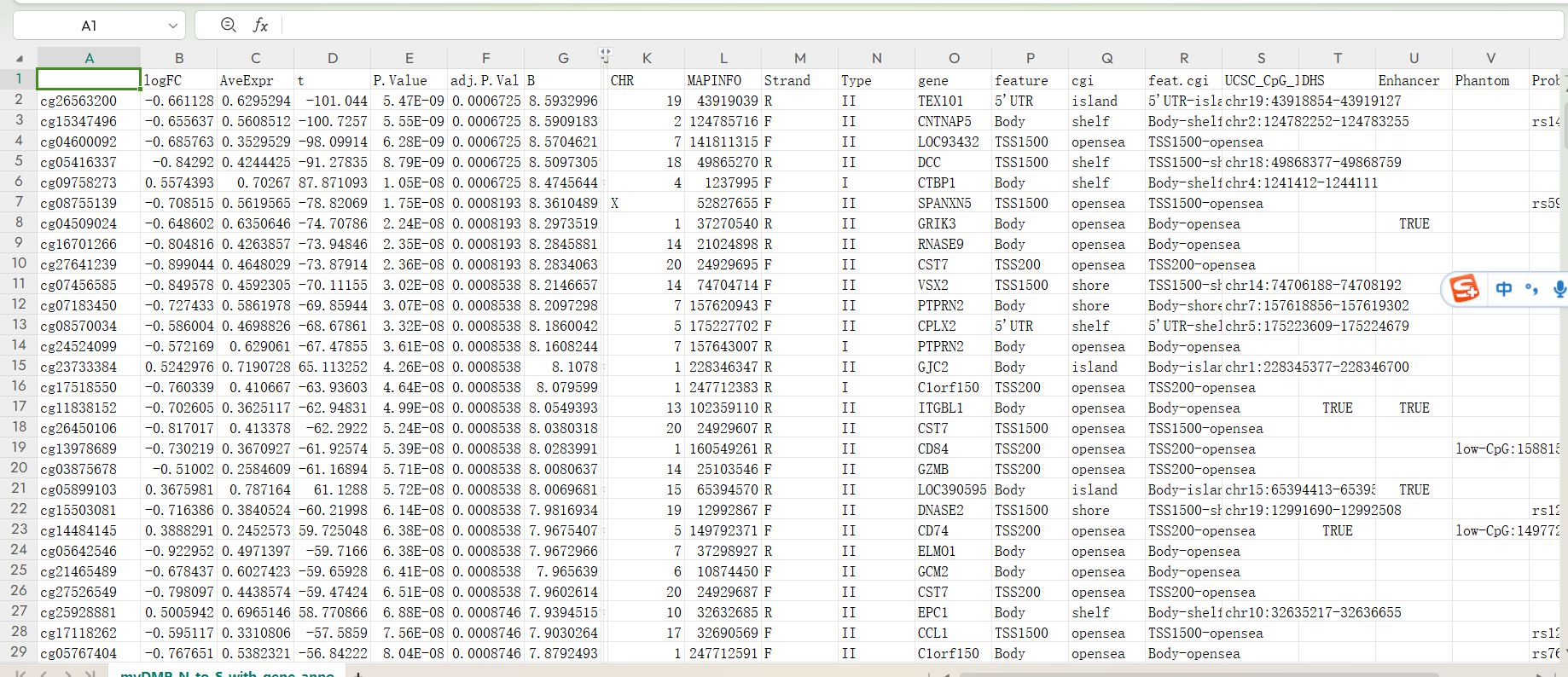
根据甲基化的差异分析结果绘制火山图

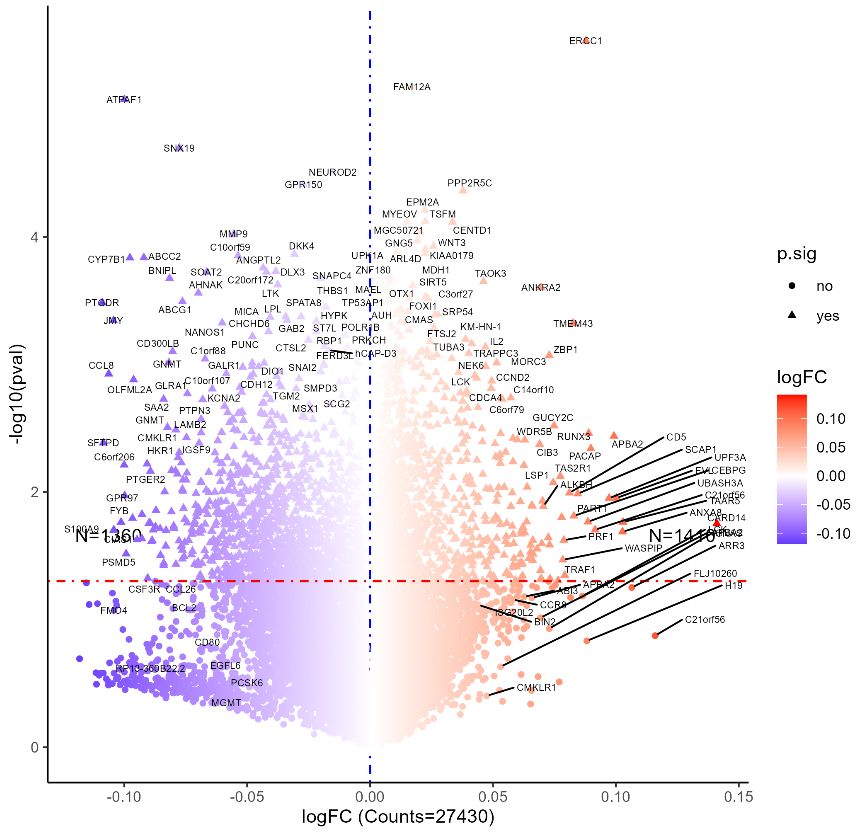
将甲基化的差异分析结果跟转录组差异分析的结果取交集
因为甲基化的foldchange都比较小,因此甲基化的logFC的阈值过滤是跟转录组差异分析的阈值过滤是不一样的,有两种方法可以让两个差异分析的结果取交集。具体方法如下:
- 甲基化位点的差异分析结果先进行过滤筛选,用过滤后的甲基化差异分析结果跟转录组的差异分析结果取交集并绘制韦恩图。
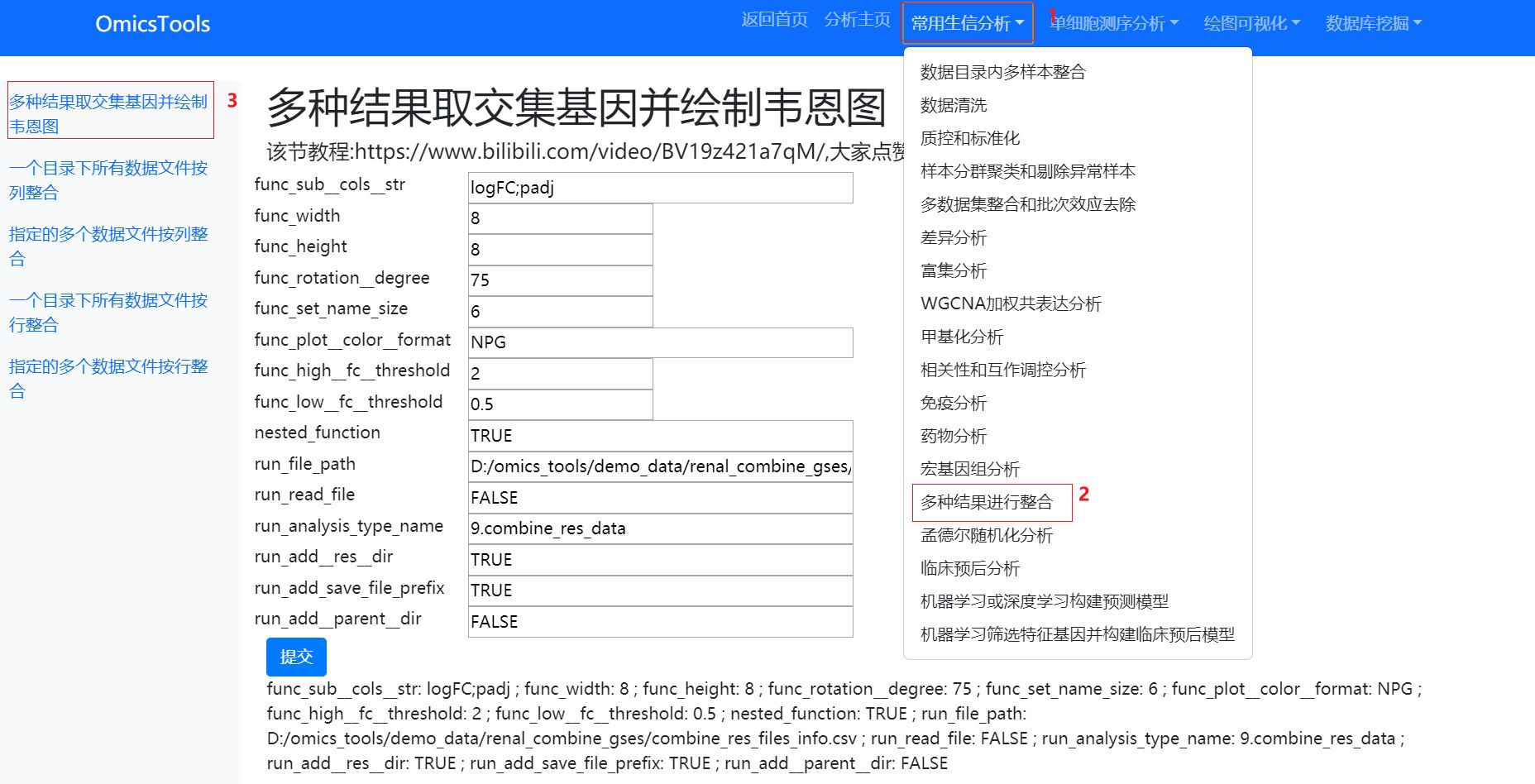
- 我后面会更新多个文件取交集绘制韦恩图的模块,新的模块在合并多个差异分析结果文件取交集时候,可以支持对每个差异分析结果文件指定特定的logFC值和p值。
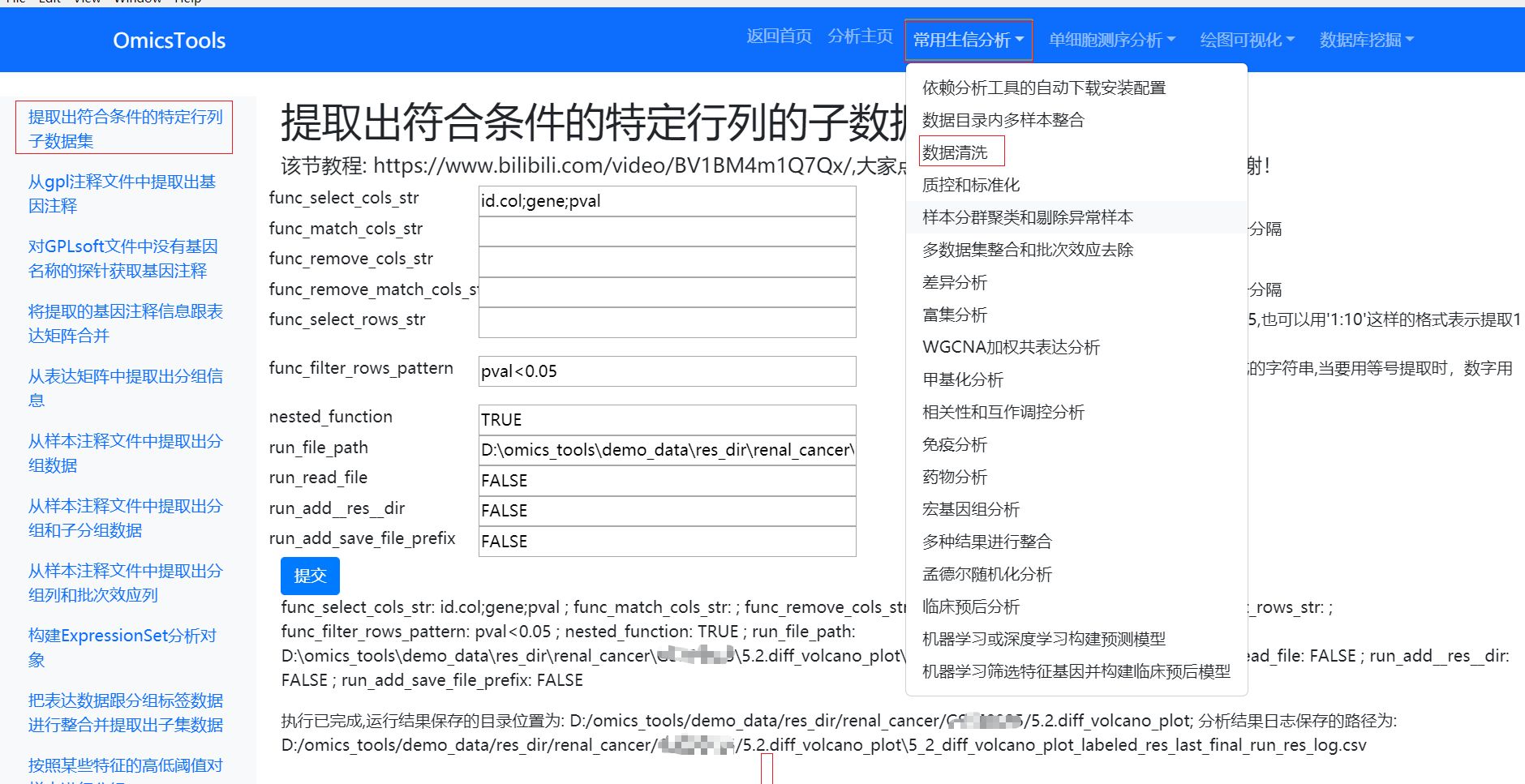
5.1 先对甲基化位点的差异分析文件进行过滤,再跟转录组的差异分析结果进行合并
5.1.1 先对甲基化位点的差异分析文件进行过滤筛选
5.1.2 将筛选好的甲基化位点的差异结果跟转录组的差异结果进行合并

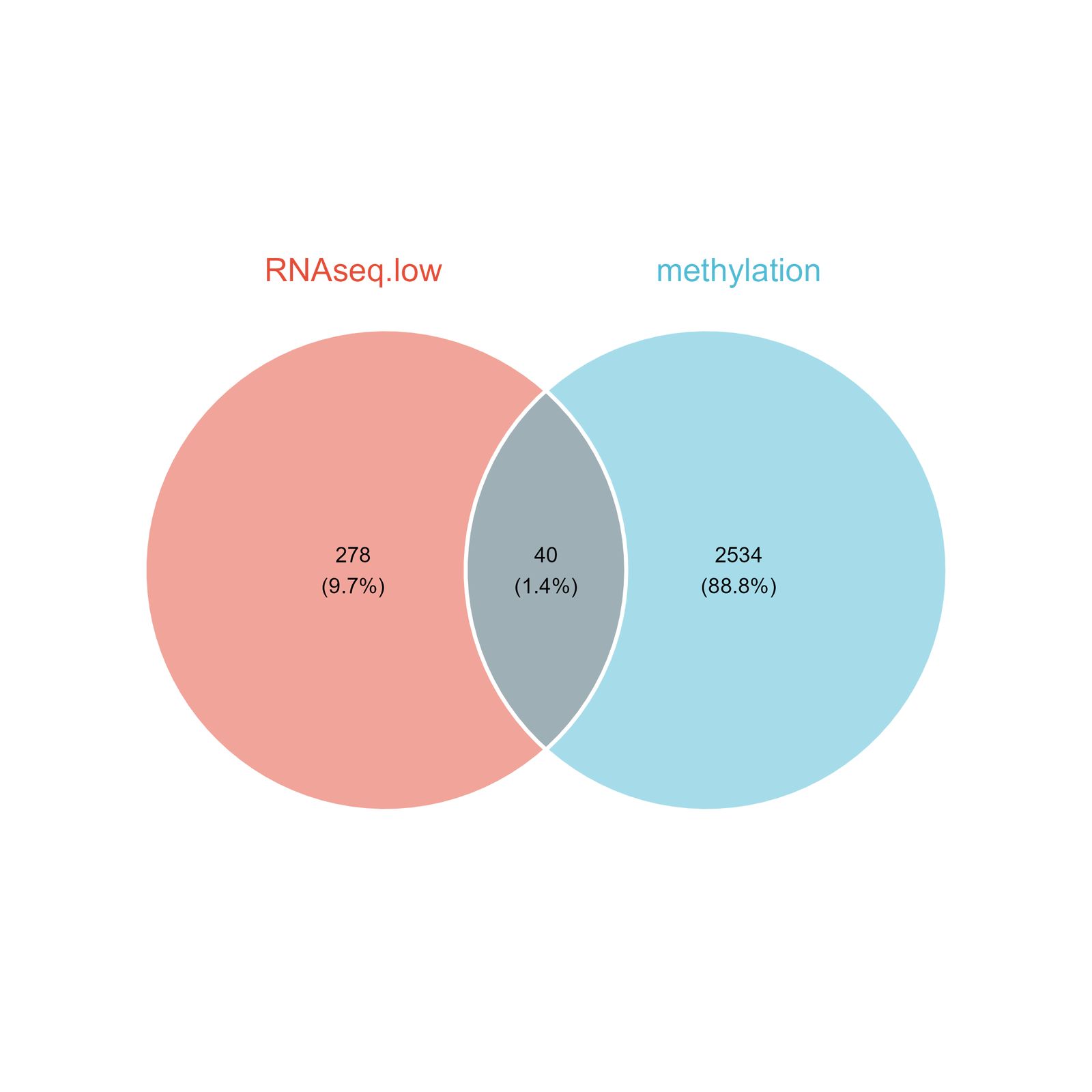

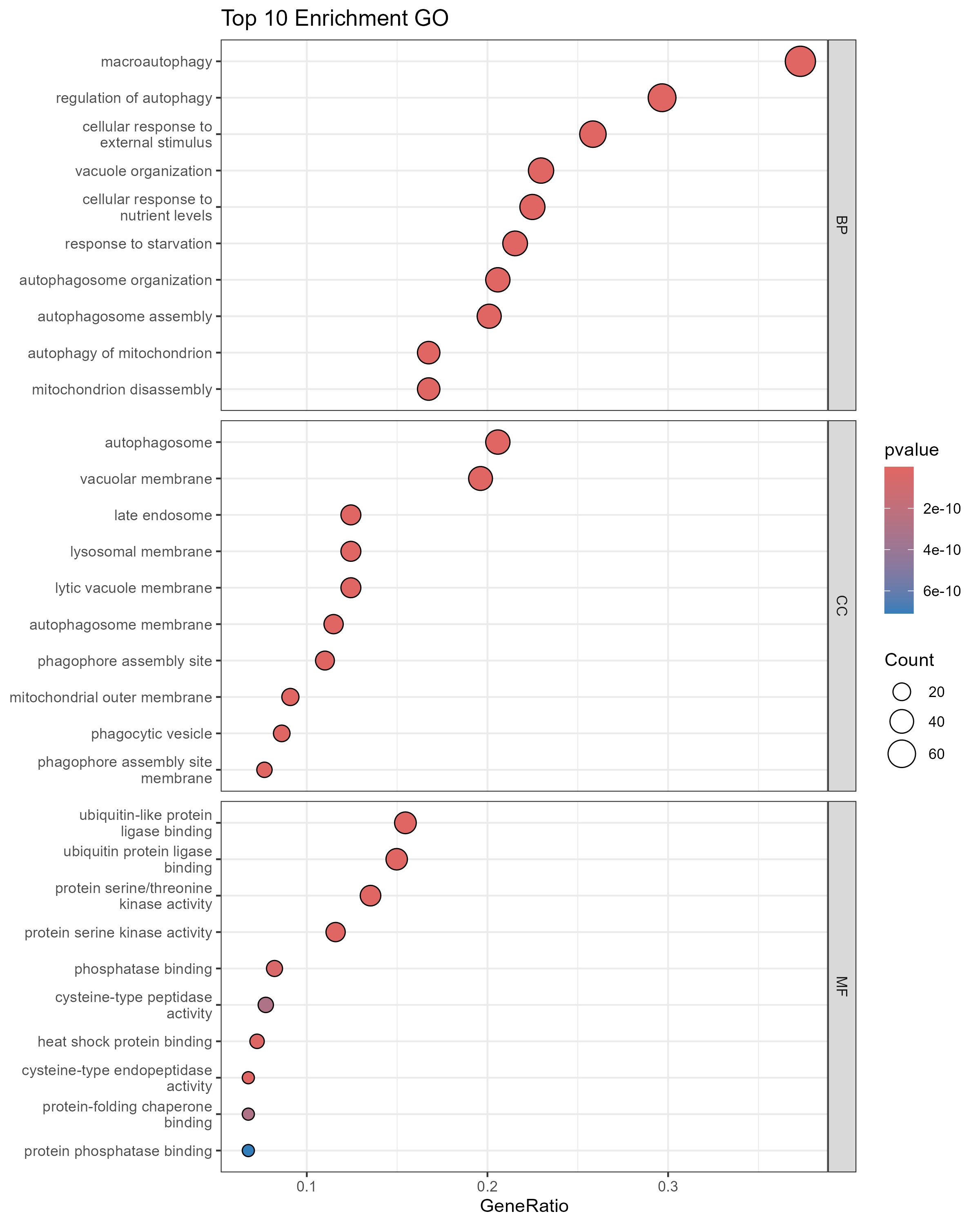
甲基化的差异基因跟转录组的差异基因的交集基因的通路富集分析

go通路富集分析结果
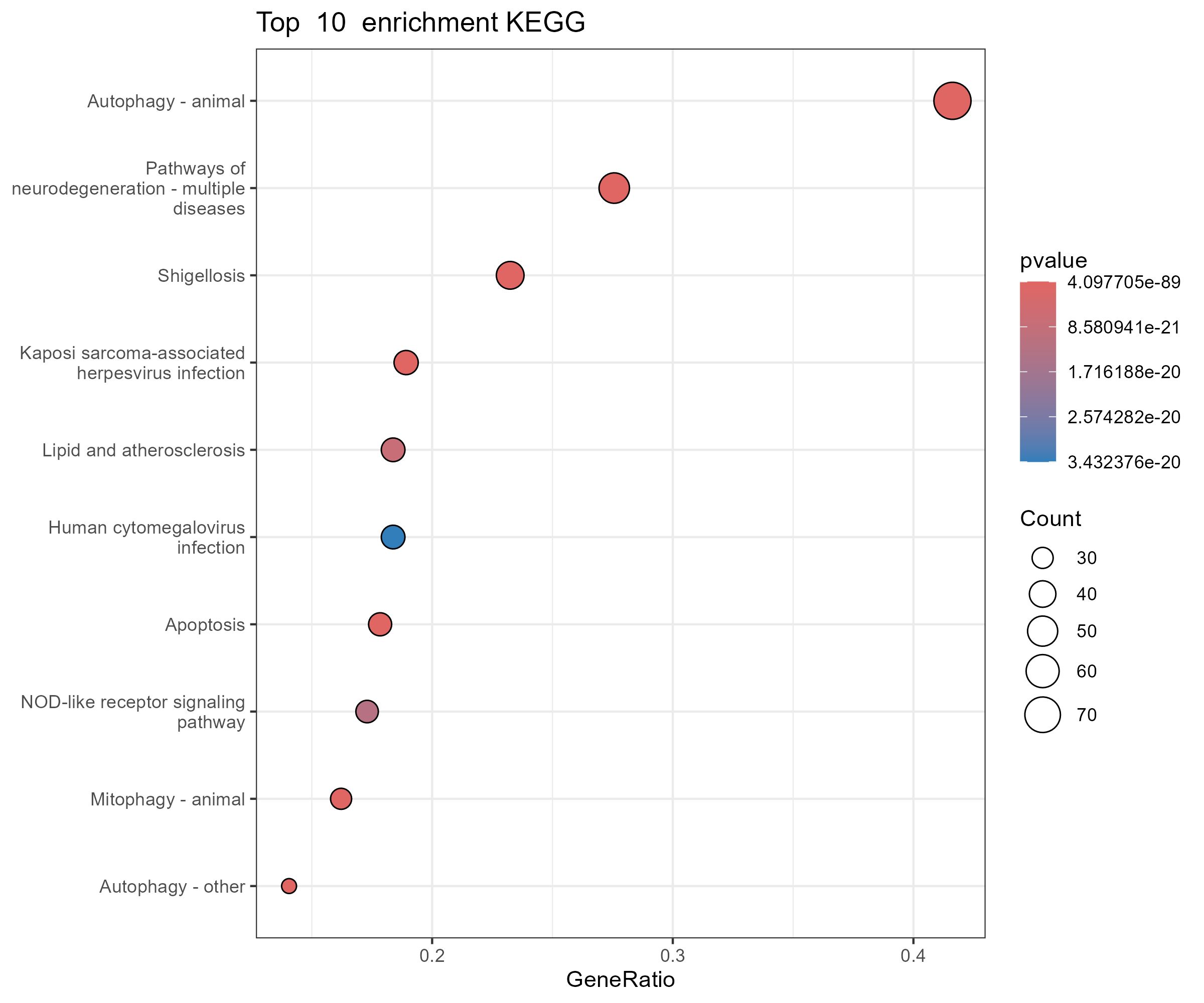
kegg通路富集结果