Ggtree:用于系统发育树及相关数据存储与可视化的数据结构

https://onlinelibrary.wiley.com/doi/10.1002/imt2.56
SHORT COMMUNICATION
● 2022年9月28日,南方医科大学基础医学院余光创团队在iMeta在线发表了题为“Ggtree: a serialized data object for visualization of a phylogenetic tree and annotation data”的文章。
● 该文章设计了ggtree对象用于存储系统发育树,相关数据以及可视化指令,提高了系统发育数据的可重复性与可重用性。
● 第一作者:徐双斌
● 通讯作者:余光创 (gcyu1@smu.edu.cn)
● 合作作者:Lin Li (李林);Xiao Luo (罗晓);Meijun Chen (陈玫君);Wenli Tang (唐文丽);Li Zhan (詹历);Zehan Dai (戴泽翰);Tommy T. Lam (林讚育),Yi Guan (管轶)
● 主要单位:南方医科大学基础医学院生物信息学系;香港大学公共卫生学院新发传染病国家重点实验室;汕头大学病毒学联合研究所
亮 点
● 系统发育树与形式多样的相关数据可以存储在同一个ggtree图形对象中从而提高了系统发育数据的可重复性与可重用性
● 系统发育树与相关数据可以从ggtree对象中提取出来,并重新分析以辅助各个学科进行系统发育信息的综合比较研究
● ggtree图形对象可以被渲染成静态图像,并且保存在该对象中的可视化指令可以类似于Microsoft Word 格式刷操作对不同的树对象进行可视化
摘 要
尽管系统发育树与相关数据已越来越容易生成,但是对它们进行重复使用,组合以及比较仍是困难的。因为当前系统发育树通常是以图片形式进行发布,而且相关数据的存储形式往往也互不兼容。为了提高系统发育数据的可重复性与可重用性,我们设计了ggtree对象用于存储系统发育树,相关数据以及可视化指令。ggtree对象本身是一个图形对象,一方面其可以渲染成静态图像,更重要的是,可视化中的系统发育树与相关数据可以从该图形对象中提取出来,使得ggtree能够成为发布系统发育树(图像、树与数据在一个对象中)的理想数据结构,从而增强数据的重用性,分析的可重复性以及促进综合和比较研究。
视频解读
Bilibili:https://www.bilibili.com/video/BV16m4y1w7TG/
Youtube:https://youtu.be/u_rZY57LFno
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
系统发育数据具有重要的再利用潜力,因为系统发育正成为生态学、进化生物学、流行病学与分子生物学等研究的主要内容之一。对于系统发育数据的重新利用有助于在许多学科中对系统发育信息进行综合比较分析。然而,之前的一项调查表明,大约60%的已发表系统发育数据在科学社区中已永远丢失。造成这一情况的原因之一是系统发育数据的发表共享通常是以静态图像为主,缺乏可互通的共享文件。将相关数据(如分类信息,元数据、表现数据或者流行病学数据)注释到系统发育树图上是一种常用操作。尽管用于系统发育树的可视化以及注释的工具正在逐渐增加,但是这些工具的目的仍然是为了生成可发表的系统发育树图,其中还涉及选择注释数据(如bootstrap值)并将其注释到系统发育树上(比如作为文本标签或者分支颜色)。而这一过程也往往是单向无法逆转地产生一个静态图形,使得一些基础信息无法重用。我们需要从这种生成静态图形的单向目的转换成系统发育树,相关数据以及可视化指令组成的统一数据对象又可渲染成可视化图形的模式。
结 果
用于管理和存储系统发育树以及相关数据的图形对象
在这里,我们提出了一个数据结构,即ggtree对象,定义在ggtree软件包中。Ggtree软件包是一个可用于系统发育树以及各种相关数据的可视化注释的R/Bioconductor软件包。我们之前提出了两种方法将相关数据映射并可视化到系统发育树上。分类单元相关数据可以通过%<+%运算符链接到ggtree对象内的树结构,复杂的相关数据可以使用facet_plot或者geom_facet功能在独立的面板中通过特定的几何图层在与树结构对齐的基础上进行可视化。使用这两种方法映射与可视化的相关数据均被保存在ggtree对象中,且可以被提取出来。如图1所示,我们使用%<+%操作符将相关数据添加到系统发育树上,然后将后验概率值(posterior values)映射到系统发育树上的圆点用颜色深浅表示(图1A)。输出的结果为ggtree对象,其是一个可以被渲染成静态图形的图形对象(图1B)。该对象保留了系统发育树的信息以及相关数据(图1C)。用户可以将此图形对象转换为phylo(只有系统发育树结构没有注释数据)或者treedata(既有系统发育树结构又有数据)树对象。树对象可以使用tidytree或者treeio软件包进行进一步处理,并且也可以导出为Newick、Nexus或者BEAST Nexus,从而可以将相关数据存储为带注释的元素。ggtree图形对象还包含可视化指令与可视化样式,其被可重复,即可以用于新的树对象的可视化(图1D),这类似于Microsoft Word中的格式刷。在facet_plot中使用的相关数据有可能是复杂多样的,比如泛基因组层面的遗传信息与物种丰度分布。虽然数据没有直接映射到树上,但也可以从ggtree对象中提取出来。如图1E,各个物种的体重信息可以使用facet_plot可视化为与树并排对齐的柱状图,并且可以使用facet_data函数从ggtree图形对象中提取出来(图1F)
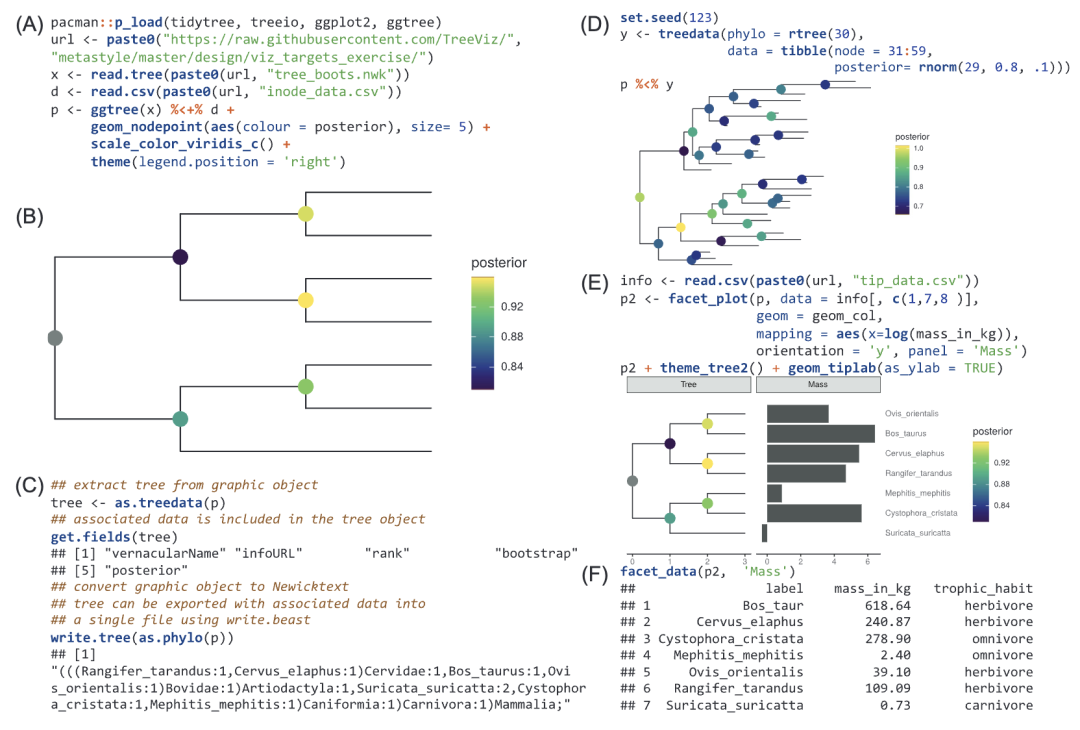
图1. 使用 ggtree 对象存储系统发育树、关联数据和可视化指令的示例图
(A)Newick格式的树文件以及存放内部系统发育树内部节点的posterior的相关数据被读入R中分别存为x与d,通过ggtree()以及%<+%操作符,相关数据与树结构整合为一个ggtree图形对象p。(B)该图形对象可以使用print方法渲染为静态图形。(C)ggtree对象包含了系统发育树与相关数据,它可以被转换为treedata (含有树与相关数据,包括posterior,boostrap值或者其他添加到ggtree对象p的相关数据d),这个treedata对象又可以被进一步导出为Newick 文本(没有相关数据)和 BEAST Nexus 文件(有相关数据)。(D)ggtree对象中含有可视化指令,其可以被重新利用以对新的树对象进行可视化。(E)复杂的相关数据可以使用facet_plot或者geom_facet添加到ggtree对象中。(F)并且通过facet_plot或者geom_facet添加的数据可以facet_data从ggtree对象中提取出来
讨 论
除了系统发育树树本身之外,使用 ggtree 中提出的方法在系统发育树上映射和可视化的所有信息都是可重用的,包括呈现为视觉特征以显示系统发育树的数据和用于生成与树对齐的图形的相关数据。通过使用 ggtree,系统发育数据以及用于生成在出版物的静态图像的可视化指令均被无缝存储在单个对象中。这使系统发育的可视化变得更加便携和透明,也使得高质量的系统发育信息能够在不同的项目中被共享和重用。它还为合作作者之间的树数据远程合作修改沟通提供了巨大的潜力。如果使用 ggtree 图形对象发布了一棵树(即作为补充文件或存放到公开的数据存储库),其他人就可下载该文件并将其读入 R 以重现已发布的结果,并且可以通过重复使用对象中的数据或者其他来源的数据对该图形对象进行添加或修改注释图层。用户也可以从 ggtree 对象中获取树结构和相关数据,再用于配合使用相关的 R 包进行整合和比较分析。这种从生成静态图形到生成 ggtree 这样的数据对象的模式转变不仅提高了系统发育分析的可重复性,而且也确保了数据的可重用性,并促进了综合和比较研究来鉴定并追寻新问题。随着来自各个学科的系统发育数据变得广泛可用,这种模式转变应该成为设计下一代树可视化工具的标准。这个想法也可扩展到生物医学研究的其他领域,以开发可访问、可互操作和可重复使用的科学数据现代可视化工具。
方 法
ggtree软件包是基于ggplot2中实现的图形语法开发的。熟悉ggplot2的用户会发现ggtree非常易于使用,几乎没有学习成本。ggplot2和ggtree的使用体验是一样的,并且可以互相促进。ggtree软件包中定义的ggtree对象继承了ggplot2包中定义的ggplot对象。用户可以使用 ggtree 函数构造一个ggtree对象,该函数支持R中定义的多个树对象,例如phylo、phylo4、pvclust、hclust、diana、phylog、phyloseq等。可以利用%<+%操作符添加与树相关的数据到ggtree对象中,它将数据链接到树结构中并将信息存储在输出的ggtree对象中。这进一步扩展了树对象和相关数据之间的集成,便于数据探索和呈现,因为在R语言中为不同学科定义的许多树对象不支持存储外部数据。从这个角度来看,ggtree对象可以是一个链接,它将其他树对象与外部数据进行整合,然后也可将其转换为treedata对象。这使得tidytree、treeio、ggtree和ggtreeExtra这一系统的软件包能够被更广泛地使用,因为它们可以与treedata对象一起工作,并且可以通过对不同的简单注释图层进行组合以此来处理复杂的系统发育注释,有助于呈现更多样化的与树相关的数据。%<% 运算符提取的可视化指令也可像Microsoft Word格式刷应用于其他树对象的可视化。这也是可视化可重复的重要组成部分,但长期以来却一直被忽视。如果期刊上发表的图表的可视化模式能够支持这样的应用,将极大地促进数据的呈现。而 ggtree 实现的 %<% 操作符证明了这个想法的可能性。
代码和数据可用性
Ggtree软件包可以在https://www.bioconductor.org/packages/ggtree/免费获得,在线书籍https://yulab-smu.top/treedata-book/也提供了ggtree的完整参考。所有的补充材料(文本、图、表、中文翻译版本或视频)也可从线上获取http://www.imeta.science/。
引文格式:
Shuangbin Xu, Lin Li, Xiao Luo, Meijun Chen, Wenli Tang, Li Zhan, Zehan Dai, Tommy T. Lam, Yi Guan, Guangchuang Yu. 2022. “ Ggtree: A serialized data object for visualization of a phylogenetic tree and annotation data.” iMeta. e56. https://doi.org/10.1002/imt2.56
作者简介

徐双斌(第一作者)
● 南方医科大学珠江医院博士后
● 主要研究方向为微生物组学数据挖掘分析。其主导开发了ggtreeExtra,MicrobiotaProcess,ggstar等软件包,也是tidytree,treeio,ggtree,ggbreak等软件包的主要开发者之一。以第一作者(含共一)在Molecular Biology and Evolution,iMeta等国际学术期刊上发表论文6篇

余光创(通讯作者)
● 南方医科大学基础医学院生物信息学系主任、教授、博导
● 一直专注于生物信息学方法工具开发及应用,在系统发育、组学数据分析和数据可视化上开发了多款广受同行认可的生物信息学工具。在Molecular Biology and Evolution, Methods in Ecology and Evolution, Briefings in Bioinformatics, Bioinformatics, The Innovation, iMeta等国际学术期刊上发表论文40余篇,其中有7篇ESI高被引论文,他引频数超过2万次,连续两年入选爱思唯尔中国高被引学者,连续两年入选全球前2%顶尖科学家榜单终身科学影响力和年度科学影响力排行榜