16S扩增子测序是一种广泛应用于微生物群落分析的技术,主要用于研究环境样本中微生物的种类、丰度及其生态关系。
然而,传统的16S扩增子测序通常只能提供相对丰度数据,无法准确反映样本中各微生物的绝对数量,导致在一定程度上掩盖了对微生物潜在的病理学、生理学和生态学意义深入挖掘。因此,通过微生物绝对定量,可以更加准确对微生物的丰度进行测量,对于校正样本间及实验条件差异造成的系统性偏差也是十分必要的。
凌恩生物特推出16S微生物多样性绝对定量(spike-in),即利用高通量测序技术和spike-in内标法来实现对细菌的绝对定量。

1、16S多样性绝对定量Spike-in的定义以及分析流程
Spike-in是指在样品中添加已知拷贝数的内标序列,这些序列通常是人工合成的,与样品中的目标DNA同时进行扩增和测序。通过这种方式,研究人员可以:
建立标准曲线:通过比较测序得到的内标序列的reads数与其已知的拷贝数,绘制标准曲线,从而计算样品中各微生物的绝对拷贝数。
提高定量准确性:Spike-in内标的使用可以校正由于PCR扩增效率差异引起的偏差,使得绝对定量结果更为可靠。

2、16S多样性绝对定量结果展示
以下展示的分别为医口样本和环境样本中TOP物种的16S多样性绝对定量以及相对定量的结果,由图可知,两种方法下物种组成基本一致,而16S多样性绝对定量能够更加直观并真实还原样本本身的细菌含量。
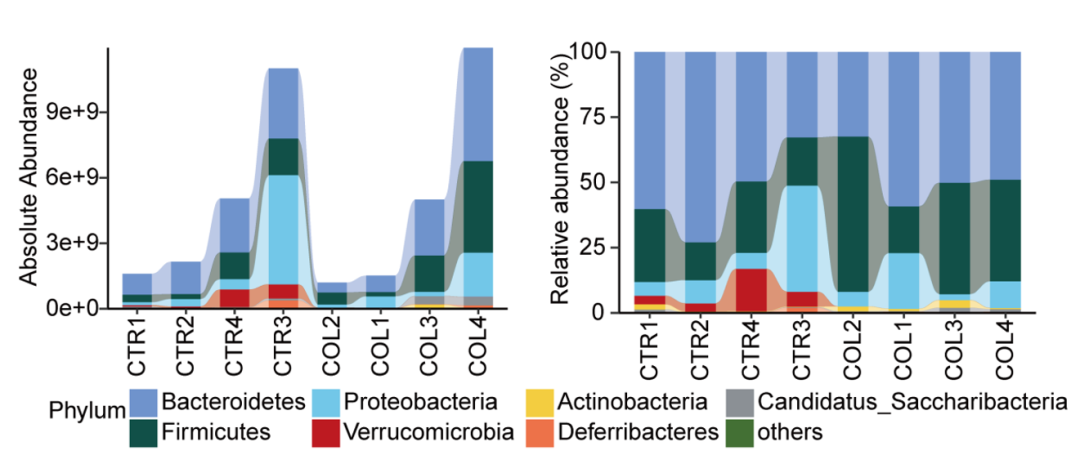
粪便样本:绝对丰度和相对丰度堆积柱状图[1]
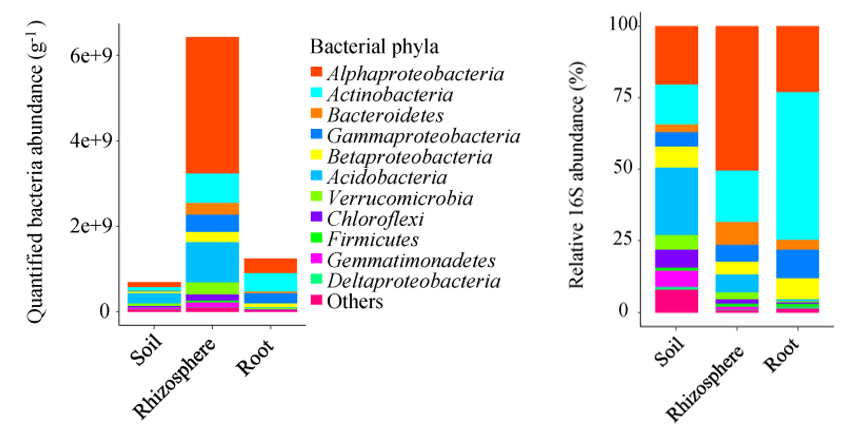
土壤、根际土和根样本:绝对丰度和相对丰度柱状图[1]
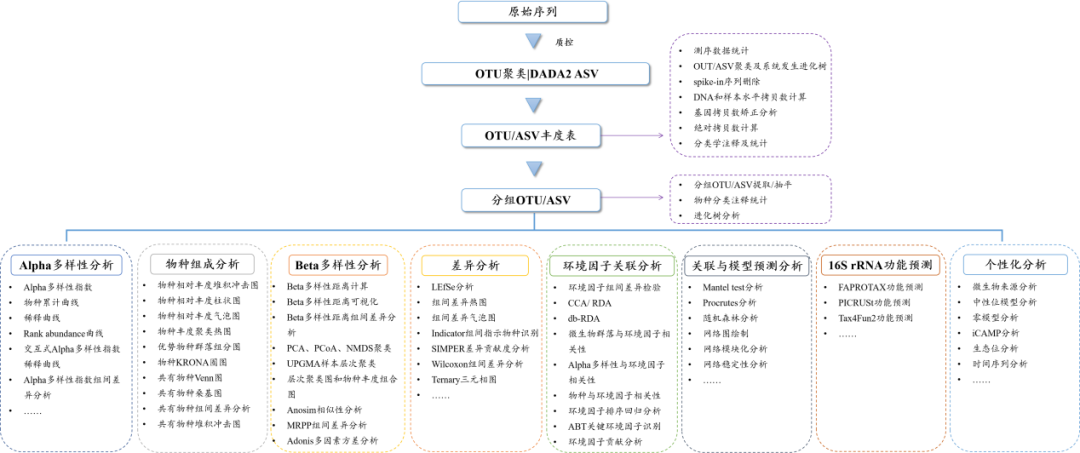
16S多样性绝对定量分析流程图
经典案例
凌恩案例:通过抑制宿主角质层微生物群,有助于昆虫的真菌感染

期刊:Microbiology Spectrum
客户单位:中国科学院上海植物生理与生态学研究所
本研究主要报道了绿僵菌种的HA生物合成有助于真菌局部感染昆虫。HA能够有效抑制从D. melanogaster中分离出的G+细菌的增殖,并在存在不同G+细菌的情况下促进真菌孢子的萌发和穿透玻璃纸膜。定量微生物组分析揭示,HA的产生有助于真菌通过产生抑菌而非杀菌作用来抑制果蝇表皮微生物群落。本研究数据揭示了HA的化学生态学,并强调了真菌病原体在成功感染宿主之前必须应对宿主表皮微生物群落这一事实。
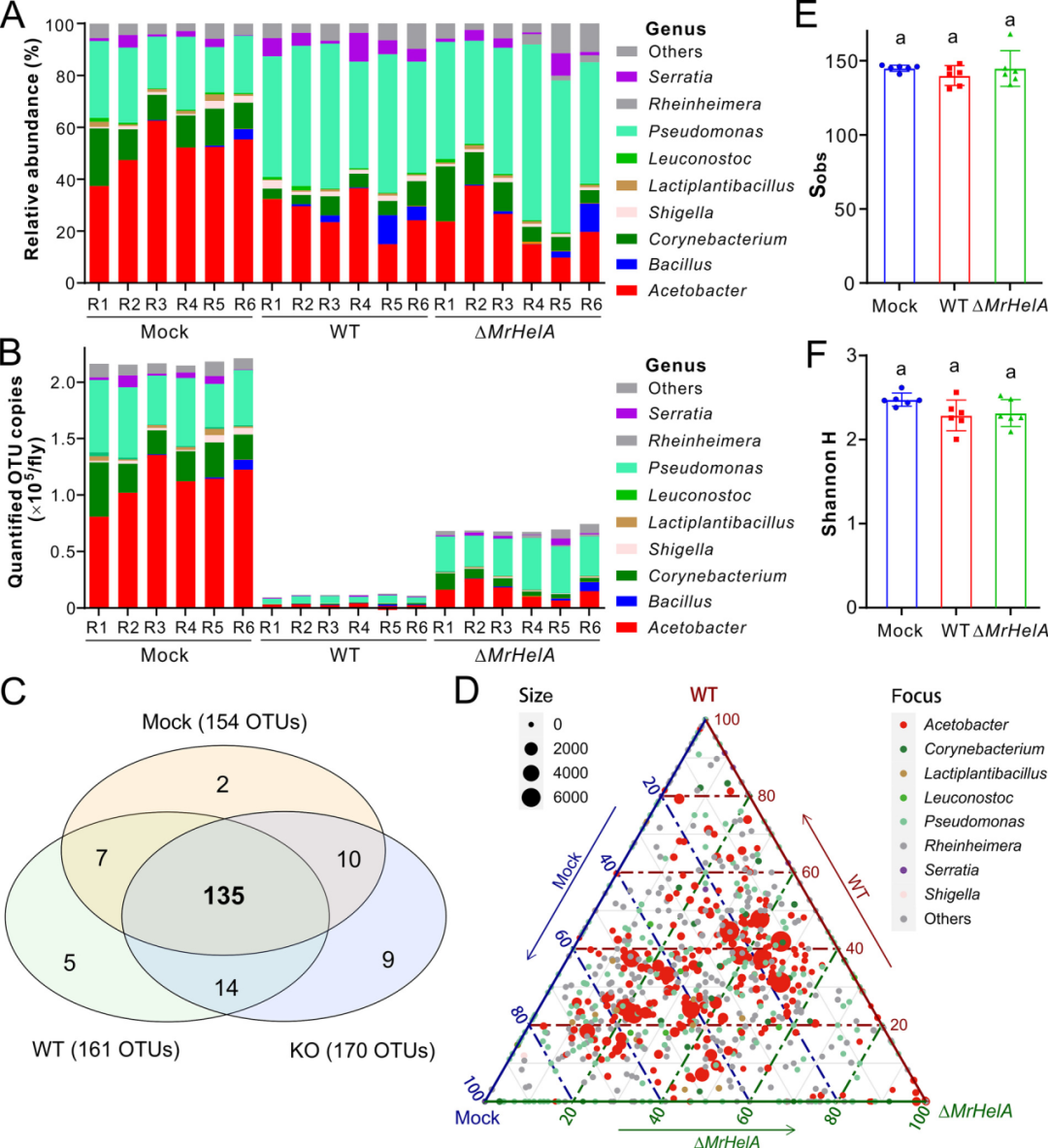
图 (A和B)蝇表面微生物群落的定量分析,显示不同处理在细菌属水平上的相对(A)和(B)绝对丰度差异。(C)维恩图分析显示,检测到的细菌OTUs在不同处理之间基本相同。(D)三元相图分析显示样品之间共有的核心细菌分类群。(E和F)观察到的OTU总数(Sobs)和Shannon H指数在不同处理间没有变化。
参考文献
[1] Inhibition of inosine metabolism of the gut microbiota decreases testosterone secretion in the testis. mSystems, 2024.
[2] An amplification-selection model for quantified rhizosphere microbiota assembly. Science Bulletin, 2020.
[3] Production of Helvolic Acid in Metarhizium Contributes to Fungal Infection of Insects by Bacteriostatic Inhibition of the Host Cuticular Microbiomes. Microbiology Spectrum, 2022.