小罗碎碎念
今天分享的这篇文章是关于一种名为HookNet的新型语义分割模型,它专为病理学全切片图像设计,于2021年发表于《Med Image Anal》,目前IF=10.7。


| 作者角色 | 姓名 | 单位(中文翻译) |
|---|---|---|
| 第一作者 | Mart van Rijthoven | 荷兰Radboud大学医学中心诊断图像分析组及病理学系 |
| 通讯作者 | Mart van Rijthoven | 荷兰Radboud大学医学中心诊断图像分析组及病理学系 |
HookNet结合了编码器-解码器卷积神经网络的多个分支,通过不同分辨率的同心区域来捕获上下文信息和细节。
这种模型通过一种称为“钩连”的机制将不同分支的中间表示结合起来,以实现高分辨率的语义分割。文章介绍了如何设计和训练HookNet,并引入了约束条件以确保在钩连过程中特征图的像素对齐。
研究者展示了HookNet在两个病理学图像分割任务中的优势,这两个任务是:
- (1) 依赖于上下文信息的乳腺癌多类组织分割;
- (2) 肺癌中三级淋巴结构和生发中心的分割。
与单一分辨率的U-Net模型以及最近发布的其他多分辨率模型相比,HookNet展现出了优越性。作者还公开了HookNet的源代码,并提供了基于grand-challenge.org平台的网络应用程序。
- https://github.com/DIAGNijmegen/pathology-hooknet
- https://grand-challenge.org/algorithms/
文章还讨论了在医学图像分析中,如何通过结合上下文信息和细节来提高分割的准确性,尤其是在处理具有广泛细胞种类和数量的组织样本时。
此外,文章还介绍了用于训练和评估HookNet性能的两个数据集:一个乳腺癌数据集和一个肺癌数据集,并详细描述了数据的采集和注释过程。
最后,文章总结了HookNet的主要贡献,包括提供了一个有效的框架来结合上下文和细节信息,并展示了其在乳腺癌和肺癌组织学图像分割中的应用。作者还讨论了未来的研究方向,包括优化HookNet的参数和扩展其应用范围。
一、引言
语义图像分割是一种通过将属于同一概念的像素分组,从而分离不同概念的技术,其目的是简化图像的表示和理解。
在医学成像领域,肿瘤的检测与分割是诊断和疾病特征描述的必要步骤。这在组织病理学中尤为重要,因为病理学家需要分析具有特定背景下广泛种类和数量的细胞组织样本,以达到诊断目的。
高分辨率、高通量的数字扫描仪的引入,实际上已经变革了病理学领域,通过数字化组织样本并生成吉像素级的全切片图像(WSI)。在这种情况下,WSI的数字特性使得计算机算法能够用于自动化组织病理学图像分割,这可为病理学家提供一种有价值的诊断工具,用以识别和描述不同类型的组织,包括癌症。
1.1. 组织病理学中的上下文与细节
已知,尽管单个癌细胞可能在形态学特征上相似,但它们生长成特定模式的方式对患者的预后有着深远的影响。
例如,在苏木精-伊红(H&E)染色的乳腺癌组织样本中,可以区分不同组织学类型的乳腺癌。侵入性导管癌(IDC)起源于乳腺导管,其生长模式多样;而起源于乳腺小叶的侵入性小叶癌(ILC)则以单个肿瘤细胞排列为特征。同样类型的导管癌细胞可能局限于乳腺导管内(导管原位癌,DCIS)或扩散至导管外成为侵入性肿瘤(IDC)(见图1)(Lakhani, 2012)。
为了区分这些癌症类型,病理学家通常结合观察结果,例如,他们会观察组织样本的整体结构组成,并分析每个组织成分的上下文,包括癌症,以识别导管(包括健康和潜在癌性)和其他组织结构的存在。此外,他们会在感兴趣的区域放大观察,以高分辨率获取癌细胞细节,并根据肿瘤的局部细胞组成对其进行特征描述。
另一个例子是病理学家利用上下文和细节的优势来观察免疫细胞的空间分布,这些免疫细胞可能在肿瘤内炎症或癌区域基质部分被检测到,也可能在特定聚集群体中,称为三级淋巴结构(TLS),这是对癌症的反应而发展起来的。
在TLS成熟的后阶段,滤泡中心(GC)在TLS内形成(见图1)。
Fig. 1 展示了在乳腺组织中的导管原位癌(DCIS)、侵润性导管癌(IDC)和侵润性小叶癌(ILC)的例子,以及在肺组织中的三级淋巴结构(TLS)和生发中心(GC)。
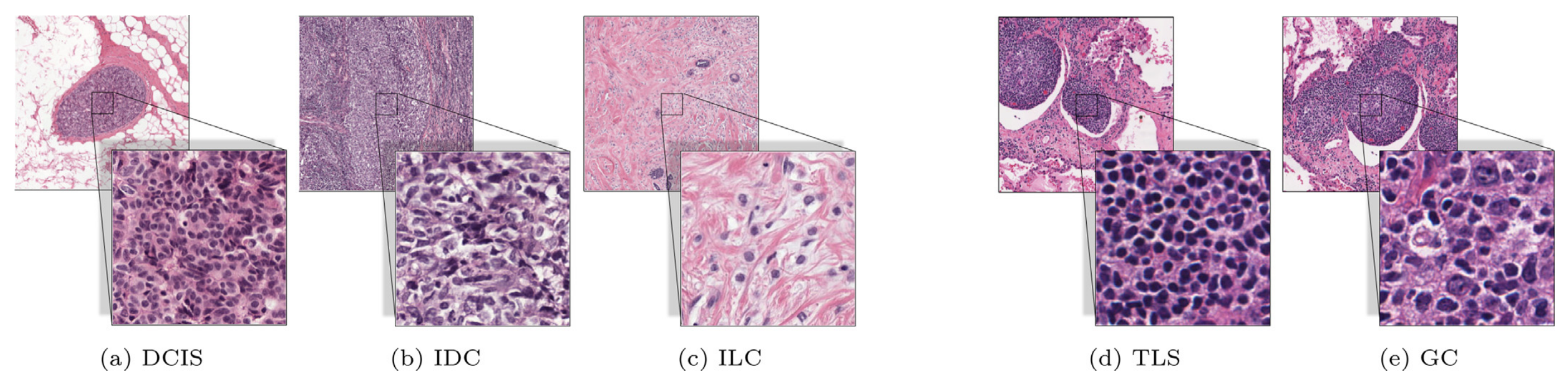
对于每个示例,都展示了多分辨率/多视场(MFMR)图像块:包括一个低分辨率/大视场和同心的高分辨率/小视场图像块。
- 导管原位癌(DCIS):这是一种非侵润性乳腺癌,癌细胞局限在乳腺导管内,没有扩散到周围组织。
- 侵润性导管癌(IDC):是最常见的乳腺癌类型,癌细胞已经突破导管限制,开始侵润周围正常组织。
- 侵润性小叶癌(ILC):这种癌症起源于乳腺小叶,其特点是由单独排列的肿瘤细胞组成。
在肺组织中:
- 三级淋巴结构(TLS):这是在慢性炎症或癌症中发现的免疫细胞聚集,对癌症患者的预后具有重要意义。
- 生发中心(GC):位于TLS内,是B细胞增殖和分化的场所,对适应性免疫反应至关重要。
MFMR图像块是:
- 低分辨率/大视场:提供了更广阔的视野,有助于观察组织的全局结构和上下文信息,但缺乏细节。
- 高分辨率/小视场:提供了更小区域的详细视图,有助于识别细胞级别的特征和模式。
这种多分辨率和多视场的方法允许模型同时捕获到组织结构的上下文信息和细胞细节,对于病理学家进行准确的诊断和疾病特征描述至关重要。在自动化图像分析中,这种方法可以帮助提高算法对不同类型组织的识别能力,尤其是在区分具有相似形态特征的癌症类型时。
研究表明,含有GC的TLS的发展对患者的生存具有显著意义,是理解肿瘤发展和治疗的关键因素(Sauts-Fridman等,2016;Silić等,2018)。GC总是位于TLS内。TLS包含高密度的淋巴细胞,其细胞质不易见,而GC则与密度较低的组织,如肿瘤巢相似。
为了识别TLS区域并在TLS与GC之间进行区分,需要精细的细节和上下文信息。
1.2. 感受野与视场
近年来,绝大多数图像分析算法基于卷积神经网络(CNN),这是一种深度学习模型,能够处理包括语义分割在内的多种计算机视觉任务(Long等,2015;Jégou等,2017;Chen等,2018)。
在语义分割中,像素级别的标签预测依赖于感受野(RF),即模型可观察的输入区域的大小。CNN的RF大小取决于滤波器大小、池化因子以及卷积和池化层的数量。通过增加这些参数,RF也会增大,使模型能够捕获更多的上下文信息。
然而,这通常以输入尺寸的增加为代价,导致由于大型特征图而造成的高内存消耗。因此,在模型优化中常常需要应用一些隐含的限制,如减少模型的参数数量、特征图数量、小批量大小或预测输出的大小,这可能导致训练效率低下和推理效率低下。
另一个与可观察信息相关的方面是视场(FoV),即输入图像中像素覆盖的面积。
FoV依赖于输入图像的空间分辨率。FoV对RF有影响:相同的模型、相同的输入尺寸和相同的RF尺寸,通过考虑较低分辨率的图像,可以包含更宽的FoV,因为像素覆盖的面积被压缩(即更少的像素揭示与原始分辨率相同的FoV)。
因此,使用原始输入图像的下采样表示,模型可以受益于更多的上下文聚合(Graham和Rajpoot,2018),代价是失去高分辨率细节。此外,上下文聚合受输入维度限制,意味着RF大小只能通过使用填充的人工输入像素来超过输入维度(通常称为使用相同填充的技术),而这些填充像素不包含上下文信息。
虽然减少原始输入维度可以用来关注尺度信息(Kausar等,2018;Li等,2018),但潜在的上下文信息保持不变。
1.3. 多视场多分辨率补丁
全切片图像(WSI)是包含多分辨率吉像素图像的金字塔型数据结构,其中包括原始图像的下采样表示。
在CNN模型开发背景下,由于WSI包含的像素数量达到数十亿,单个现代GPU的内存容量无法在一次RF中捕获完整WSI的全分辨率图像。一种常见的克服这一限制的方法是使用补丁(即WSI的子区域)来训练CNN。
由于WSI的多分辨率特性,补丁可以来自不同的空间分辨率,以微米/像素(μm/px)表示。通过在WSI中选择一个位置、大小和特定分辨率来提取补丁。
在最高可用分辨率下提取补丁时,潜在的上下文信息并未耗尽,因为补丁周围还有未被RF考虑的可用组织。通过提取同心(即以全切片图像的同一位置为中心)的补丁,大小相同但分辨率更低,相同的RF会聚集更多的上下文信息,并包括之前不可用的信息。
从不同分辨率提取的多个同心补丁,可以解释为具有多个FoV。因此,我们称这些补丁为多视场多分辨率(MFMR)补丁(见图1)。
迄今为止,从组织病理学图像中提取MFMR补丁的研究主要集中在结合从MFMR补丁获得特征进行补丁分类(Alsubaie等,2018;Sirinukunwattana等,2018;Wetteland等,2019)。然而,使用补丁分类进行语义分割会导致粗糙的分割图或由于需要滑动窗口方法而造成的大量计算,该方法用于对每个像素进行分类。
对于分割任务,使用MFMR补丁并非直接可行:在结合从MFMR补丁获得的特征时,应强制执行像素对齐。Gu等(2018)提出使用U-Net架构(Ronneberger等,2015)处理高分辨率补丁,并使用附加编码器处理较低分辨率的补丁。随后,从附加编码器获得的特征图被裁剪、上采样并与U-Net解码器部分的跳过连接处进行拼接。来自附加编码器的特征图在上采样时没有跳过连接,这牺牲了定位精度。
此外,他们提出的模型在解码器的每个深度处拼接特征图,这可能是冗余的,并且会导致内存消耗高的模型。而且,考虑到像素对齐的必要性,他们的模型限于使用相同填充,这可能引入伪影。
在已知类别受上下文和细微细节影响的多类别问题中,可以从MFMR补丁集成的信息中受益。然而,这仍是一个未解决的问题。挑战在于同时输出基于高分辨率下可检测的细微细节的高分辨率分割,并融入上下文特征。
1.4. 我们的工作贡献
本文介绍了HookNet,这是一个基于卷积神经网络的多分支分割框架,能够同时融入上下文信息和高清细节,以生成组织病理学图像的精细分割图。从多分支分割模型的角度看,所提出的框架引入了几个新颖的技术贡献:
(1)它通过挂钩机制直接且有效地跨分支结合像素对齐的特征图;
(2)它在所有分支中允许跳过连接,从而支持定位精度,同时使用有效卷积,并保持结合特征图的像素对齐。
从数字病理学应用的角度看,这项工作通过两个多类别和多器官分割模型做出了贡献,这些模型解决了组织受高清细节和上下文影响的问题。
- 第一个应用专注于组织病理学乳腺组织中的DCIS、IDC和ILC分割。
- 第二个应用专注于肺组织中的GC、TLS和肿瘤分割。据我们所知,这些组织类型从未被单个模型同时且分别分割。
这两个模型以网络应用程序的形式发布。
二、材料与方法
为了训练和评估HookNet的性能,我们收集了两个数据集:乳腺数据集和肺数据集。
2.1 乳腺数据集
我们收集了86份乳腺癌组织切片,包含IDC(n = 34)、DCIS(n = 35)和ILC(n = 17)病例。
对于DCIS和IDC病例,我们使用了H&E染色的组织切片,这些切片最初是为了常规诊断而制作的。所有组织切片均按照Radboud大学医学中心病理科(荷兰)的实验室协议准备。切片使用Pannoramic P250 Flash II扫描仪(3DHistech,匈牙利)以0.24 μm/px的空间分辨率进行数字化。
对于ILC,我们切割新的组织切片并进行H&E染色,之后使用与IDC/DCIS病例相同的扫描仪和扫描协议进行扫描。数字化WSIs后,H&E染色的ILC切片进行了脱色和后续的重新染色(Brand等,2014),使用P120连环蛋白抗体(P120)(Canas-Marques和Schnitt,2016),这种抗体在癌细胞质中染色,而不是在正常上皮细胞的膜上染色。P120染色的切片随后使用与H&E切片相同的扫描仪和协议进行扫描。这一过程使我们能够获得同一组织切片的H&E和免疫组化(IHC)图像。
三位人员参与了手动标注的制作:两名医学研究助理,他们在病理科接受了专门的监督培训,以识别和标注组织病理学切片中的乳腺癌组织;以及一位具有六年诊断和研究经验的住院病理医师(MB)。
为了指导ILC的标注过程,住院病理医师在HE切片上视觉识别并标注了包含肿瘤主体的区域(见图2)。
Fig. 2 描述了在乳腺癌组织切片中手动标注侵润性小叶癌(ILC)区域的过程。
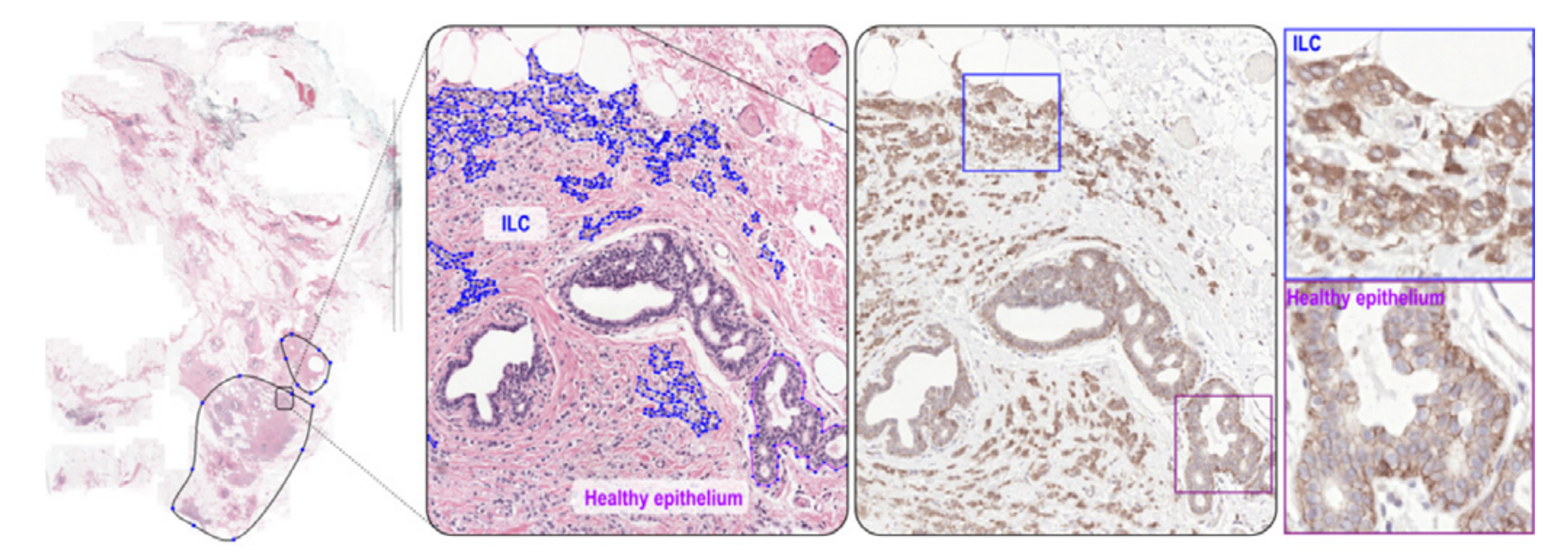
该图由两个部分组成:
-
左侧图(H&E染色):
- 展示了使用苏木精-伊红(H&E)染色的组织切片。
- 在这张图上,标注者手动标出了包含肿瘤主体的区域。
- 同时,还展示了对ILC和健康上皮细胞的手动(稀疏)标注细节。
- 这种标注可能是非密集型的,意味着并非所有该类型组织的实例都被标注,且同一实例内并非所有像素都被包括在绘制的轮廓内。
-
右侧图(免疫组化染色):
- 展示了同一组织样本的免疫组化染色图像,该样本先前被H&E染色后又去除了H&E染色,并使用P120抗体重新染色。
- P120抗体特异性地染色了小叶癌细胞的细胞质,与正常上皮细胞的膜染色模式不同。
- 这种染色方法被医疗研究助理用来指导手动标注,并帮助识别ILC细胞。
在手动标注过程中,涉及三名人员:
- 两名医疗研究助理,他们接受了病理系的监督培训,专门识别和标注病理组织学切片中的乳腺癌组织。
- 一位具有六年数字病理学诊断和研究经验的住院病理医师(MB),负责检查所有标注并必要时进行更正。
通过这种结合H&E染色和免疫组化染色的方法,研究团队能够更准确地标注出ILC细胞,这对于病理学诊断和研究至关重要。标注结果将用于训练和验证像HookNet这样的模型,使其能够自动识别和分割不同类型的组织和细胞。
随后,研究助理利用这一信息以及可用的IHC切片来识别和标注ILC细胞。此外,研究助理还对DCIS、IDC、脂肪组织、良性上皮和其他组织类别(包含炎症细胞、皮肤/乳头、红细胞和基质)进行了标注。所有标注最终由住院病理医师检查,并在需要时进行修正。
使用内部开发的开放源代码软件ASAP(Litjens等,2018)进行手动标注。结果标注了6,279个区域,其中1,810个包含ILC细胞。组织区域的标注是稀疏的,意味着绘制的轮廓可能既不全面(即没有标注该组织类型的所有实例),也不密集(即同一实例的所有像素并未包含在绘制的轮廓中)。
图2展示了稀疏标注的示例。因此,在这个数据集中标注了6个类别。为了训练、验证和测试目的,WSIs被分为训练集(n = 50)、验证集(n = 18)和测试集(n = 18),所有集合都包含类似的癌症类型分布。
2.2 肺数据集
我们从癌症基因组图谱肺鳞状细胞癌(TCGA-LUSC)数据集中随机选取了27份诊断用的H&E染色数字切片,这些数据在基因组数据共享(GDC)数据门户中公开可用(Grossman等,2016)。
对于这个数据集,一位具有六年以上肿瘤免疫学和病理学经验的高级研究员(KS)进行了TLS、GC、肿瘤和其他肺实质的稀疏标注,并由一位住院病理医师(MB)进行检查。
结果在这个数据集中标注了1,098个注释,包括4个类别。为了模型开发和性能评估,我们使用了3折交叉验证,这允许我们在所有可用的切片上测试所呈现模型的性能。所有三个折包含了12:6:9的切片用于训练:验证:测试。我们确保所有分割都具有适当的类别平衡。
三、HookNet:多分支编码器-解码器网络
在本节中,我们介绍了一种用于语义分割的卷积神经网络模型“HookNet”,该模型通过多个编码器-解码器模型的分支处理同心多分辨率MFMR补丁,并通过“挂钩”机制结合不同分支的信息(见图3)。
Fig. 3 描述了 HookNet 模型的架构,该模型是一个用于处理多分辨率/多视场(MFMR)图像块的双编码器-解码器网络。
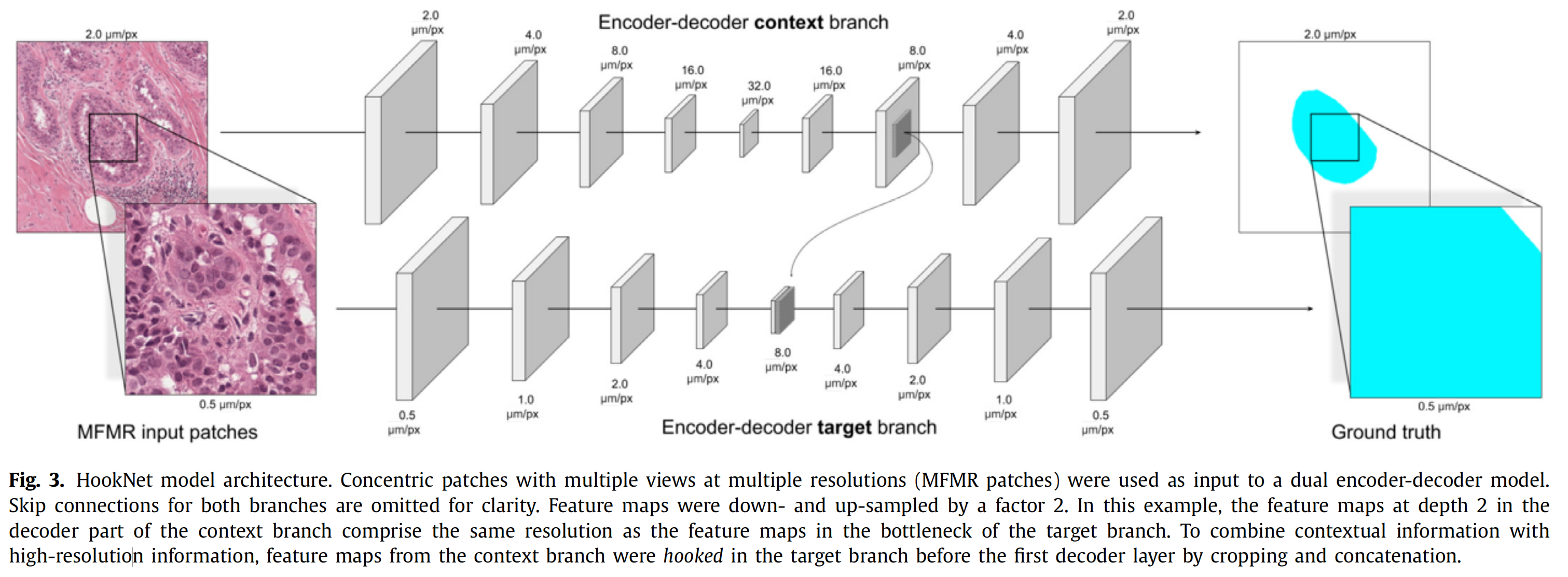
-
MFMR 图像块输入:
- HookNet 接受在不同分辨率下获取的同心图像块作为输入,这些图像块包含了从低分辨率的大视场到高分辨率的小视场的多种视图。
-
双编码器-解码器模型:
- 模型由两个分支组成,每个分支都是一个编码器-解码器结构,基于 U-Net 架构设计。
- 这两个分支不共享权重,但具有相同的架构。
-
特征图的下采样和上采样:
- 在编码器中,特征图通过最大池化层进行下采样,步长为2,这减少了特征图的空间尺寸。
- 在解码器中,特征图通过最近邻上采样和随后的卷积层进行上采样,恢复了特征图的空间尺寸。
-
钩连机制:
- 为了将上下文信息与高分辨率信息结合起来,来自上下文分支的特征图在目标分支的第一个解码器层之前通过裁剪和连接的方式进行“钩连”。
- 这种钩连是在特征图具有相同分辨率的情况下进行的,确保了特征图之间的像素对齐。
-
特征图分辨率匹配:
- 在示例中,上下文分支解码器部分的深度2处的特征图与目标分支瓶颈处的特征图具有相同的分辨率。
-
跳过连接(Skip Connections):
- 为了清晰起见,图示中省略了两个分支的跳过连接。
- 跳过连接是 U-Net 的关键特性之一,它将编码器中的高级特征与解码器中的低级特征相结合,有助于改善模型的细节捕获能力。
-
特征图裁剪和连接:
- 在钩连过程中,上下文分支的特征图被裁剪,以确保它们与目标分支的特征图在空间尺寸上匹配,然后进行连接。
-
模型的深度和结构:
- 每个分支由多个卷积层和池化层组成,形成编码路径,随后是解码路径,其中包括上采样和卷积操作。
HookNet 的这种设计允许模型同时利用来自低分辨率图像块的上下文信息和高分辨率图像块的细节信息,以实现更准确的语义分割。这种多分辨率处理对于处理具有复杂结构和细节的病理学全切片图像尤其重要。
HookNet的目标是结合(1)低分辨率补丁的大视场信息,这些补丁携带上下文视觉信息,以及(2)高分辨率补丁的小视场信息,这些补丁携带精细的视觉信息。
为此,我们提出HookNet作为一个模型,包含两个编码器-解码器分支,即一个上下文分支,用于提取包含上下文信息的输入补丁的特征,以及一个目标分支,用于从最高分辨率的输入补丁中提取细粒度的细节以进行目标分割。
该模型的关键思想是通过跨分支拼接特征图来结合细粒度和上下文信息,从而类似于病理学家在观察组织时缩放和缩放的过程。
我们按照应该设计的顺序来介绍HookNet的四个主要组件,即
- (1)分支架构和属性
- (2)MFMR补丁的提取
- (3)“挂钩”机制的约束
- (4)目标和损失的处理
3.1. 上下文和目标分支
HookNet设计的第一步是定义其分支。为了不失一般性,我们假设模型设计满足以下假设:
- (1)两个分支具有相同的架构但权重不共享
- (2)每个分支都基于U-Net(Ronneberger等,2015)架构的编码器-解码器模型。
与原始U-Net模型类似,每个卷积层执行有效的3×3卷积,步长为1,后跟具有2×2下采样因子的最大池化层。
对于上采样路径,我们采用了Odena等人(2016)提出的由最近邻2×2上采样和卷积层组成的方法。
3.2. MFMR输入补丁
HookNet的输入是一对(PC, PT)的(M×M×3)MFMR RGB同心补丁,分别从两个不同的空间分辨率rC和rT(以μm/px为单位)提取,分别用于上下文(C)和目标(T)分支。
为了获得无缝的分割输出并避免由于编码器-解码器分支中的特征图错位而产生的伪影,应特别选择输入补丁的大小和分辨率。首先,M必须选择,以确保在编码路径中的所有特征图在每次池化层之前都有偶数大小。
最初在Ronneberger等人(2015)中引入,这个约束对于HookNet至关重要,因为不均匀大小的特征图不仅通过跳过连接导致特征图错位,而且在跨分支中也会导致特征图错位。因此,这个约束确保了跨两个分支的特征图保持像素对齐。
其次,rT和rC应该选择,以便在给定的分支架构下,跨分支的解码路径中的特征图对具有相同的分辨率。这对接下来的“挂钩”机制至关重要。在实践中,给定编码器-解码器架构的深度D(即池化层的数量),rC和rT应取值,使得以下不等式成立: 2 D 2^D 2DrT ≥ rC。
3.3. 挂钩机制
我们提出通过简单地拼接来自两个分支的解码路径中提取的特征图来组合上下文分支的信息到目标分支中。我们选择拼接作为组合特征图的操作,是基于原始U-Net中跳过连接的成功,这些连接也使用拼接。此外,与预定义的硬编码操作,如特征图的激活值乘法或特征图之和相比,拼接允许下游卷积层在参数优化过程中学习最佳的组合特征操作。
上下文和目标分支的特征图不应该在瓶颈层之前拼接,以便在单独进行语义编码。我们假设拼接最好在目标分支的解码路径的开头进行,以便最大限度地利用解码路径中的内在上采样,其中拼接的特征图可以从目标分支中的每个跳过连接中受益。我们称这种拼接为“挂钩”,为了保证特征图的像素对齐,我们定义特征图的空间分辨率为 S R F = 2 d ∗ r SRF = 2^d*r SRF=2d∗r,其中d是编码器-解码器模型中的深度,r是输入补丁的分辨率(以μm/px为单位)。
为了定义挂钩可以发生的位置之间的相对深度,我们定义特征图对之间的SRF比为:
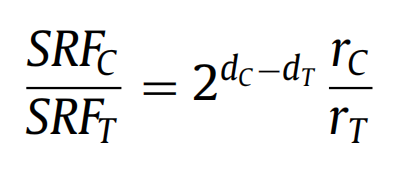
其中dT和dC分别是目标分支和上下文分支的相对深度。在实践中,挂钩可以在特征图对具有相同分辨率时发生:SRFC/SRFT = 1。
因此,上下文分支中深度dC的特征图中的中央方形区域对应于目标分支中深度dT的特征图。这个中央方形区域的大小与目标分支的特征图大小相同,因为两个特征图具有相同的分辨率。为了实际挂钩,可以简单地裁剪,以便上下文分支的特征图与目标分支的特征图像素对齐地拼接在一起。
3.4. 损失函数
在训练HookNet时,可以为每个分支计算一个单独的损失。我们提出一个损失函数L = λLhigh + (1 - λ)Llow,其中Lhigh和Llow分别是目标分支和上下文分支的像素级分类交叉熵,λ控制每个分支的重要性。
3.5. 基于像素的采样
补丁在特定组织类型的中心位置进行采样,即类标签。由于地面真实标签的稀疏性,一些补丁包含的地面真实像素少于其他补丁。
在训练过程中,我们确保每个类标签通过以下基于像素的采样策略得到同等代表。在第一个小批量中,补丁是随机采样的。在所有后续的小批量中,补丁采样是基于之前小批量中看到的每个类的地面真实像素数量的积累。包含较少像素积累的类有更高的采样机会,以补偿未充分代表的类。
3.6. 模型训练设置
补丁的尺寸为284×284×3,我们使用12个样本的小批量大小,这使得每个批量可以包含两倍于类别的数量。
卷积层使用有效的卷积、L2正则化和ReLU激活函数。每个卷积层后都跟随批量归一化。两个分支都包含4个深度(即4个下采样和4个上采样操作)。
如第3.1节所述,对于下采样和上采样操作,我们使用最大池化和最近邻,后跟卷积层。为了预测软标签,我们使用softmax激活函数。通过调整λ值,可以控制目标分支和上下文分支损失的贡献。我们测试了λ = 1(忽略上下文损失)、λ = 0.75(给予目标分支更多重要性)、λ = 0.5(给予上下文分支和目标分支同等重要性)和λ = 0.25(给予上下文损失更多重要性)。
此外,我们使用了学习率为5×10^-6的Adam优化器。我们自定义了所有模型的过滤器数量,以便每个模型大约有5000万个参数。
我们训练了200个周期,每个周期包括1000个训练步骤,然后计算验证集上的F1分数,以确定最佳模型。为了增加数据集的变化,并考虑染色变化引起的颜色变化,我们应用了空间、颜色、噪声和染色(Tellez等,2018)增强。本工作中未使用染色标准化技术。
四、实验
为了评估HookNet,我们将其与五个使用以下分辨率提取的补丁训练的独立U-Net模型进行了比较:0.5、1.0、2.0、4.0和8.0 μm/px。这些模型分别表示为U-Net(rt)和HookNet(rt, rc),其中rt和rc分别是目标分支和上下文分支的输入分辨率。
HookNet的目标是输出高分辨率的分割图,因此目标分支将处理从0.5 μm/px分辨率提取的输入补丁。对于上下文分支,我们提取了中间(2.0 μm/px)和极端(8.0 μm/px)分辨率的补丁,这些分辨率对于单分辨率模型进行了测试,并显示了在单分辨率性能指标中的潜在价值。
对于乳腺数据,这些分辨率是2.0和8.0 μm/px(见表1)。
Table 1 展示了在Radboudumc测试集上,使用不同输入分辨率的U-Net模型对乳腺癌组织类型的性能表现,性能以每种组织的F1分数来报告。
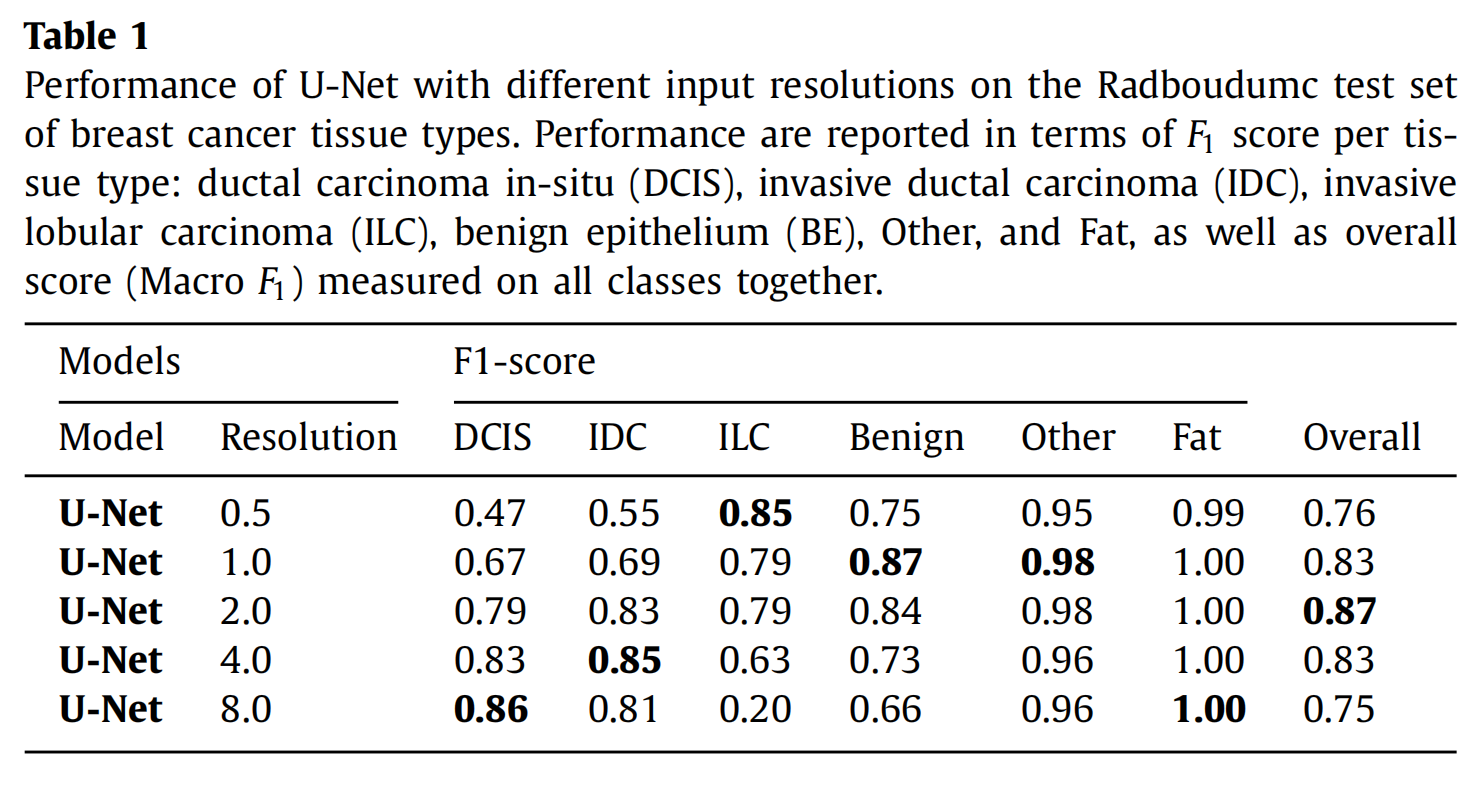
-
组织类型:表格列出了六种不同的组织类型,包括导管原位癌(DCIS)、侵润性导管癌(IDC)、侵润性小叶癌(ILC)、良性上皮(BE)、其他(Other)和脂肪(Fat)组织。
-
分辨率:U-Net模型在五种不同的分辨率(0.5, 1.0, 2.0, 4.0, 8.0 μm/px)下进行了训练和测试。
-
F1分数:对于每种分辨率和每种组织类型,表格报告了相应的F1分数,这是一个综合了精确度和召回率的性能指标,用来衡量模型在特定组织类型上的分割性能。
-
整体性能(Macro F1):除了单个组织类型的F1分数外,表格还提供了一个整体的Macro F1分数,这是将所有类别的F1分数平均计算得出的,用以衡量模型在整个数据集上的平均性能。
具体分析如下:
- 在0.5 μm/px分辨率下,模型在区分ILC和Fat组织上表现良好,但在区分DCIS和IDC上表现较差。
- 当分辨率增加到1.0 μm/px时,DCIS、IDC和ILC的F1分数都有所提高,表明增加一些上下文信息有助于改善性能。
- 在2.0 μm/px分辨率下,模型在所有组织类型上都展现出了更好的性能,特别是DCIS和IDC,这表明在保持一定细节的同时增加上下文信息是有益的。
- 然而,在4.0 μm/px分辨率下,尽管IDC和BE的F1分数仍然很高,但DCIS和ILC的F1分数有所下降,表明过多的上下文信息可能会损害对细节的识别。
- 在8.0 μm/px分辨率下,模型在区分ILC上表现极差,这可能是因为在这种高分辨率下,模型丢失了必要的上下文信息,导致难以区分ILC和其他组织。
总体而言,表格显示了分辨率对模型性能的影响,以及在不同分辨率下模型对不同组织类型的识别能力。这些结果对于理解在设计分割模型时如何平衡上下文信息和细节的重要性提供了有价值的见解。
对于肺数据,只有中间分辨率2.0 μm/px显示了潜在价值(见表3)。
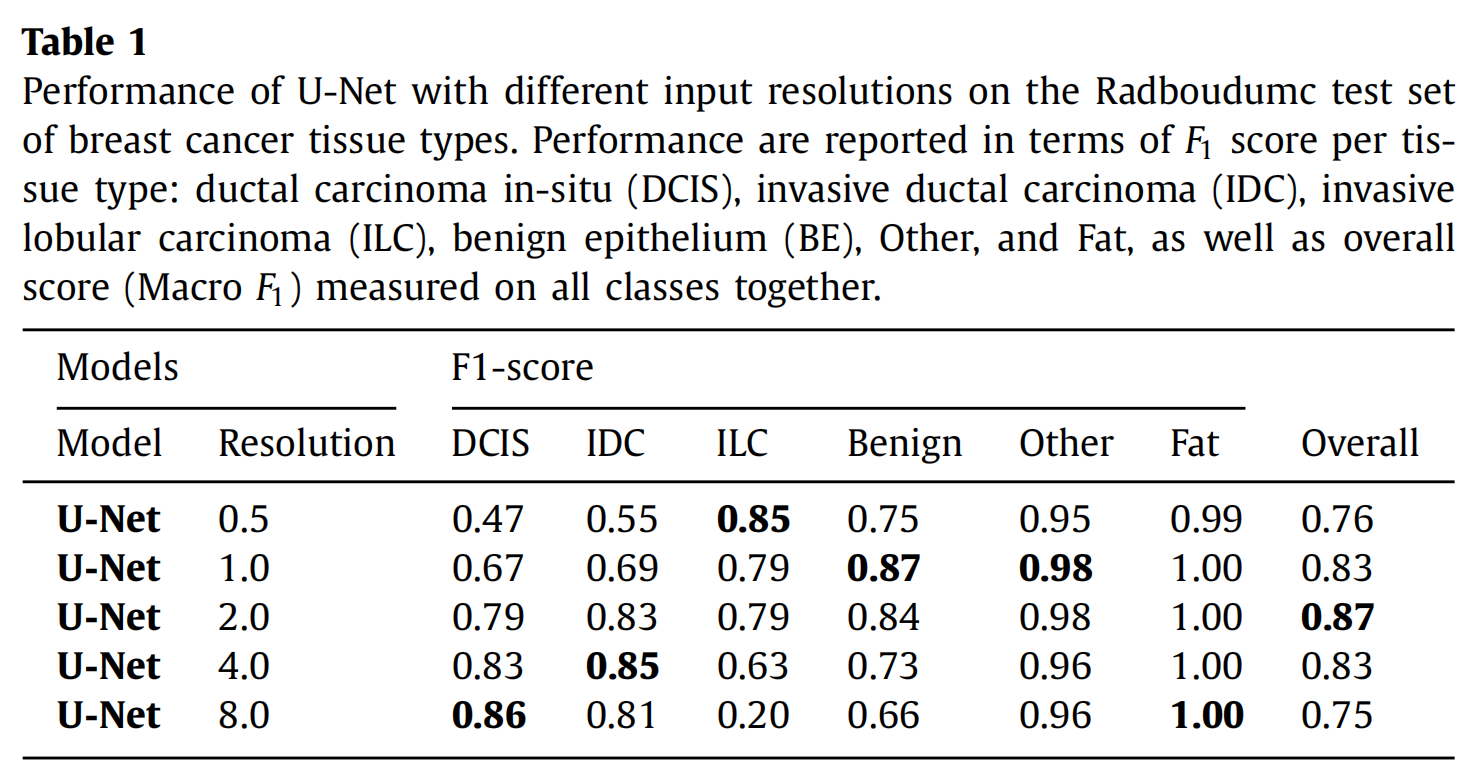
Table 2 比较了三种不同的模型——U-Net(0.5)、多分辨率网络(MRN)和HookNet——在Radboudumc乳腺癌组织测试集上的性能表现。性能以每种组织类型的F1分数来报告,并给出了整体的Macro F1分数。
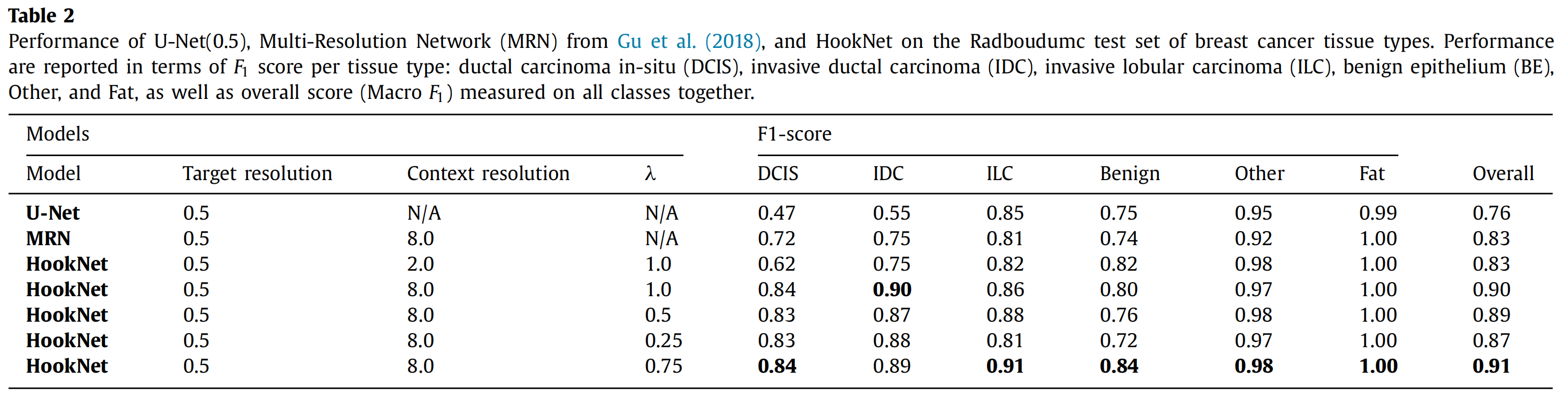
-
U-Net(0.5):
- 这是使用0.5 μm/px分辨率训练的U-Net模型。
- 该模型在区分ILC和Fat组织上表现良好,但在DCIS、IDC和其他组织类型上的F1分数较低。
-
MRN:
- MRN是Gu等人(2018年)提出的多分辨率网络模型。
- MRN在DCIS和IDC上的F1分数较高,但在ILC上的F1分数较低,这表明MRN在区分某些组织类型上可能存在局限性。
-
HookNet:
- HookNet是本文提出的模型,它结合了不同分辨率的信息以提高分割性能。
- 表格中列出了HookNet在不同λ值(用于平衡目标分支和上下文分支的损失)下的性能。
- 当λ=1.0时,即不考虑上下文分支的损失,HookNet在TLS和GC上的F1分数略高于U-Net(0.5),但在Tumor上的F1分数略有下降。
- 当λ=0.75时,HookNet在所有组织类型上的F1分数都有所提高,尤其是在DCIS、IDC和ILC上,这表明适当的上下文信息对性能有积极影响。
-
组织类型:
- 表格中列出了五种不同的组织类型:DCIS、IDC、ILC、BE、Other和Fat。
- 每种模型在每种组织类型上的F1分数都有所不同,这反映了模型在区分不同组织类型上的能力。
-
整体性能(Macro F1):
- Macro F1是对所有类别的F1分数进行平均计算得出的,用以衡量模型在整个数据集上的平均性能。
- HookNet在不同的λ值下展现出了不同的整体性能,其中λ=0.75时的整体性能最佳。
-
模型比较:
- 通过比较三种模型的F1分数,可以看出HookNet在结合多分辨率信息方面的优势,尤其是在λ=0.75时,其在多个组织类型上的性能都有所提高。
-
上下文信息的重要性:
- 通过改变λ值,研究了上下文信息在分割任务中的重要性。结果表明,适当的上下文信息可以显著提高分割性能。
-
统计显著性:
- 文中提到使用Wilcoxon检验来验证HookNet与其他模型性能差异的统计显著性。对于乳腺癌数据集,HookNet与U-Net和MRN的差异是显著的。
总体而言,Table 2 展示了不同模型在处理乳腺癌组织分割任务时的性能,并强调了HookNet在结合多分辨率信息方面的潜力和优势。
在HookNet模型中,从上下文分支到目标分支的“挂钩”发生在两个分支的特征图具有相同分辨率的相对深度,这取决于输入分辨率。考虑到目标分辨率0.5 μm/px,我们分别从上下文编码器的中间深度2(从中间开始)和上下文解码器的末端深度0(从末端开始)到目标解码器的开始或瓶颈深度4进行“挂钩”。
据我们所知,Gu等人(2018)提出的模型MRN与HookNet最相似。因此,我们将HookNet与MRN进行了比较。然而,HookNet与MRN的区别在于:
- (1)使用“有效”而不是“相同”的卷积
- (2)使用一个额外的包含编码器-解码器的分支(这使得可以多损失模型)而不是一个仅包含编码器的分支
- (3)使用目标分支解码器的单上采样而不是多个独立的上采样
我们使用了一个额外的编码器实例,并将MRN的输入大小设置为256 × 256 × 3。MRN中的卷积使用相同的填充,这导致与使用有效卷积相比输出尺寸更大,从而在每个输出预测中允许更多的像素示例。因此,为了允许MRN在单个GPU上进行训练,我们使用了6个小批量大小而不是12。
所有U-Net模型和只使用单损失(其中λ = 1)的HookNet模型在约2天内完成训练。使用额外上下文损失的HookNet和MRN的训练时间约为2.5天。我们认为,训练时间的增加是由于HookNet中的额外损失和MRN中特征图的大尺寸,这是由于使用了“相同”填充。
所有训练时间都是使用GeForce GTX 1080 Ti和10个CPU进行并行补丁提取和数据增强测量的。
五、结果
在本节中,我们报告了乳腺数据集和肺数据集在语义分割任务中的定量性能。
对于乳腺数据集,我们报告了所有U-Net模型在每个考虑的分辨率下的F1分数以及整体Macro F1(Haghighi等,2018),如表1所示。
对于目标分辨率0.5 μm/px的所有模型(即U-Net(0.5)、MRN和HookNet),我们报告了表2中的定量性能。
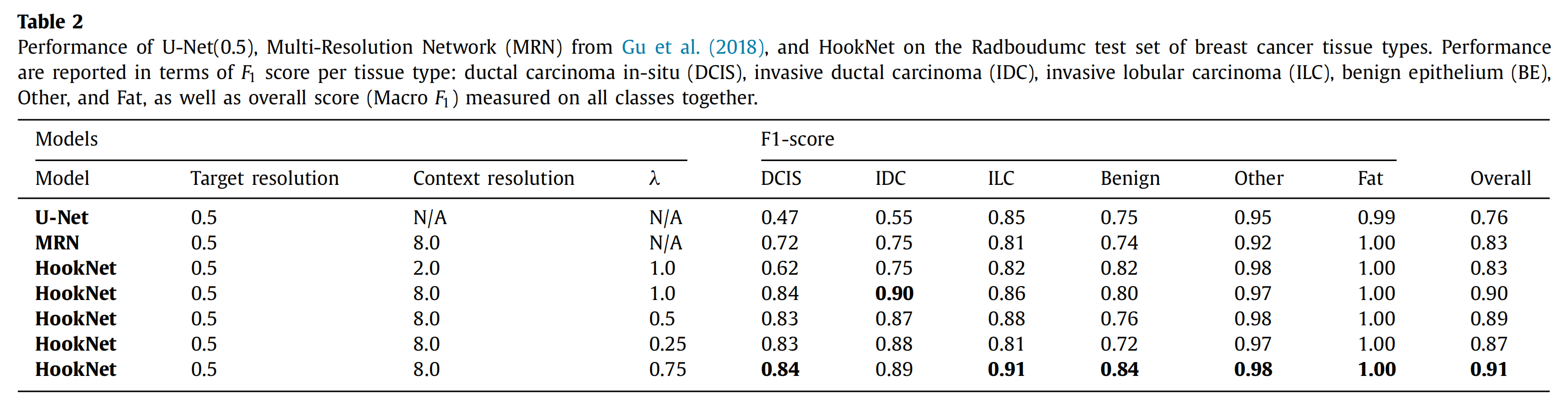
对于肺数据集,所有U-Net模型的定量性能报告在表3中。
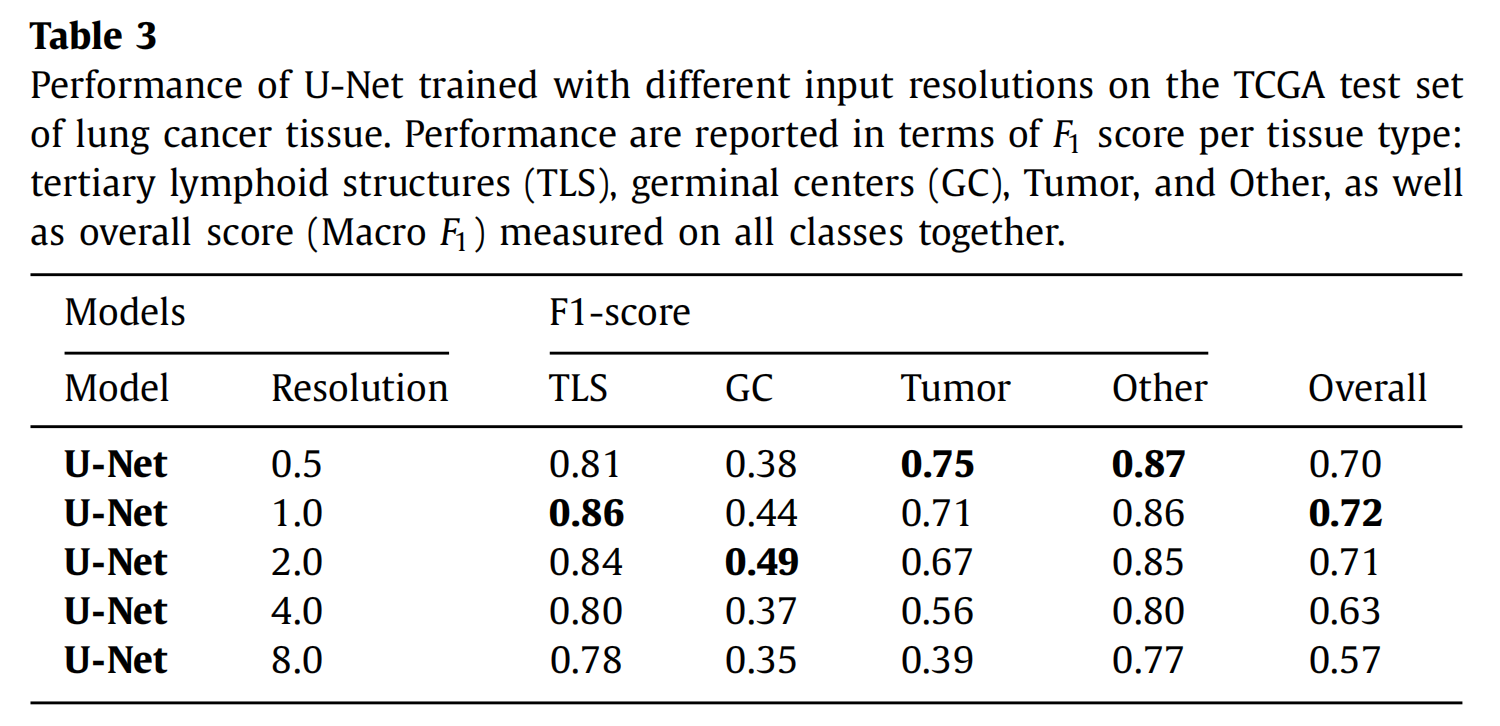
对于目标分辨率0.5 μm/px的所有模型(即U-Net(0.5)、MRN和HookNet),我们报告了表4中的定量性能。
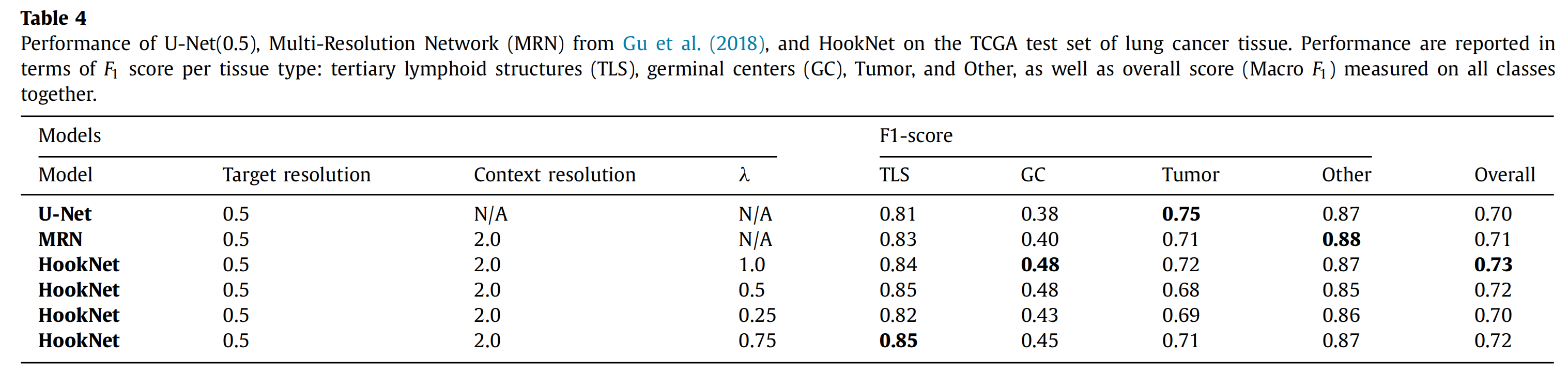
在乳腺数据集中,单分辨率U-Net的实验结果证实了我们的初始假设,即对于DCIS,随着上下文(每像素微米数)的增加,性能与分辨率增加相关(例如,从0.5 μm/px的F1 = 0.47增加到8.0 μm/px的F1 = 0.86),而对于ILC则完全相反(例如,从0.5 μm/px的F1 = 0.85减少到8.0 μm/px的F1 = 0.20),这证实了多分辨率模型的需求。
正如预期的那样,上下文不足导致了U-Net(0.5)中DCIS和IDC之间的混淆,因为乳腺导管结构由于有限的视场而不可见,而细节不足导致了U-Net(8.0)中ILC和IDC之间的混淆(见图4),其中所有松散的肿瘤细胞都被解释为单个团块的一部分。
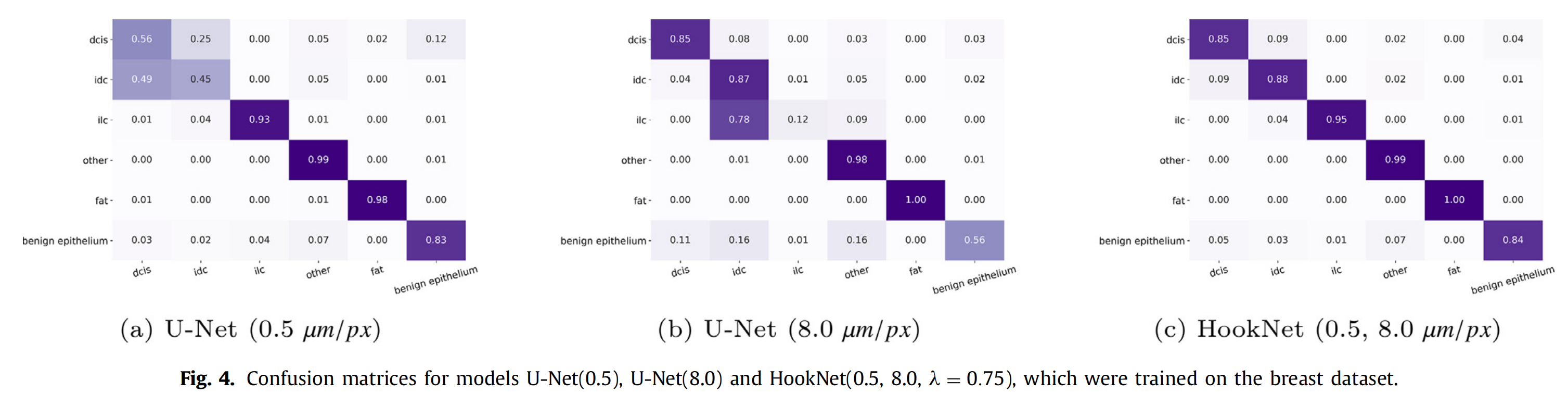
Fig. 4 展示了在乳腺癌数据集上训练的三种模型的混淆矩阵:U-Net(0.5)、U-Net(8.0)和HookNet(0.5, 8.0, λ = 0.75)。
混淆矩阵是一种表格,用于描述分类模型性能的可视化工具,它显示了每个类别的真实标签与模型预测标签之间的匹配情况。
-
混淆矩阵组成:
- 混淆矩阵通常由行和列组成,行表示真实类别,列表示预测类别。
- 矩阵中的每个单元格显示了在特定真实类别和预测类别组合下的样本数量。
-
主对角线:
- 混淆矩阵的主对角线上的值表示正确分类的样本数量。高主对角线值意味着模型具有高分类精度。
-
非对角线:
- 非对角线上的值表示分类错误的样本数量。如果某个非对角线上的值很高,这表明模型在区分这两个类别上存在困难。
-
U-Net(0.5):
- 该模型使用0.5 μm/px分辨率的图像进行训练。混淆矩阵将显示该模型在不同类别上的性能,特别是可能难以区分的类别。
-
U-Net(8.0):
- 该模型使用8.0 μm/px分辨率的图像进行训练,可能在捕捉细节方面存在不足。混淆矩阵将揭示这种分辨率对模型性能的影响。
-
HookNet(0.5, 8.0, λ = 0.75):
- HookNet模型结合了0.5 μm/px和8.0 μm/px分辨率的图像,λ = 0.75表示在损失函数中给予目标分支较高的权重。
- 混淆矩阵将展示这种多分辨率方法如何影响模型在不同类别上的分类性能。
-
性能比较:
- 通过比较三种模型的混淆矩阵,可以分析不同分辨率和多分辨率方法对模型性能的影响。
- 可以观察到在特定类别上,多分辨率方法是否减少了分类错误,提高了模型的准确性。
-
分类挑战:
- 混淆矩阵可以帮助识别模型在哪些类别上存在分类挑战,以及可能的原因。
-
数据集特性:
- 结合混淆矩阵和数据集的特性(如类别不平衡或类别间的相似性),可以更深入地理解模型性能。
-
改进方向:
- 分析混淆矩阵可以为模型的进一步训练和优化提供指导,例如通过调整模型结构、损失函数或数据增强策略。
Fig. 4 中的混淆矩阵是评估模型性能的重要工具,它们提供了对模型在特定任务上强项和弱点的直观理解,有助于指导未来的研究和模型改进。
对于IDC和良性乳腺上皮,最佳的性能是在相对中等的分辨率下观察到的。在所有模型中,脂肪组织的分割性能相当,而其他组织的分割性能在使用相对低分辨率(4.0和8.0 μm/px)或高分辨率(0.5 μm/px)时降低。
对于肺组织,我们观察到随着上下文和分辨率的增加,性能也相应增加。这主要是因为U-Net(2.0)中GC的F1分数增加,类似于DCIS的增加,而细节不足导致了U-Net(2.0)中肿瘤和其他之间的混淆,类似于乳腺组织中ILC的混淆。
在乳腺组织分割中,HookNet的性能强烈依赖于组合的视场。我们获得了HookNet(0.5, 8.0)的最佳结果,总体F1分数为0.91,λ = 0.75,这显著不同于HookNet(0.5, 2.0)。HookNet(0.5, 8.0)对于输出分辨率0.5 μm/px的所有组织类型总体上都有所提高,除了U-Net(8.0)的DCIS性能略有下降外,这主要是由于ILC分割的改进,这可能增加了模型对IDC的精确度。
值得注意的是,DCIS是唯一一个在最低考虑的分辨率下由U-Net表现最佳的类(F1 = 0.86)。然而,U-Net在ILC的F1分数仅为0.2。HookNet(0.5, 8.0)处理相同的低分辨率输入,但与U-Net(8.0)相比,ILC的F1分数提高了0.66,与U-Net(0.5)相比,DCIS的F1分数提高了0.37。
总体上,HookNet(0.5, 8.0)在所有类别上的F1分数都优于U-Net(0.5)和U-Net(8.0),除了DCIS分割的小差异(0.02 F1分数)。对于单U-Net模型,所有HookNet模型在脂肪组织和其他组织类别上的表现相当,如图6所示。
Fig. 6 展示了 HookNet 在乳腺癌组织分割上的结果,包括导管原位癌(DCIS)、侵润性导管癌(IDC)、侵润性小叶癌(ILC)、良性上皮(Benign epithelium)、其他(Other)和脂肪(Fat)组织。
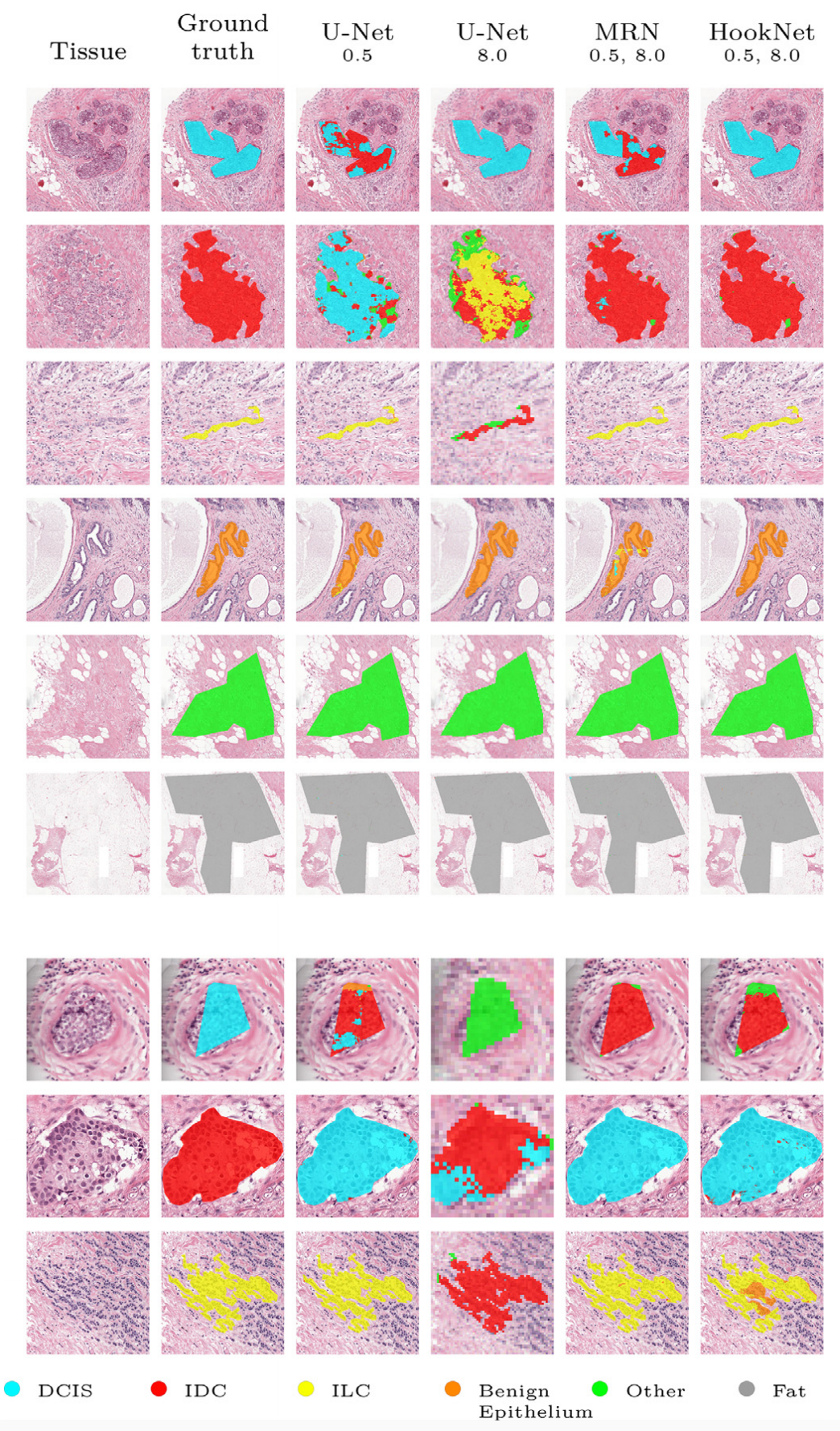
这些结果显示了在 λ=0.75 参数设置下的分割效果。
-
分割效果展示:
- 图片展示了不同组织类型的分割结果,每个组织类型都有其特定的颜色或标记,以便于区分。
-
参数 λ=0.75:
- λ 参数用于平衡 HookNet 模型中目标分支和上下文分支的损失。λ=0.75 表示在损失函数中给予目标分支较高的权重,这可能有助于模型更专注于高分辨率的细节信息。
-
成功案例:
- 前几行的图片可能展示了 HookNet 成功分割的例子,其中不同组织类型的边界清晰,分类准确。
-
失败案例分析:
- 最后三行专注于 HookNet 分割失败的例子,这些案例对于理解模型的局限性和需要改进的地方至关重要。
- 失败案例可能展示了模型在某些情况下无法准确区分不同组织类型,或者在复杂区域中出现了分类错误。
-
错误类型:
- 分析失败案例时,可以识别出不同类型的错误,例如假阳性(将非肿瘤组织错误分类为肿瘤)或假阴性(未能识别出肿瘤组织)。
-
上下文信息的作用:
- 失败案例可能揭示了在某些情况下,模型未能充分利用上下文信息来提高分割精度。
-
模型改进方向:
- 通过分析失败的案例,研究人员可以确定模型改进的方向,比如调整网络结构、优化训练过程或增强特定类型数据的标注。
-
数据集的多样性和复杂性:
- 分割结果的展示也反映了乳腺癌组织在实际样本中的多样性和复杂性,这对于训练鲁棒的模型是一个挑战。
-
可视化工具的重要性:
- 可视化分割结果对于理解模型性能至关重要,它允许研究人员直观地评估模型的准确性和泛化能力。
-
进一步的验证和测试:
- 失败案例需要进一步的验证和测试,以确定是否需要更多的训练数据或更复杂的模型来解决这些问题。
Fig. 6 通过展示 HookNet 在不同 λ 参数设置下的分割结果,提供了模型性能的直观评估,并且通过分析失败案例,为进一步的研究和模型优化提供了宝贵的信息。
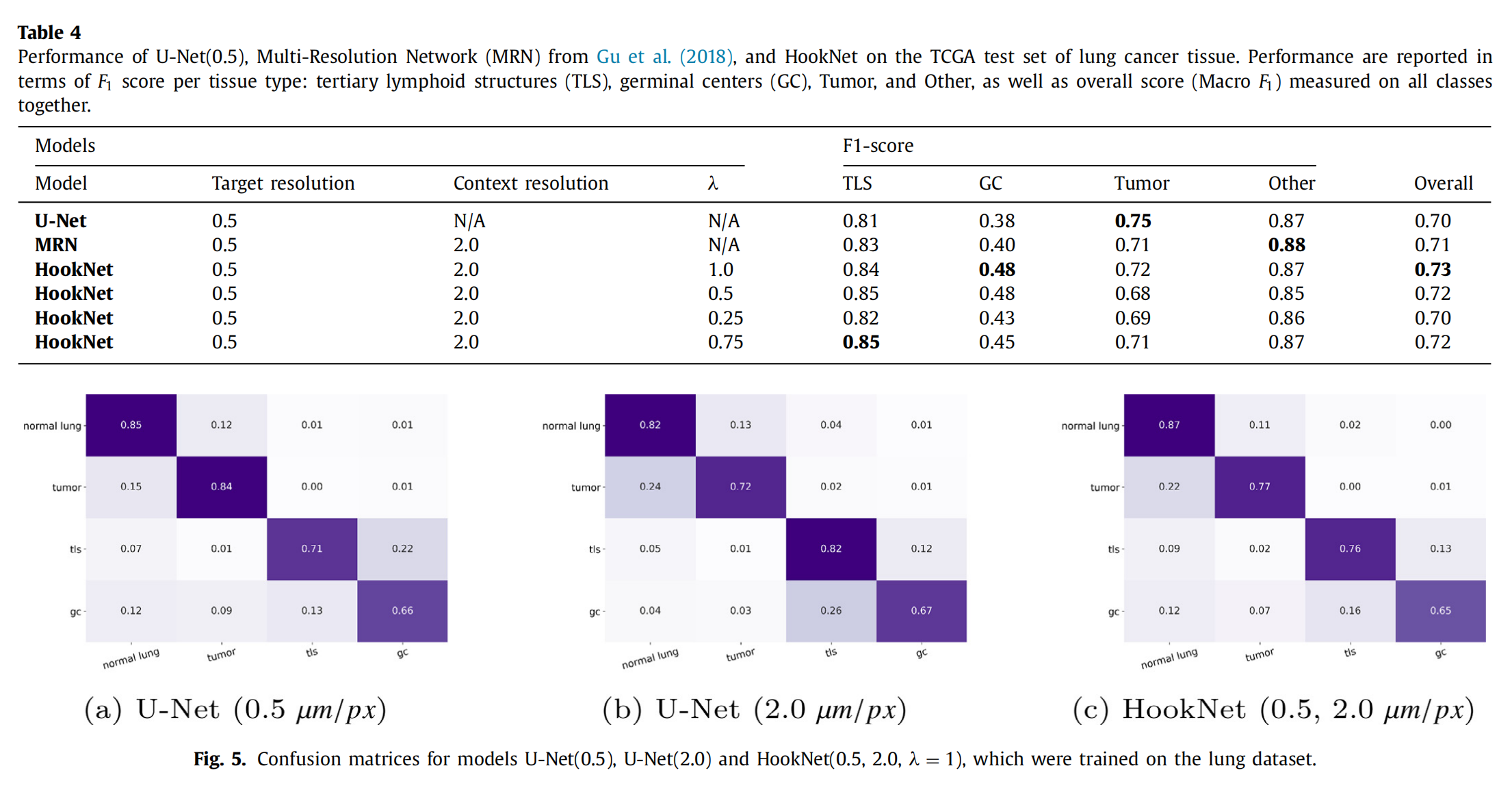
在肺组织分割中,最佳HookNet(λ = 1.0)在TLS和GC类别上优于U-net(0.5),F1分数分别提高了0.03和0.1,而在同一时间,肿瘤的F1分数下降了0.01。两个模型在“其他”类别的F1分数相同。混合不同模型的表现优于HookNet在所有不同的类别上(U-Net(1.0)用于TLS,U-Net(2.0)用于GC,U-Net(0.5)用于Tumor,和MRN用于Other)。然而,HookNet在整体F1分数上优于所有考虑的模型。
我们观察到,HookNet使用与MRN相同的视场,但在乳腺和肺癌组织分割中,HookNet的性能在整体F1分数上优于MRN。最后,我们观察到,对于乳腺组织分割,HookNet(0.5, 8.0)在给予更多重要性给目标分支(即λ = 0.75)时表现最佳,而 for lung tissue segmentation the best F1 scores were obtained when ignoring the context loss (i.e., λ = 1).
为了验证HookNet与其他具有相同目标分辨率(即0.5 μm/px)的模型之间是否存在显著差异,我们计算了每个测试滑片的F1分数,并应用了Wilcoxon检验。结果显示,对于乳腺数据集,HookNet与U-Net(p-value = 0.004)和HookNet与MRN(p-value = 0.001)之间的差异是统计显著的。然而,对于肺数据集,HookNet与U-Net(p-value = 0.442)和HookNet与MRN(p-value = 0.719)之间的差异不具有统计显著性。
这些结果表明,HookNet显著受益于广泛的上下文信息(例如,输入分辨率为8.0 μm/px),而当相关上下文信息受到限制时(例如,输入分辨率为2.0 μm/px),上下文的价值可能不那么明显,但仍然有益。
尽管如此,根据表4中改进的F1分数和图5中的混淆分数,我们主张HookNet可以减少那些受上下文信息影响的类之间的混淆。
六、讨论
本研究论文的主要成果分为两个方面。
第一个成果是一个有效结合组织病理学图像中上下文信息和细节信息的框架。我们展示了它在分割任务中的效果,与其他单分辨率方法相比,以及与最近提出的一个多分辨率方法的比较。
所提出的框架采用MFMR补丁作为输入,并应用一系列卷积和池化层,确保特征图:
(1)相同的空间分辨率进行组合,不需要任意上采样和插值,如Gu等人(2018)所做的那样,但允许直接将上下文分支的特征图拼接到目标分支;
(2)像素级对齐,有效地与有效卷积相结合,这可以减轻输出分割图中伪影的风险。
HookNet两个分支中视场的最佳组合是通过实验确定的。我们首先测试了单分辨率U-Net模型,然后将两个分支中表现最佳的视场组合起来,这两个视场对于特定分割任务的两个关键方面,即乳腺数据集的DCIS和ILC分割,以及肺数据集的肿瘤和GC分割。
目前,不存在事先选择最佳空间分辨率组合的程序,需要进行基于案例的实证分析。
第二个成果包括两个用于乳腺和肺癌组织病理学样本多类别语义分割的模型,这些样本用H&E染色。
在这两种情况下,我们都将肿瘤作为一个要分割的类别,以及可以在肿瘤组织空间中存在的其他类型的组织,并对三种乳腺癌症亚型,即DCIS、IDC和ILC进行了特定的区分。
虽然本研究中使用的乳腺癌和肺癌组织样本中的特定类别作为展示HookNet潜力的应用,但所提出的方法是通用的,可以扩展到任意数量的类别,并且适用于其他器官的组织病理学图像。
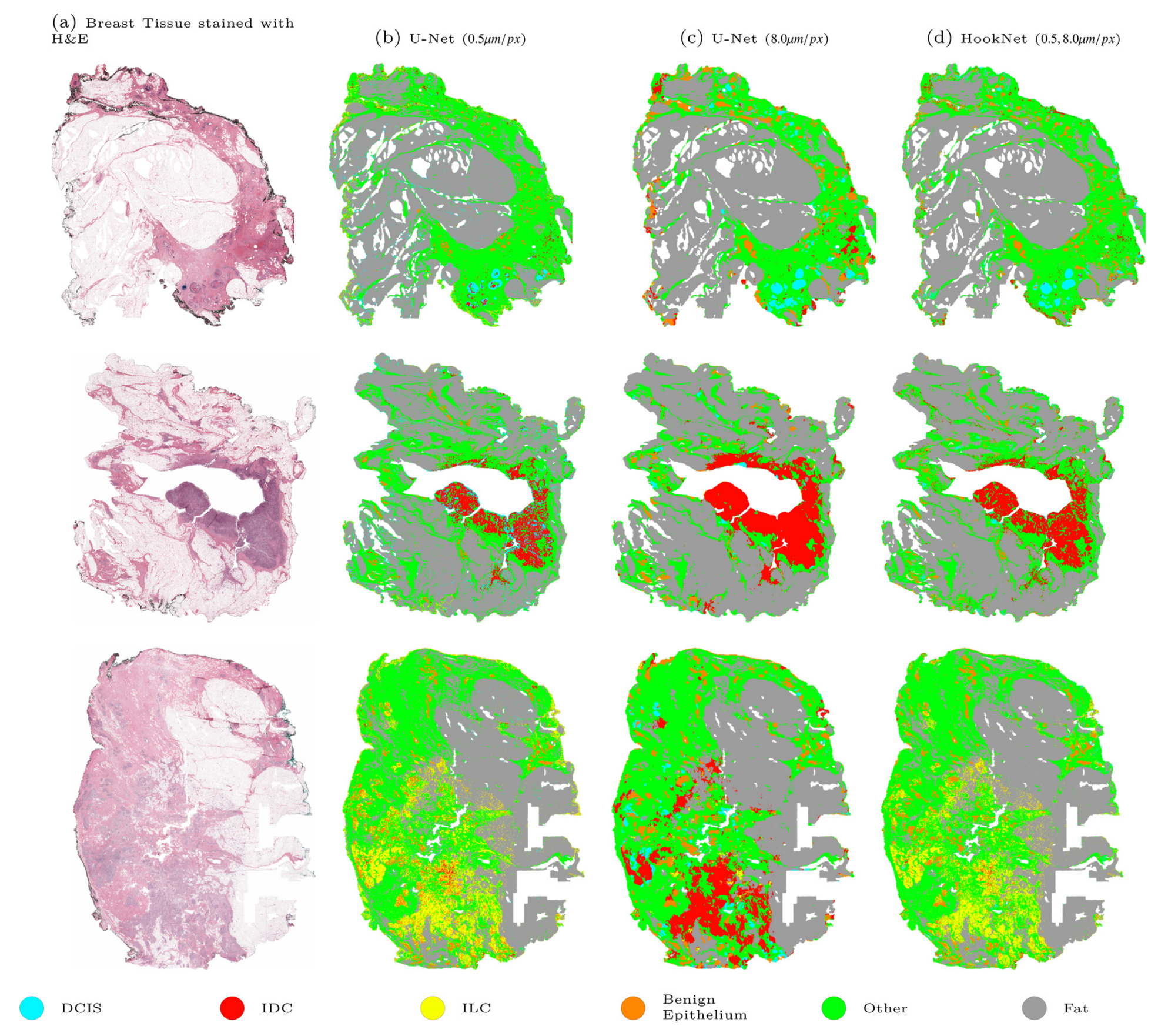
图8展示了分割输出在整张切片图像层面的定性示例,展示了本论文的结果在多个应用中的潜力。
Fig. 8 展示了不同模型对全切片图像(Whole Slide Image, WSI)的预测结果,分别对应于三种不同的乳腺癌类型:导管原位癌(DCIS)、侵润性导管癌(IDC)和侵润性小叶癌(ILC)。
-
WSI预测展示:
- 每个模型对同一类型的乳腺癌WSI进行了预测,结果在不同的行中展示,以便于比较。
-
组织类型的特异性:
- DCIS、IDC和ILC是三种不同的乳腺癌亚型,它们在组织形态和细胞特征上有所区别。
-
模型性能比较:
- 通过在同一行中展示不同模型对同一种类型乳腺癌的预测,可以直观地比较各个模型的性能。
-
分割准确性:
- 预测结果的准确性可以通过观察模型是否能够准确地识别和区分不同类型的组织来评估。
-
颜色和标记:
- 不同的组织类型可能使用不同的颜色或标记来区分,以便于视觉识别和评估。
-
上下文和细节:
- 预测结果展示了模型如何在保持组织结构上下文的同时捕捉到重要的细胞细节。
-
DCIS预测:
- 第一行展示了针对DCIS的预测结果,可能显示了模型在识别导管内肿瘤细胞方面的性能。
-
IDC预测:
- 第二行展示了针对IDC的预测结果,可能突出了模型在区分侵润性肿瘤和周围组织方面的能力。
-
ILC预测:
- 第三行展示了针对ILC的预测结果,可能揭示了模型在识别小叶癌特征方面的效果。
-
模型的泛化能力:
- WSI预测结果反映了模型对不同类型乳腺癌的泛化能力,即模型在实际应用中的潜在效果。
-
临床相关性:
- 准确的WSI预测对于临床决策和治疗计划的制定具有重要意义,因此这些结果对于病理学家和研究人员来说非常有价值。
-
进一步研究的指导:
- 如果某些模型在特定的癌症亚型上表现不佳,这可能指导未来的研究,以改进模型或增加特定类型的训练数据。
Fig. 8 中的WSI预测结果提供了对不同模型在乳腺癌组织学图像分割任务上性能的全面评估,有助于理解模型在实际临床应用中的潜力和局限性。
肺癌鳞状细胞癌中TLS和GC的分割可以用于自动检测肺癌组织病理学图像中的TLS,这将使我们能够轻松地扩展分析到大量案例,以进一步研究TLS计数的预后和预测价值。
同时,肿瘤和其他组织类型的分割可以描述肿瘤微环境中的形态和组织结构,例如确定肿瘤主体的区域,或者确定肿瘤与基质之间的界面,这是免疫肿瘤学研究中的一个活跃话题,因为肿瘤浸润淋巴细胞(TILs)在肿瘤相关基质(Salgado等人,2015)以及肿瘤主体和浸润边缘(Galon等人,2006;2014)中的作用必须评估。
此外,乳腺癌中良性细胞和恶性上皮细胞的分割可以作为乳腺癌分级自动化流水线的第一个步骤,其中必须识别肿瘤区域以进行有丝分裂计数,并将健康和癌症上皮细胞区域进行比较,以评估核多形性。
为了展示多分辨率方法在组织病理学图像语义分割中的优势,与单分辨率模型相比,本文做出了一些设计选择。
我们的未来研究将专注于研究HookNet相对于使用约束的一般适用性和设计。
首先,U-Net被用作HookNet分支的基础模型。这一选择是基于编码器-解码器U-Net模型的有效性和灵活性,以及存在跳过连接。其他编码器-解码器模型也可以被采用来构建HookNet模型。其次,受WSI多分辨率特性的启发,我们开发并仅将HookNet应用于组织病理学图像。
然而,我们主张HookNet具有潜在的通用性,可以用于任何需要结合上下文和细节以产生准确分割图的应用。HookNet在医学成像中可以找到多种应用,但它也有潜力扩展到自然图像。
第三,我们展示了使用两个分支可以利用单分辨率模型在乳腺癌数据中的明显趋势,如DCIS和ILC(见图1)。然而,当我们专注于IDC类时,我们注意到单分辨率U-Net在中间分辨率下表现最佳。这促使我们进一步研究包含更多分支,以包括中间视场。
第四,我们将HookNet,以及用于比较的模型限制为5000万个参数,这允许使用具有11 GB RAM的单个现代GPU进行模型训练。引入更多分支可能需要采用多GPU方法,以便进行更深的/更宽的网络实验,并加快推理时间。
我们将HookNet与单损失(λ = 1)和多损失设置(λ = 0.75、0.5或0.25)进行了比较。
我们的结果显示,当给予目标分支更多重要性(例如,λ = 0.75)时,多损失模型在乳腺癌组织分割中表现最佳,而单损失模型(例如,λ = 1.0)在肺组织分割中得分最高。
未来的工作将重点进行广泛的优化搜索,以确定λ的值。最后,我们根据稀疏手动注释报告了Radboudumc和TCGA数据集的结果,以F1分数的形式。
尽管这是在医学成像中获取大量异构数据集的常见方法,但我们观察到这种方法限制了在不同组织类型过渡区域中性能的评估。将评估扩展到额外的一组密集注释数据也是我们未来的研究以及生成此类手动注释的努力。