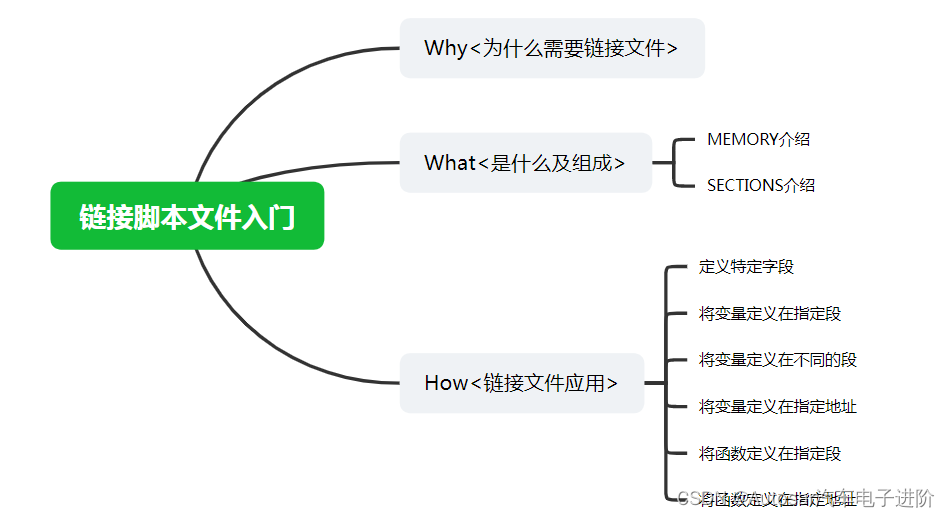
将NMF应用于之前用过的Wild数据集中的Labeled Faces。NMF的主要参数是我们想要提取的分量个数。通常来说,这个数字要小于输入特征的个数(否则的话,将每个像素作为单独的分量就可以对数据进行解释)。
首先,观察分类个数如何影响NMF重建数据的好坏:
import mglearn.plots
import numpy as np
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.datasets import fetch_lfw_people
people=fetch_lfw_people(data_home = "C:\\Users\\86185\\Downloads\\",min_faces_per_person=20,resize=.7)
image_shape=people.images[0].shape
mask=np.zeros(people.target.shape,dtype=np.bool_)
for target in np.unique(people.target):
mask[np.where(people.target==target)[0][:50]]=1
X_people=people.data[mask]
y_people=people.target[mask]
X_train,X_test,y_train,y_test=train_test_split(X_people,y_people,stratify=y_people,random_state=0)
mglearn.plots.plot_nmf_faces(X_train,X_test,image_shape)
plt.show()
反向变换的数据质量与使用PCA时类似,但要稍差一些。这是符合预期的,因为PCA找到的是重建的最佳方向。NMF通常并不用于对数据进行重建或编码,而是用于在数据中寻找有趣的模式。
尝试仅提取一部分分量,初步观察一下数据:
from sklearn.decomposition import NMF
nmf=NMF(n_components=15,random_state=0)
nmf.fit(X_train)
X_train_nmf=nmf.transform(X_train)
X_test_nmf=nmf.transform(X_test)
fig,axes=plt.subplots(3,5,figsize=(15,12),subplot_kw={'xticks':(),'yticks':()})
for i,(component,ax) in enumerate(zip(nmf.components_,axes.ravel())):
ax.imshow(component.reshape(image_shape))
ax.set_title('{}.component'.format(i))
plt.show()

这些分量都是正的,因此比PCA分量更像人脸模型。例如,上图分量9显示了稍微向右转动的人脸,分量12显示了稍微向左的人脸。
再来看一下一些分量特别大的图像:
compn=9
inds=np.argsort(X_train_nmf[:,compn])[::-1]
fig,axes=plt.subplots(2,5,figsize=(15,8),subplot_kw={'xticks':(),'yticks':()})
for i,(ind,ax) in enumerate(zip(inds,axes.ravel())):
ax.imshow(X_train[ind].reshape(image_shape))
plt.show()
compn=12
inds=np.argsort(X_train_nmf[:,compn])[::-1]
fig,axes=plt.subplots(2,5,figsize=(15,8),subplot_kw={'xticks':(),'yticks':()})
for i,(ind,ax) in enumerate(zip(inds,axes.ravel())):
ax.imshow(X_train[ind].reshape(image_shape))
plt.show()

正如所料,分量9系数较大的都是向右看的人脸,分量12系数较大的人脸都是向左看。提取这样的模式最适合与具有叠加结构的数据,包括音频,基因表达和文本数据。