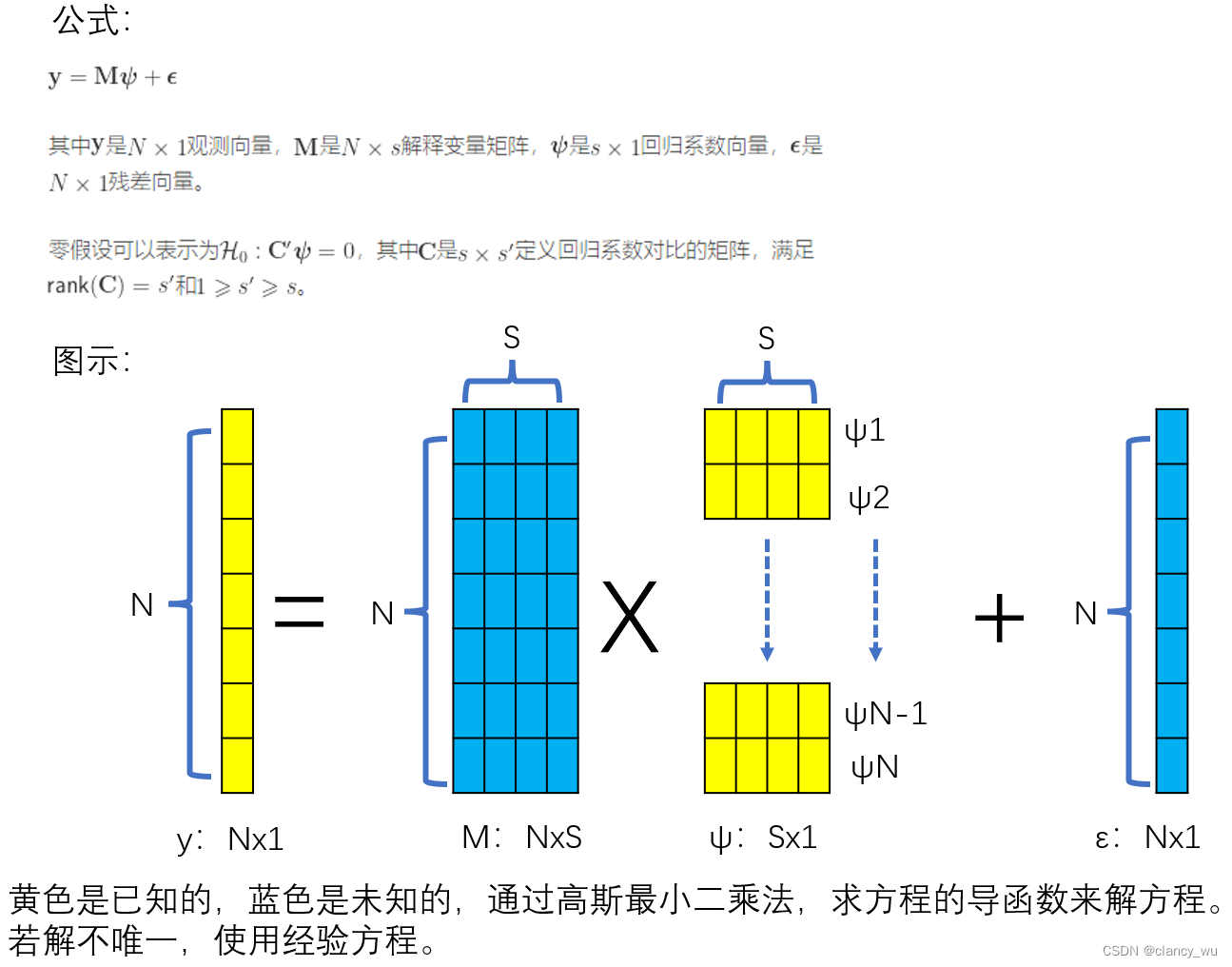
下面是我对GLM模型的理解:
数据编码的方式
在一般统计中,常用的coding方式有dummy,effect和cell.mean,这个在R和python中都可以实现。
dummy coding 举例
假设有4个组别A, B, C, D,它的自由度是4-1=3,因此它可以用3个不同位置的1来编码代表4个组(有一个组作为reference组,其编码全为0).
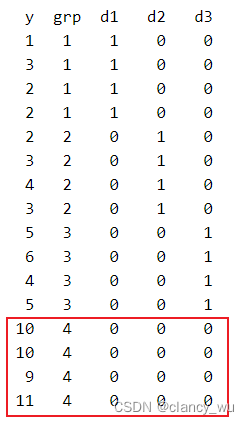
假设如下的表格数据:

把g4组作为参考组,使用dummy coding转换后如下:
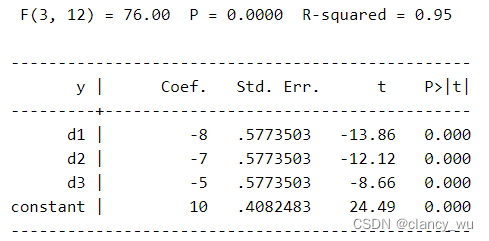
如此以后,进行F检验,得到的值,其截距就是参考组的平均值。而其他系数就是其均值差,例如-2=8-10,-7=3-10等等。
这个在python中就是独热编码过程。
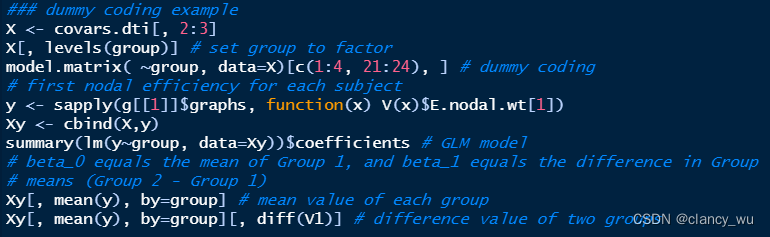
R中就是如下:
effect coding 举例
还是考虑上述数据,使用effect coding转换后如下:
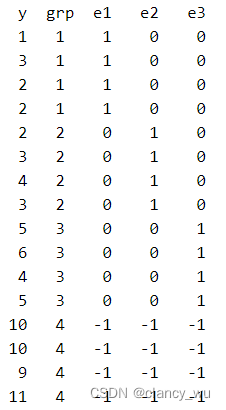
同样以g4组为参考组,所以能够看出,effect coding 和 dummy coding的区别在于,前者参考组为[-1, -1, -1]的矩阵,后者参考组为[0, 0, 0]的矩阵,其他就没有区别。

在effect coding后,算出来的截距是所有组平均值的平均值,即
y
‾
\overline{y}
y (等于5)。这个时候每组的coef,是其组的平均值和所有值的总平均值(constant值)的差值,即-3=2-5, -2=3-5。
不平衡的数据使用effect coding的注意事项
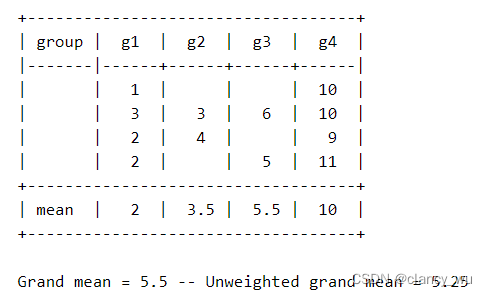
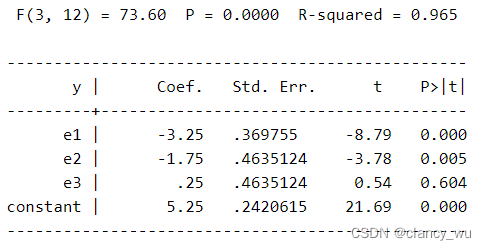
不平衡数据算出来的截距,是所有组平均值的平均值,而不是所有观察值的平均值。在这个数据中,一共有12个观察值,把12个值相加除以12,其平均值是5.5。而若把每一组进行组内平均,再把所有组的平均值进行平均,得到的值就是5.25,也就是F检验中的截距。
这个是需要注意和记住的地方。通过这个例子,也会对参数检验中对比均值的F检验有一个更为深刻的认识,即不管数据平衡与否,它只比较每组的平均值有无差异。很好用。
如何选择dummy coding和effect coding,两者区别?
如果模型中有多个分类变量,dummy coding 和 effect coding没有区别,只需要注意截距和系数的意义就行。
但是如果只有2种分类变量,dummy coding (编码0)和effect coding(编码-1)是有区别的,即effect coding的编码方式可以分析两个变量的交互效应。原理:设想一下,当分类变量只有2个时,比如病人组和健康人组,那么使用dummy coding只有1和0,而使用effect coding 有1和-1,中间还有0态,所以effect coding可以分析交互作用。因此这个时候使用effect coding可以获得对简单效应和交互效应的合理估计。
仔细回想一下,FSL的FEAT模块就是通过上述的方式去编码matrix design的。
R中如下:
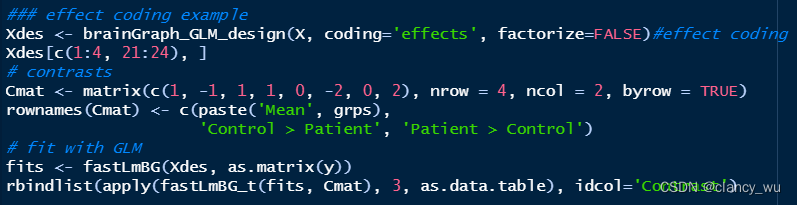
fastLmBG (fast lm brain graph) solve the least squares problem to estimate model coefficients, residuals, etc. for brain network data.
fastLmBG_t and fastLmBG_f calculate contrast-based statistics for T or F contrasts, respectively. It accepts any number of contrasts (i.e., a multi-row contrast matrix).
cell means coding 举例
SPM里的2-nd-level comparison就是cell means coding方法,这个方法只用于组别为2的数据。
因为只有2个组,所以不需要设置参考组,所以就是用[1, 0]和[0, 1]来表示两个组,然后design matrix对应着就是1-0=1和0-1=-1。
与上述两个方法相比,这个是对比均值差异,没有设置参考组,较为简单。
- 单元格均值模型仅用于使估计语句中的对比更易于编写的目的。唯一考虑的输出部分是与对比度估计相关的部分(这通常位于输出的底部)。
- 用单元格编写估计语句意味着模型更容易,因为它只包含一个向量(用于最高阶交互)。使用分析模型,同一对比度的估计语句可能包含多个向量和/或矩阵,因此更难以正确指定。
在R中的实现:
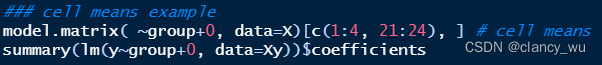
model.matrix是R自带的base函数,可以实现dummy和cell means coding,前者就是常规的 y —— g r o u p y —— group y——group,后者是 y —— g r o u p + 0 y —— group+0 y——group+0。
brainGraph_GLM_design是brainGraph包的函数,其中coding方式可以选择dummy,cell meaning 和 effects三种方式。
图论中的常用指标
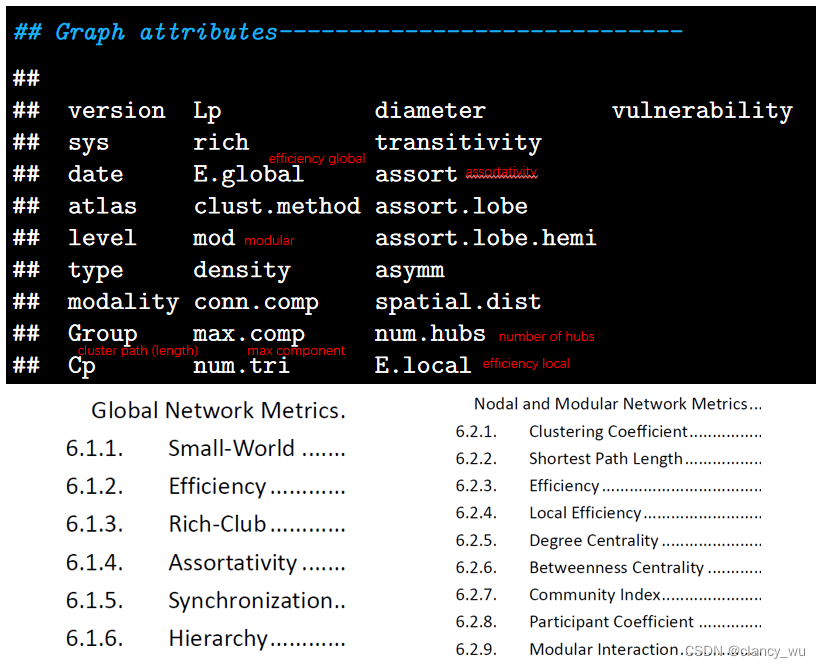
图的常用指标
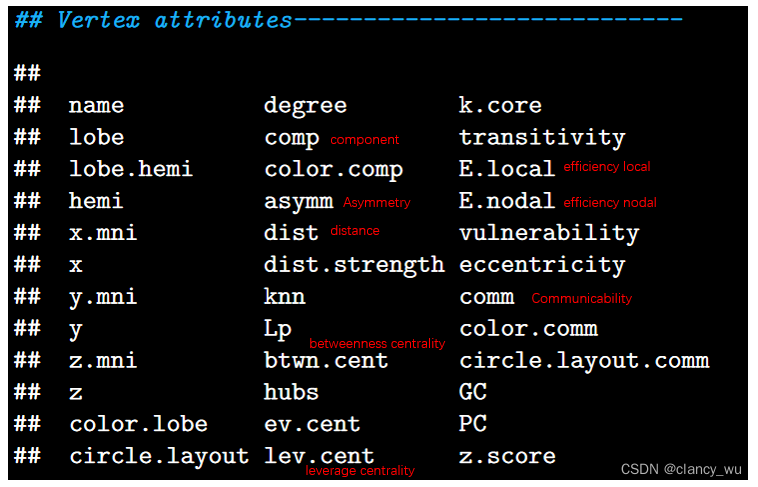
顶点的常用指标
边的常用指标
以上这些指标还可以加入.wt来查看weighted值和非weighted值(binary)。
GLM实例
我们在创建图的时候,常规会使用50个阈值【0.10,0.11.。。0.49,0.50】去生成图,DPABI和GRETNA设置的阈值参数会有些许差异,但是过程都是一样。
在GLM分析中,我们只能在单一阈值下去使用GLM模型去比对单一指标。在使用GLM比较时,一共有2步需要做。第一是创建design matrix,第二是根据design matrix进行GLM统计分析。(整个过程跟FSL的FEAT一模一样,可以相互参考)
以我现在手头的数据为例,我的数据是34:37的数量,healthy group和patient group。
两组比较-不考虑混淆因素
(1)首先是基础组别信息
(2)根据组别信息创建design matrix
CompareMatrix <- matrix(c(0, -2), nrow = 1, dimnames = list('Control > Patient'))
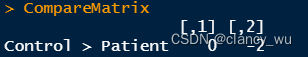
(3)根据design matrix,进行GLM分析
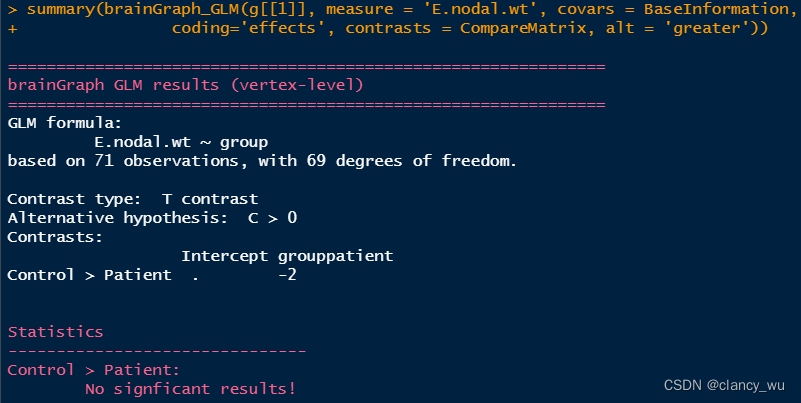
上面是单侧的分析。其实双侧的分析与上面同理,比较简单,双侧分析如下:
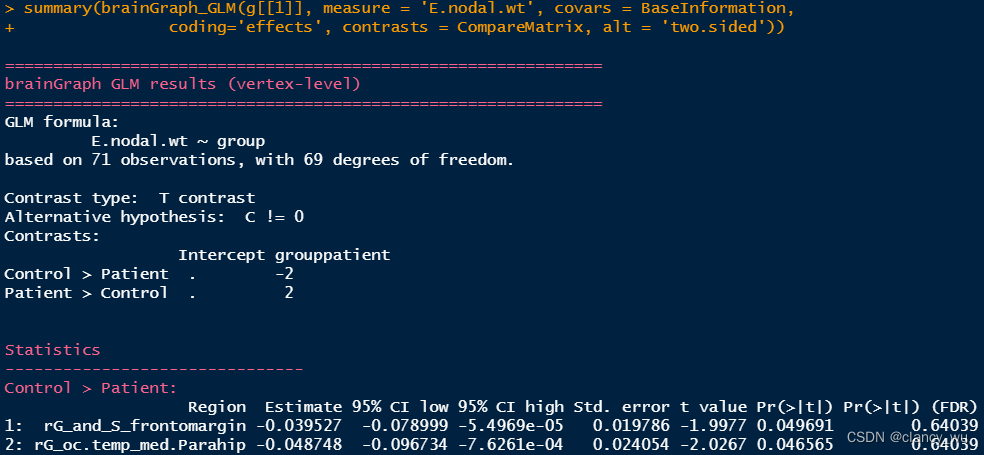
比较好的一点是,它会告诉FDR校正的结果。
两组比较-考虑混淆因素
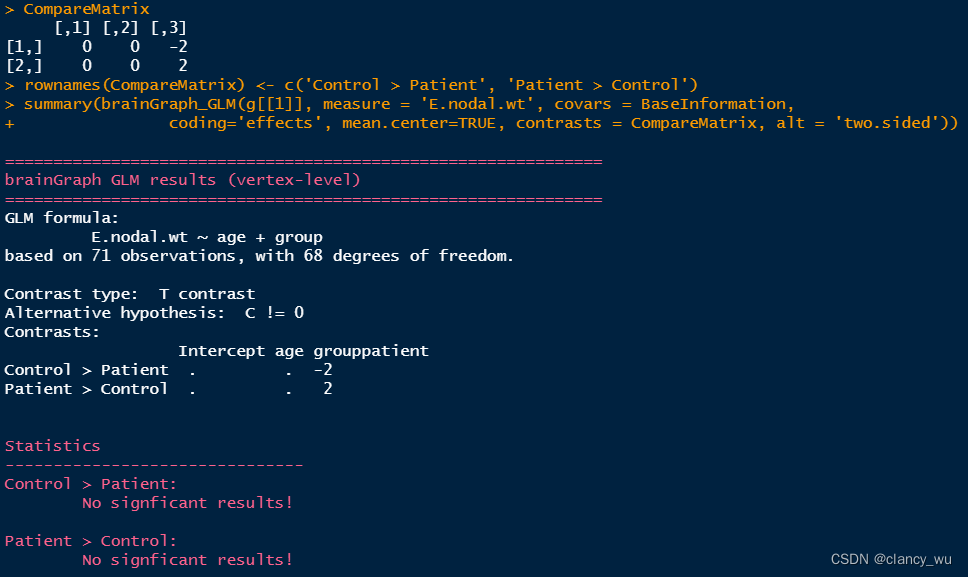
值得注意的是,有多少变量,CompareMatrix就要有多少列,函数会自动把group放到最后作为对比。