瘤胃微生物组一直以来都是研究相对不足但又极其复杂的微生物生态系统之一。瘤胃微生物能够有效降解植物纤维,将其转化为高质量的蛋白质产品,在这一过程中,由于微生物强烈的发酵,还会产生大量气体,其成分主要包括二氧化碳和甲烷等温室气体,还有少量的氮气和微量的氢气、氧气和硫化氢,这些排放的温室气体甚至会对全球环境造成较大的影响。近些年以来,在宏组学技术的推动下,对瘤胃微生态系统功能的认识已取得了显著进展。然而,复杂微生态系统的功能冗余和异质性仍然是实现突破性进展的主要障碍。为了克服这些挑战,我们亟需新的方法来提高微生物组功能研究的分辨率。
2022年3月,M20 Genomics突破性地推出VITA单细菌转录组测序技术(MscRNA-seq),该方法创新性地使用随机引物对RNA分子进行捕获,极大地推动了单细胞技术在微生物领域的应用。多篇SCI验证产品力!凌恩生物携手M20 Genomics微生物单细菌转录组!(点击查看详细内容)。
近日,M20 Genomics助力浙江大学孙会增研究团队在顶尖学术期刊Nature Microbiology (影响因子:28.3) 上发表题为《Single-cell transcriptomics across 2,534 microbial species reveals functional heterogeneity in the rumen microbiome》的学术论文[4],该研究首次成功地将M20 Genomics开发的VITA单细菌转录组测序技术应用于牛瘤胃微生物组研究,并构建了迄今为止最全面的牛瘤胃微生物组泛基因组数据库(Bovine Gastro Microbial Genome Map,以下简称“BGMGM”)。通过结合VITA单细菌转录组测序技术和基于BGMGM的算法,揭示了牛瘤胃微生物组的功能异质性。这项研究对于解决生态动力学、酶资源的勘探以及推动木质纤维素生物燃料的大规模工业化生产等全球性问题具有重要的实际意义。

01、构建牛瘤胃微生物组的参考泛基因组
相对于人类大规模且成熟的肠道微生物泛基因组研究,对牛胃肠道的相关研究较为有限。微生物组的单细菌转录组研究需要参考泛基因组进行物种注释和基因比对分析。因此,研究者利用公开数据和新测得的宏基因组数据构建了目前最大的牛胃肠道微生物泛基因组数据库,将其命名为BGMGM。BGMGM涵盖了2,311个动物样本,覆盖了10个不同的胃肠道段,其中以瘤胃为主要来源。该数据库包含13,572个非冗余基因组,包括5,676个高质量(HQ)和7,896个中等质量(MQ)基因组,其中的7,545个基因组被鉴定为已知物种(厚壁菌门、拟杆菌门、假单胞菌门等)。BGMGM含有23,755,235个非冗余基因,其中18,046,712个基因至少具有1个数据库的功能注释。作为高质量的基因组集合,BGMGM拥有丰富的注释基因和高水平的基因注释率,为牛瘤胃单细菌转录组数据的功能研究提供了坚实的基础。
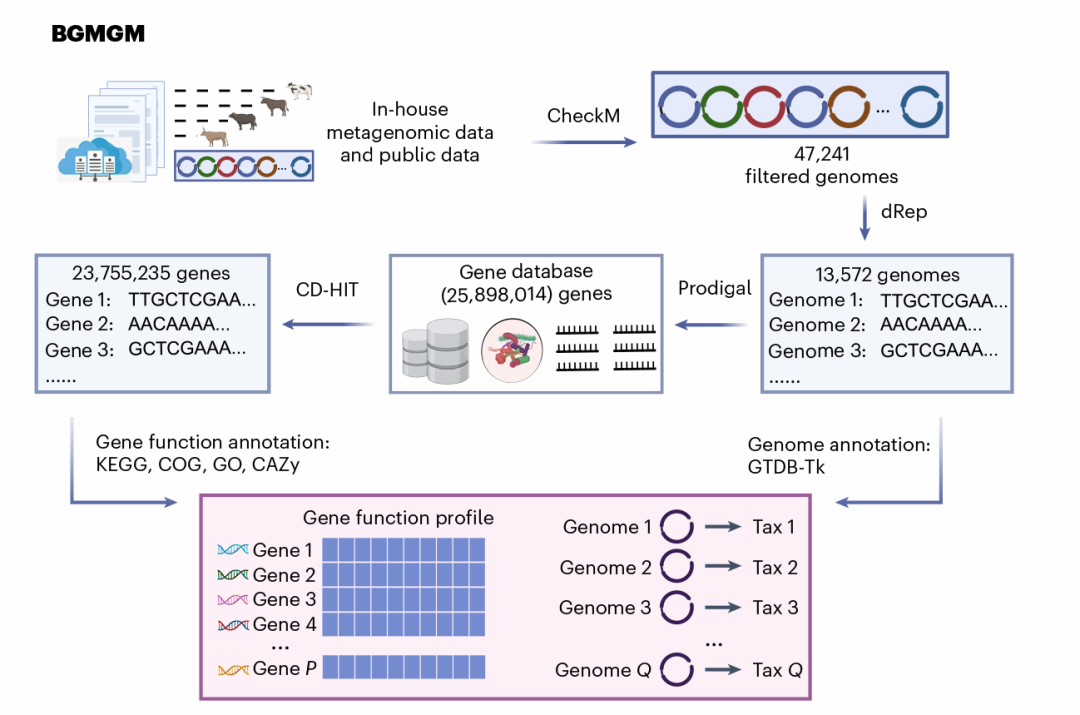
图1.BGMGM搭建流程
02、瘤胃微生物组单细菌功能景观图
研究者基于M20 Genomics开发的VITA单细菌转录组测序平台,捕获了超过200,000个瘤胃细菌。经过严格质控,保留了174,531个高质量细菌,基因中位数为182。研究者对所有细菌进行降维聚类分析,鉴定出12个功能簇,并基于特异性表达基因的生物学功能进行注释(图2A)。12个功能簇代表了瘤胃微生物在单细菌RNA水平上的功能分类,不同样本之间高度重叠,证明了功能簇分析策略的稳健性和可重复性。
该研究共鉴定出2,534种瘤胃微生物,其中172个被识别为核心活性物种(>100个细菌)(图2B)。该研究发现,164种核心活性物种分布在一个以上的功能簇中,38种核心活性物种存在于所有功能簇中。某些物种优先参与特定的功能簇。例如,Ruminococcus_D sp900104495主要存在于Sulfur MCs功能簇中,而Prevotella sp902762325主要存在于RPA+ Lipid MCs功能簇中(图3C)。
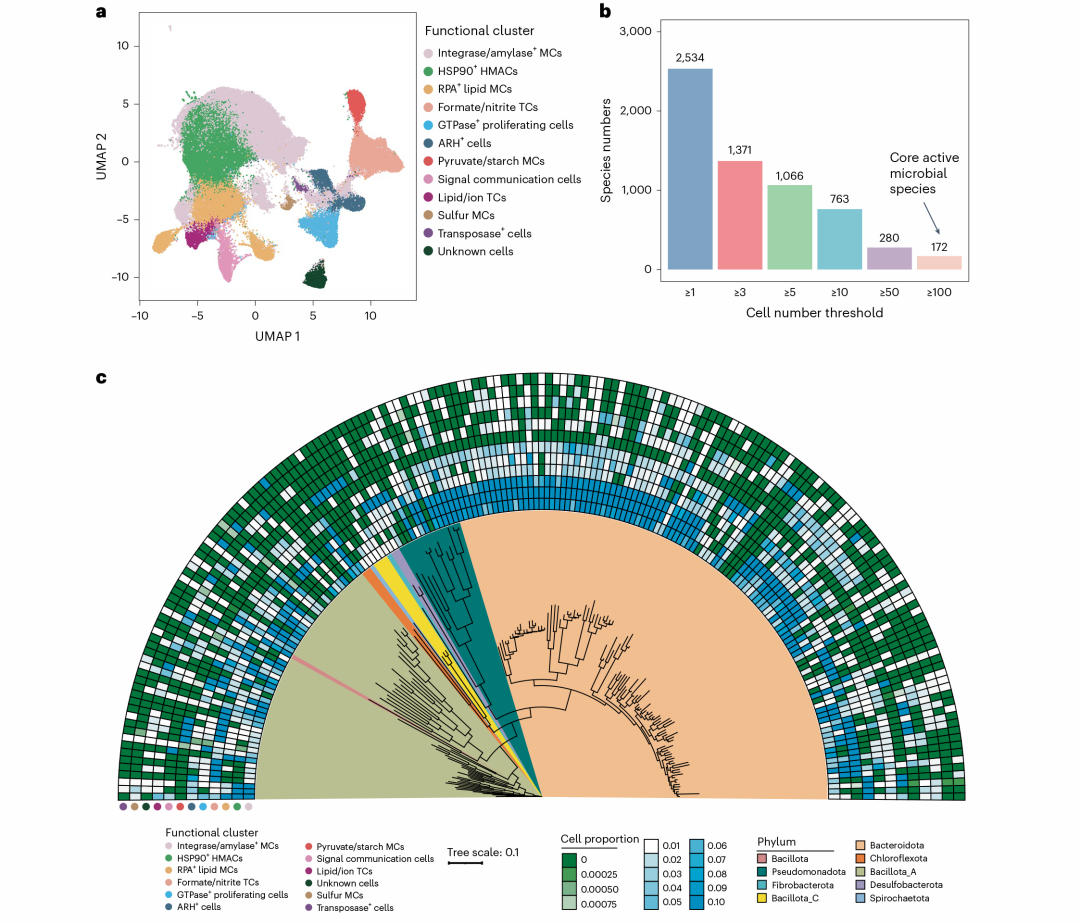
图2. 瘤胃微生物组单细菌功能景观
03、功能簇的异质性和相互作用
从瘤胃单细菌转录组数据中鉴定了12个功能簇后,研究者一步表征其标记基因,并基于标记基因的注释,展示了每个功能簇特定的生物途径(图3B)。例如,Sulfur MCs功能簇特异性表达基因CowSGB-4309-c57-3和CowSGB-4309-c116-5,它们编码的AprA和DsrA是参与硫代谢生物途径的两种重要蛋白。因此,我们将该功能簇命名为Sulfur MCs,并在图3B中展示了硫代谢的生物途径。同一功能簇的物种组成差异很大,反映出不同物种可能具有相似的功能活性。同时,来自同一物种的细菌分布在不同的功能簇中,这表明同一物种内的不同细菌个体功能活性不同。功能簇的功能异质性源于基因表达的变化,可能由物种组成差异和单个细菌活性的差异决定。
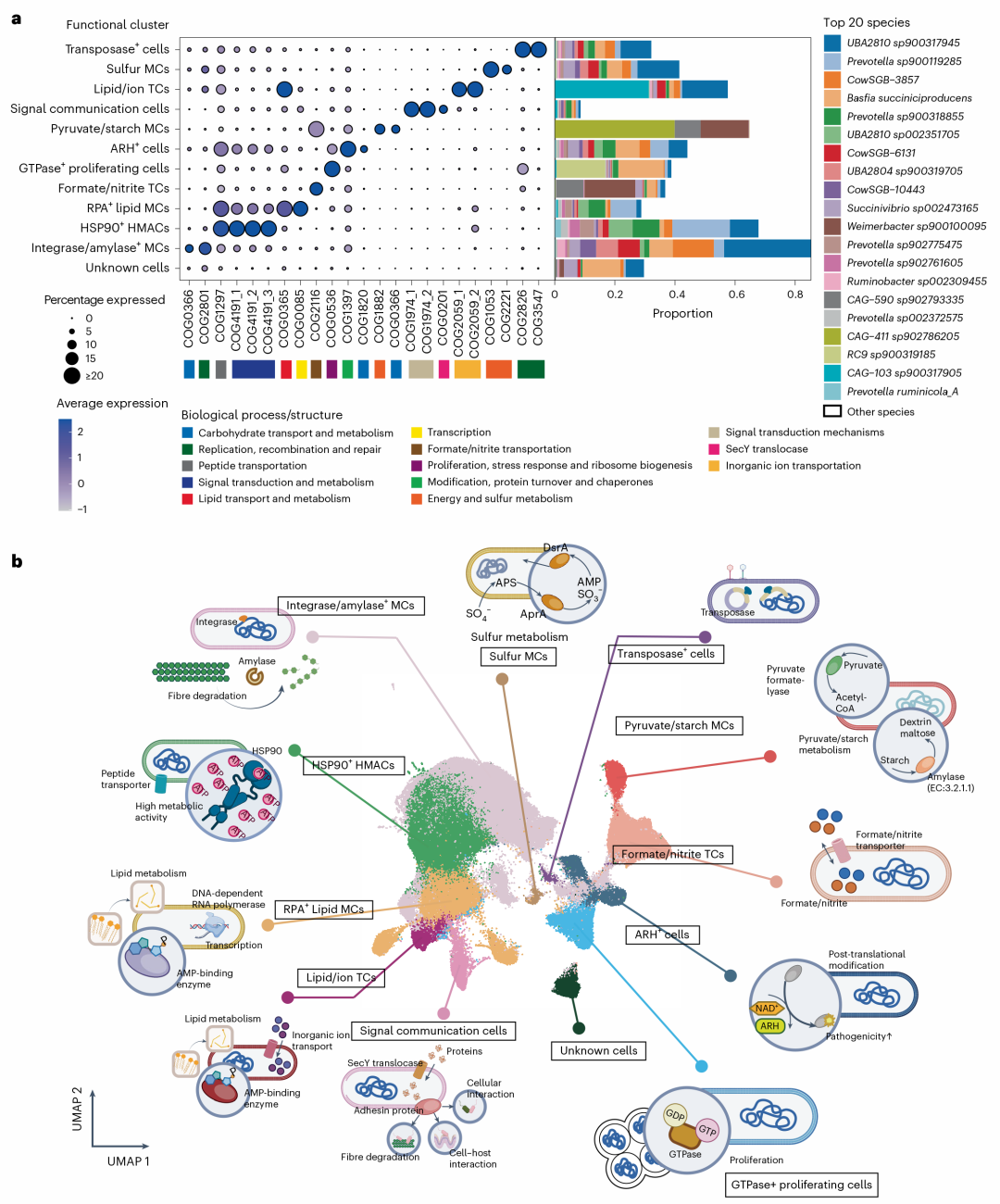
图3. 功能簇的通路异质性
基于上述发现,研究者进一步探讨同一物种的功能异质性。从所有细菌中筛选出了89个物种。通过计算单个细菌中的功能基因比例(FGP)来描述每个细菌特定途径的功能活性。在热图中展示了其中8个物种COG功能在不同功能簇之间差异P值,发现功能簇间的差异因物种而异,表明关键物种在瘤胃中的功能作用发生了显著变化(图4A)。例如,Basfia succiniciproducens(产琥珀酸)有17个COG分类的活性,在不同功能簇之间显示出显著差异。结果表明,B. succiniciproducens在不同的功能簇中的细菌个体发挥着不同的作用。
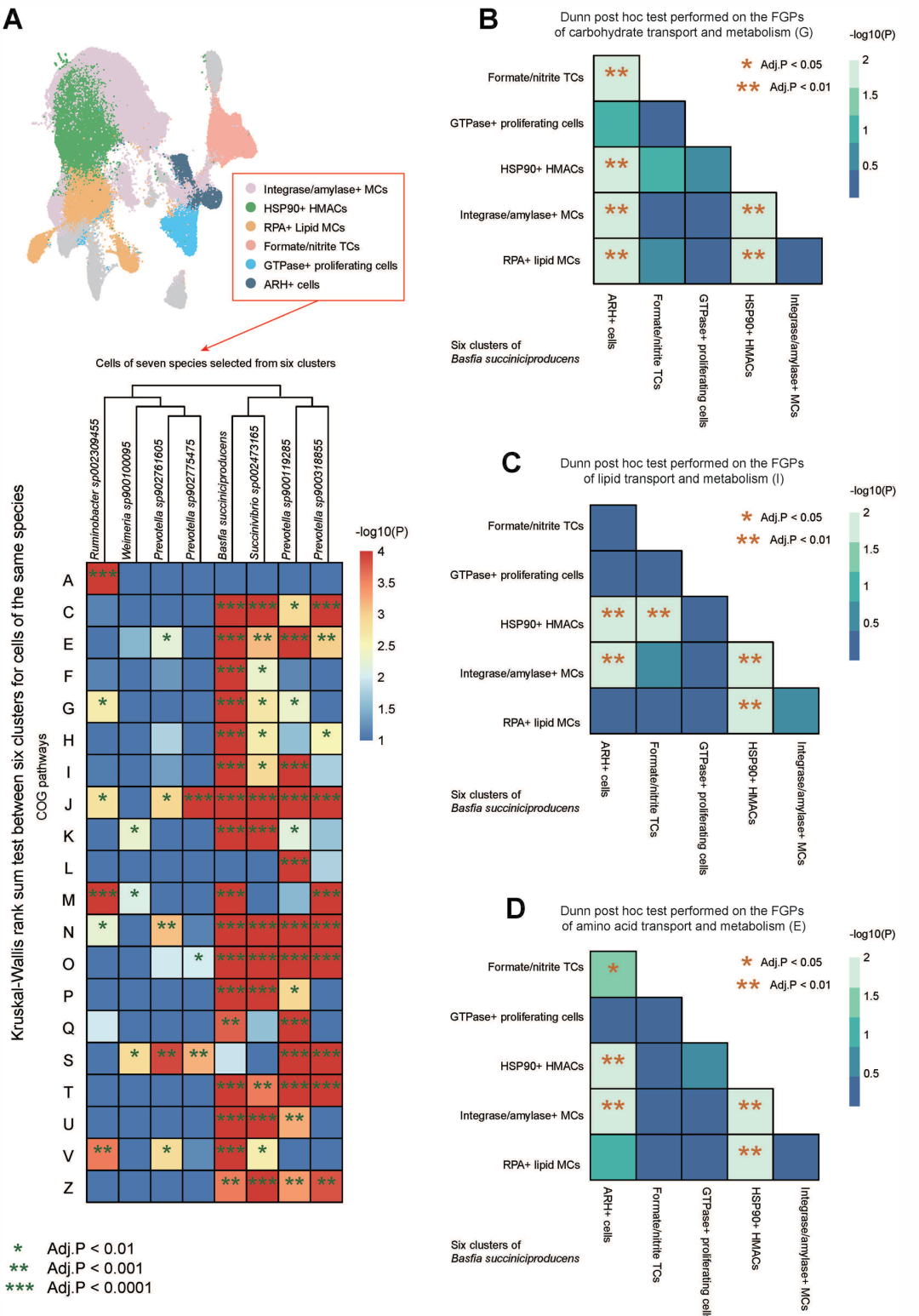
图4. 物种内部功能异质性研究
为了方便描述,研究者以“功能簇-物种”的形式表征特定细菌单元,如HSP90+HMACs-B.succiniciproducens表示HSP90+ HMACs功能簇中的B. succiniciproducens,并对它们进行互作网络分析。研究者选取了213个细菌单元(分布在3个以上的样本中,每个样本至少有10个细胞)。这些细菌单元之间总共有519个相互作用,HSP90+HMACs-B.succiniciproducens和其他六个单元之间发现了相互作用,包括来自相同/不同功能簇和物种的单位,表明功能簇之间和物种之间存在广泛的生态关联(图5)。
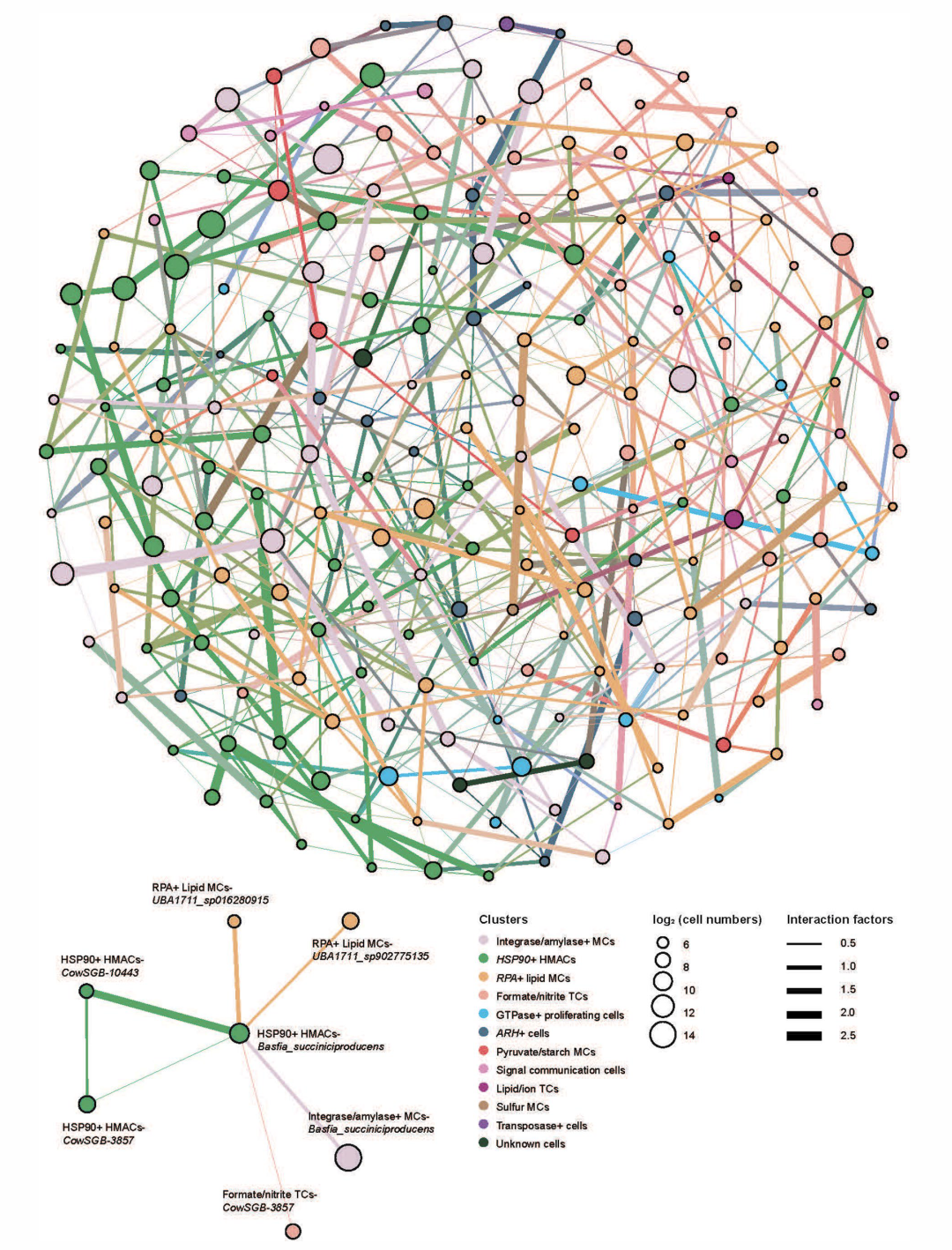
图5. 细菌单元之间的互作网络分析
04、瘤胃微生物组的单细菌代谢研究
HSP90+ HMACs功能簇是瘤胃中具有高代谢活性的细菌,该功能簇比其他细菌具有更高的总代谢FGPs和碳水化合物代谢FGPs(图5A)。对HMACs进一步降维聚类,研究人员获得10个亚功能簇(图6B、C)。为了进一步探索在瘤胃微生物发酵过程中,HMACs对碳水化合物代谢的积极作用,研究团队以关键中间代谢产物丙酮酸盐作为核心,完善了从纤维底物到挥发性脂肪酸(VFAs)的经典碳水化合物代谢途径。在10个亚功能簇中,我们观察到碳水化合物代谢途径的明显异质性,以及HMACs在纤维底物到VFAs的经典碳水化合物代谢途径中发挥的重要作用(图6D)。亚功能簇之间的异质性反映了功能簇的可以进一步细分的特性,表明通过重聚类分析可以逐步提高细菌类型的分辨率。丙酸盐代谢瘤胃中纤维降解的重要途径,是作物副产品利用的限制途径。研究者选择细胞数最多的4个亚功能簇,计算它们在从果胶到丙酸盐的4个连续步骤中的FGPs,显示这4个亚功能簇的代谢存在明显的异质性(图6E)。
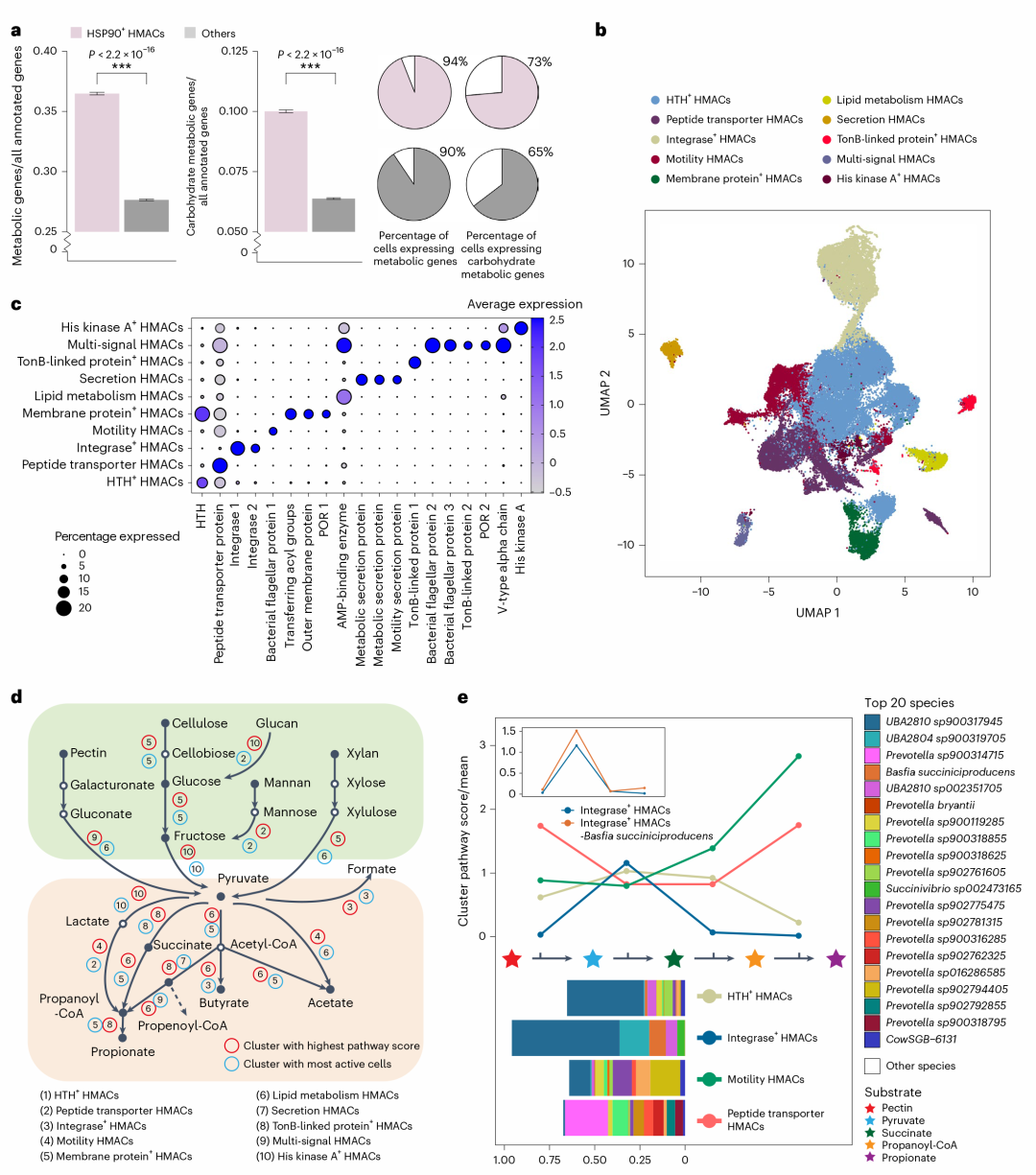
图6. 瘤胃经典碳水化合物途径的代谢分析
05、B.succiniciproducens的功能轨迹
B.succiniciproducens是以葡萄糖为底物生产琥珀酸的主要细菌,在本研究的所有样本中均检测到B.succiniciproducens。在此研究之前,尽管B.succiniciproducens的重要功能已被公认,但其在瘤胃微生物群落中的代谢模式尚不明确。本研究中,B.succiniciproducens(5,591个)降维聚类可获得8个亚群(图7A)。由于微生物也有基于基因表达的生物过程的连续进展,研究者进行了拟时间分析,发现细菌亚群明确分布在B.succiniciproducens的3个不同轨迹上(图7B)。研究人员提取了每个轨迹上细胞数量最多的亚群进行进一步分析:Transposase+ Formate/nitrite TCs(轨迹1;131个细菌)、Integrase+ cells(轨迹2;206个细胞)和Multi signal cells(初始轨迹;1,316个细胞)。基于瘤胃微生物组的生物学过程和从底物到最终产物的动力学,预测B.succiniciproducens的代谢轨迹从初始轨迹(Multi signal cells)移动到轨迹1(Transposase+ Formate/nitrite TCs)和轨迹2(Integrase+ cells)。进一步的拟时序差异基因分析证实了这种转化(图7C)。两个转化的亚群在使用丙酮酸作为底物的代谢过程表型上具有明显的异质性:Transposase+ Formate/nitrite TCs在“丙酮酸到乙酰辅酶a”的代谢过程中明显更活跃,而Integrase+ cells在代谢过程中“丙酮酸到琥珀酸”中显著更活跃(图7B和7E)。
丙酮酸是葡萄糖代谢的关键中间体,与糖、脂肪和氨基酸相互转化。其代谢与瘤胃微生物的氢代谢密切相关,对微生物发酵过程和产甲烷菌的影响重大。丙酮酸代谢可降低琥珀酸盐的氢气生成,对瘤胃甲烷减排研究有着重要意义。基于基因表达的细菌状态转换在调节微生物细胞代谢表型研究方面具有巨大潜力。
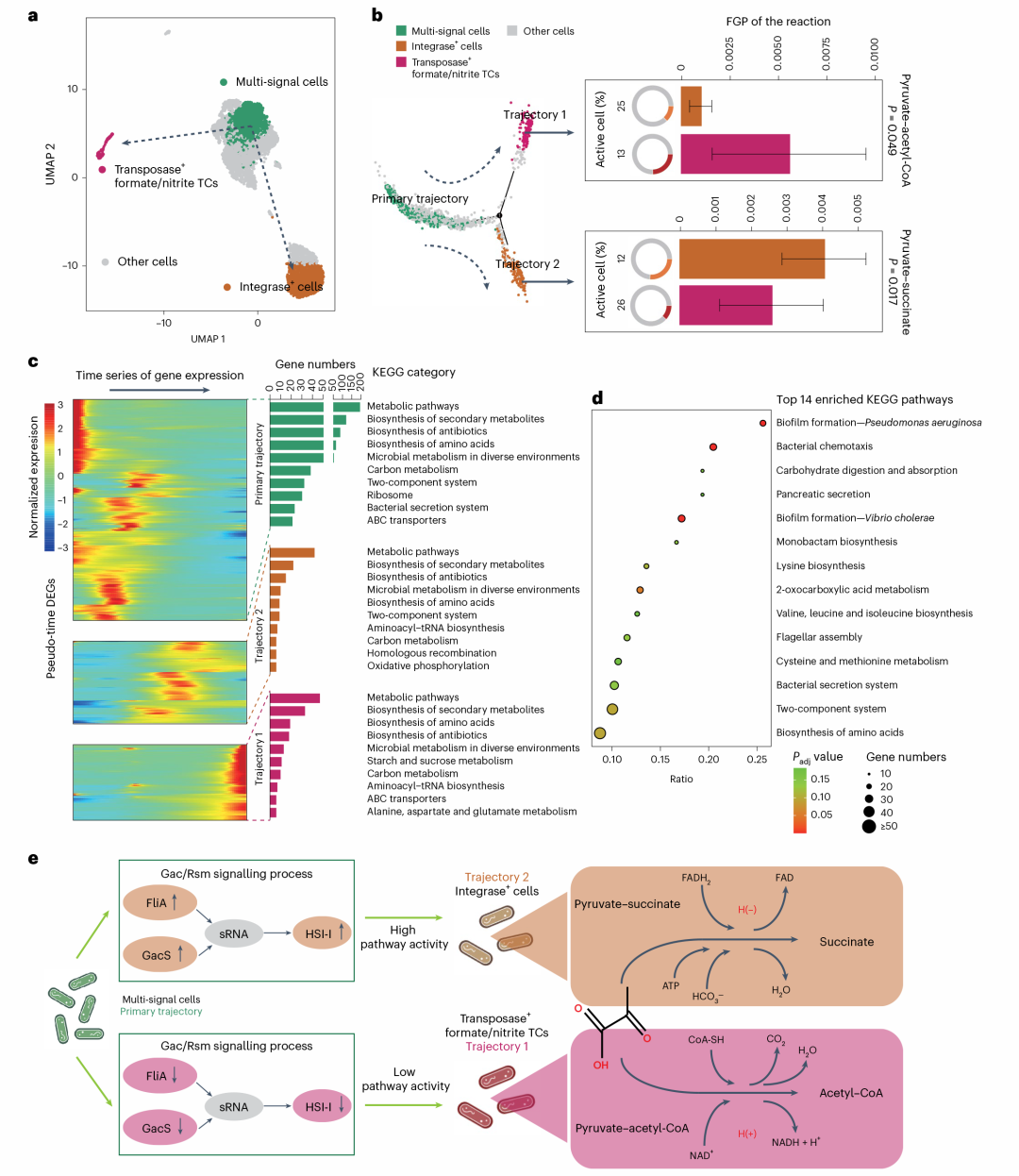
图7. B.succiniciproducens功能异质性和功能轨迹
瘤胃微生物组由多种物种组成,且发酵活跃,是微生物生态系统研究的良好模型。本研究基于VITA单细菌转录组平台及BGMGM,成功构建了瘤胃微生物组的单细菌图谱,涵盖了2,534种微生物的174,531个高质量单细菌转录组数据,成功鉴定了由多种微生物组成的功能簇,研究了功能簇的异质性及细菌之间的互作网络。此外,基于单细菌图谱,准确地表征微生物在碳水化合物代谢过程中的核心功能和关键动态特征,从而更新了对瘤胃中经典碳水化合物代谢的认识。
基于M20 Genomics开发的VITA单细菌转录组测序平台进行微生物组的单细菌转录组研究,在分辨率、准确性方面取得了显著进步。这为研究复杂微生物群落提供了一种适用的单细胞层面的研究方法,能够更全面、深入地解析微生物组的功能异质性和调控机制,在健康、农业、环境保护和生态可持续发展等领域具有巨大的潜力,能够为解决相关重大问题提供更为精确和有效的策略。
凌恩联手M20 Genomics,推出VITA微生物单细菌转录组产品,为微生物研究工作者提供最前沿的单细胞研究技术,充分满足原核生物组学研究实际所需,推动微生物单细胞转录组研究蓬勃发展。
参考文献:
[1] Xu Z, Wang Y, Sheng K, et,al. Droplet-based high-throughput single microbe RNA sequencing by smRandom-seq. Nat Commun. 2023 Aug 23;14(1):5130.
[2] Meng H, Zhang T, Zhang W, et,al. High-Throughput Host-Microbe Single-Cell RNA Sequencing Reveals Ferroptosis-Associated Heterogeneity during Acinetobacter baumannii Infection. Angew Chem Int Ed Engl. 2024 Feb 28:e202400538.
[3] Shen Y, Qian Q, Ding L, et,al. High-throughput single-microbe RNA sequencing reveals adaptive state heterogeneity and host-phage activity associations in human gut microbiome. Protein Cell. 2024 May 23:pwae027.
[4] Jia, M., Zhu, S., Xue, MY. et al. Single-cell transcriptomics across 2,534 microbial species reveals functional heterogeneity in the rumen microbiome. Nat Microbiol (2024).