1995年VAPINK 等人在统计学习理论的基础上提出了一种模式识别的新方法—支持向量机 。它根据有限的样本信息在模型的复杂性和学习能力之间寻求一种最佳折衷。 以期获得最好的泛化能力.支持向量机的理论基础决定了它最终求得的是全局最优值而不是局部极小值,从而也保证了它对未知样本的良好泛化能力。
在既往文章《R语言手把手教你进行支持向量机分析》文章中,咱们已经介绍了e1071包进行支持向量机分析,今天咱们继续来介绍survivalsvm包进行支持向量机的生存分析,继续使用我们的乳腺癌数据(公众号回复:乳腺癌,可以获得数据)我们先导入数据和R包
library(survivalsvm)
library(survival)
library(foreign)
bc <- read.spss("E:/r/Breast cancer survival agec.sav",
use.value.labels=F, to.data.frame=T)
bc <- na.omit(bc)
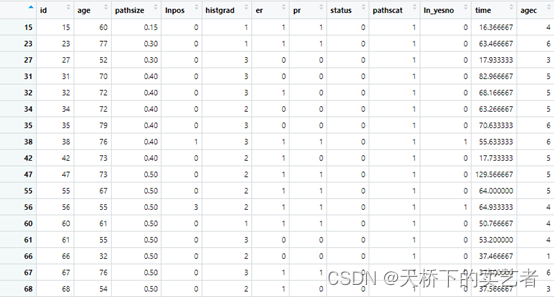
我们先来看看数据:
age表示年龄,pathsize表示病理肿瘤大小(厘米),lnpos表示腋窝淋巴结阳性,histgrad表示病理组织学等级,er表示雌激素受体状态,pr表示孕激素受体状态,status结局事件是否死亡,pathscat表示病理肿瘤大小类别(分组变量),ln_yesno表示是否有淋巴结肿大,time是生存时间,后面的agec是我们自己设定的,不用管它。
先把分类变量转成因子
bc$er<-as.factor(bc$er)
bc$pr<-as.factor(bc$pr)
bc$ln_yesno<-as.factor(bc$ln_yesno)
bc$histgrad<-as.factor(bc$histgrad)
bc$pathscat<-as.factor(bc$pathscat)
把数据分成建模集和验证集
set.seed(12345)
tr1<- sample(nrow(bc),0.7*nrow(bc))##随机无放抽取
bc_train <- bc[tr1,]#70%数据建模集
bc_test<- bc[-tr1,]#30%数验证据集
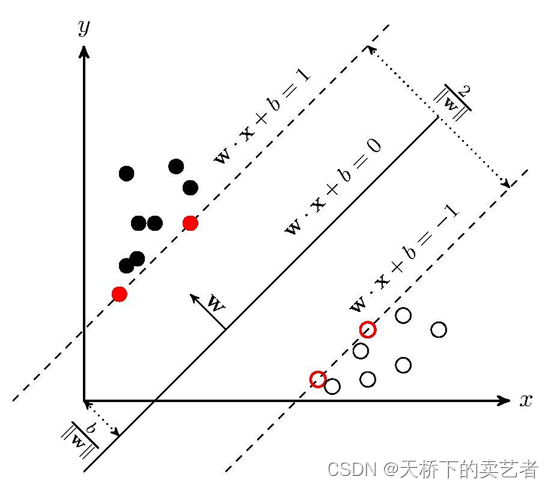
建立生存分析支持向量机的模型,和生存分析的有点类似
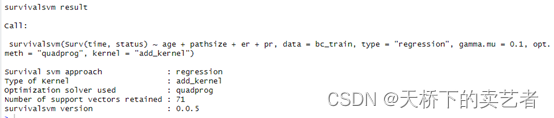
survsvm.pr1 <- survivalsvm(Surv(time, status) ~ age+pathsize+er+pr,data = bc_train,
type = "regression", gamma.mu = 0.25,
opt.meth = "quadprog", kernel = "add_kernel")
type表示需要那种向量模型,默认的是regression,指的是SVCR的回归方法,此外还有’vanbelle1’, ‘vanbelle2’ or ‘hybrid’. gamma.mu是个正则化参数。Kernel表示使用什么内核,这里使用加性内核
接下来生存预测值,并且把预测值提取出来
predsurvsvm<- predict(object = survsvm.pr1,newdata = bc_test)
predtest<-predsurvsvm[["predicted"]]
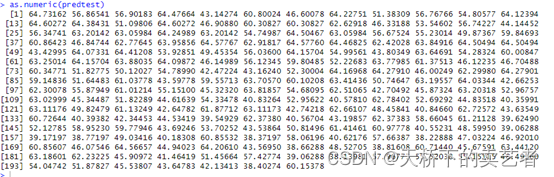
开始我看到结果有点懵,这是预测啥呀,这个不是预测概率,我看到有些文章把这个当作概率来跑,还画了roc和决策曲线什么的,肯定不对的。作者在R包内也表示,这是个预测的值,那就不是概率,主要看是什么的预测值。
捣鼓了一下发现竟然是预测生存时间,我把数据提出来
bc_test$predtest<-as.numeric(predtest)
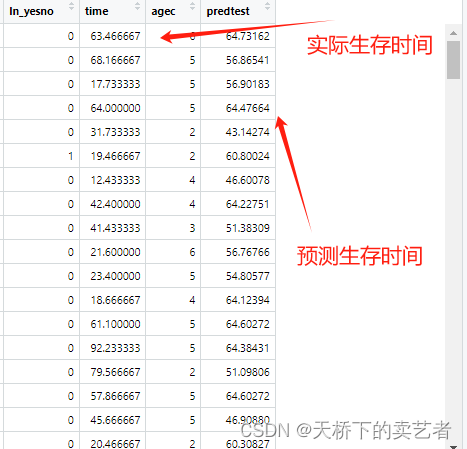
这样一看有些好像预测得还不错。
为什么我能确定这个是预测生存时间,因为作者还写了个mlr3得学习器,我又试着跑了一遍,先把辅助包安装好,需要到github上下载
library("mlr3verse")
library("mlr3proba")
library("survival")
library(mlr3extralearners)
library(survivalsvm)
建立任务
task = as_task_surv(bc,
time = "time",
event = "status",
id = "bc")
进行数据划分,splits里面装着建模和验证集
splits = partition(task, ratio = 0.7)
splits
定义学习器(就是选哪个R包来分析),这里选"surv.svm"
learner_rpart = lrn("surv.svm",
type = "regression",
gamma.mu = 1e-3)
学习器先训练一下(其实就是用R包生成模型)
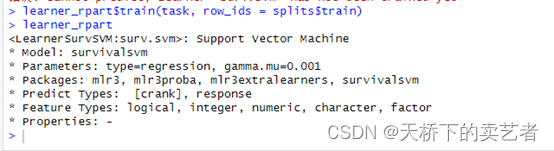
learner_rpart$train(task, row_ids = splits$train)
learner_rpart
生存验证组验证值
predictions = learner_rpart$predict(task, row_ids = splits$test)
也可以用一个连续得写法,都是一样得就是稍微难理解一点
pred = lrn("surv.svm",
type = "regression",
gamma.mu = 1e-3)$train(task, splits[["train"]])$predict(task,splits[["test"]])
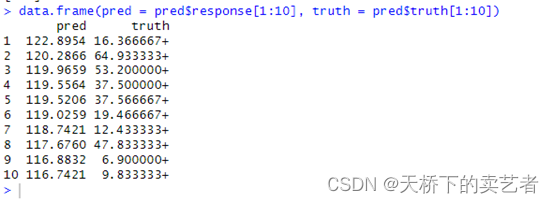
咱们看下前10行预测值和真实值得比较
再看下验证集数据
bc[splits[["test"]],]
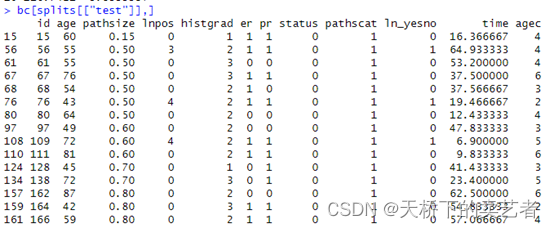
因此预测得就是生存时间啦。
实际运用一下,假设有2个乳腺癌病人数据如下,请问他能活多久

假设这个数据为aa
predict(object = survsvm.pr1,newdata = aa)[["predicted"]]
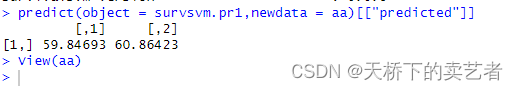
这个包美中不足得是并没有提供交叉验证优化参数得函数,我试着调整了一下gamma.mu这个参数,对预测值影响还是很大得。