🎉 进入生物信息学的世界,与Rosalind一起探索吧!🧬
Rosalind是一个在线平台,专为学习和实践生物信息学而设计。该平台提供了一系列循序渐进的编程挑战,帮助用户从基础到高级掌握生物信息学知识。无论你是初学者还是专业人士,Rosalind都能为你提供适合的学习资源和实践机会。网址:https://rosalind.info
你是否想像专业人士一样分析DNA序列?这里有一个简单的任务来帮助你入门。
📝 任务说明:
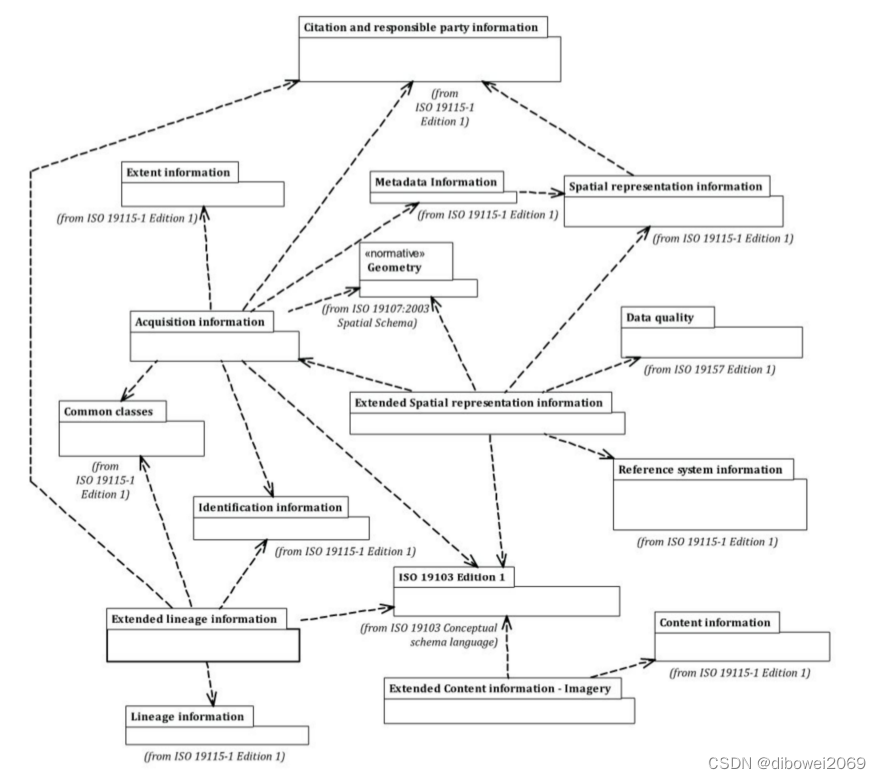
汉明距离:
在信息论中,两个等长字符串之间的汉明距离(英语:Hamming distance)是两个字符串对应位置的不同字符的个数。换句话说,它就是将一个字符串变换成另外一个字符串所需要替换的字符个数。
例如:
- 1011101与1001001之间的汉明距离是2。
- 2143896与2233796之间的汉明距离是3。
- toned与roses之间的汉明距离是3。
def mutation(seq1,seq2):
mutation=0
for base1,base2 in zip(seq1,seq2):
if base1!=base2:
mutation+=1
return mutation
def main():
seq1='GTGCCTATCGGTCAACCCAGGGGGTATACTCACTATCGTTTTTAGGGCGAACCCCCTATTACGTAAACTTCGACGGTCGAGCTACTACTACTT'
seq2='GTGCCTATCGGTCAACCCAGGGGGTATACTCACTATCGAATTTAGGGCGAACCCCCTATTACGTAAACTTCGACGGTCGAGCTACTACTACTT'
print(mutation(seq1,seq2))
if __name__=='__main__':
main()
纸上得来终觉浅,绝知此事要躬行。
公众号:BIoYfan,之后会坚持同步更新生信方面内容
与君共勉💪









![已解决Error || RuntimeError: size mismatch, m1: [32 x 100], m2: [500 x 10]](https://img-blog.csdnimg.cn/direct/aa86eef0d81e4f279688a772e92ea3bc.webp#pic_center)