一. 单倍型网络
单倍型网络图通常是指在遗传学和进化生物学中使用的一种图形表示法,用于描述不同个体或群体之间的遗传关系。这种图形通常用于研究基因或DNA序列在不同个体之间的变化和传递。在单倍型网络图中,节点表示不同的单倍型,而边表示这些单倍型之间的关系。单倍型是指一组基因座上的等位基因排列,通常由父母传递给子代。通过分析这些单倍型之间的关系,研究人员可以了解基因在群体中的传播方式,推断进化过程,以及研究种群的遗传结构等。这种图形表示法在基因组学研究中经常用于研究人类群体、动植物种群等,以揭示基因变异和演化的模式
基因单倍型网络(Genetic Haplotype Network)是一种用于分析基因组数据的工具,它可以帮助理解不同基因型之间的关系和演化历史。
二. 构建基因单倍型网络通常涉及以下步骤:
-
收集基因数据:首先,需要收集感兴趣的基因的数据。这些数据可以来自于实验室的基因测序实验、公共数据库(如dbSNP、1000 Genomes Project等)或者其他的来源。
-
数据预处理:对收集到的基因数据进行预处理是非常重要的。这可能包括去除低质量的数据点、处理缺失值、进行基因型的编码等。确保数据的质量和一致性对于构建准确的单倍型网络至关重要。
-
计算单倍型:单倍型是指在一对等位基因上的一组遗传标记的特定组合。通常,基因数据会包含多个单核苷酸多态性位点(SNP),而每个SNP有两种等位基因(如A和T)。通过分析这些SNP的组合模式,可以确定不同的单倍型。
-
构建网络:一旦获得了单倍型数据,就可以开始构建单倍型网络了。在网络中,每个节点代表一个单倍型,而边则表示单倍型之间的关系。通常,基因单倍型网络是基于遗传距离或相似性来构建的,这可以使用各种算法和技术来完成,如最小生成树算法、中位数网络算法等。
-
可视化和分析:构建网络后,通常需要对其进行可视化和分析,以便更好地理解基因型之间的关系。常见的可视化工具包括Cytoscape、Gephi等。此外,还可以使用网络分析方法来识别网络中的关键节点、社区结构等,以揭示基因型之间的演化模式和功能关联。
总的来说,构建基因单倍型网络需要对基因数据进行仔细处理和分析,并利用适当的算法和工具来揭示基因型之间的关系和演化历史。
1.基因型和单倍型:
在基因组中,基因型是指一个个体在一对基因座上的基因的组合。单倍型是指在一个特定基因座上的一组等位基因的排列方式。每个个体有两个基因,分别来自母亲和父亲。一个基因座上的两个等位基因组成一个单倍型
2.举例

在这个图中,每个节点代表一个单倍型,用A和B表示基因座上的两个等位基因。例如,节点"A1B1"表示在这个基因座上,个体拥有A1和B1两个等位基因。图中的连接表示遗传关系。
现在,我们来进行一些简单的分析:
- 亲缘关系: 通过图中的连接,我们可以看到哪些单倍型是由共同的祖先传递而来的。例如,A1B1和A1B2之间有连接,说明它们可能有共同的父母。
- 基因流动: 通过观察图中的连接模式,我们可以了解基因在群体中的传递方式。如果有很多连接穿越整个图,说明基因在群体中的流动较为频繁。
- 群体结构: 如果图中存在明显的分支结构,表示群体中可能存在亚群体,基因在这些亚群体之间的交流较少。

三.用PopART构建单体型网络和单体型地图
软件下载
直接官网下载就好,PopART。

PopART,该软件支持Windows,Mac,Linux系统,而且用起来也非常方便,支持多种常用的Network构建方法,关键是该软件支持地图的形式展示单体型分布。下面简单介绍一下该软件的使用方法。
PopART的输入文件格式为NEXUS,一般主要用到两个部分DATA和TRAITS。
| #NEXUS begin data; dimensions ntax=4 nchar=30; format datatype=dna missing=N gap=-; matrix seq1 CCACCGTTGCTAAAAATTCATGACACAAGG seq2 CCACAGTTTCTAAAAATTCGTGATACAAGG seq3 CCACAGTTGCTACAAATTCATGATACAAGG seq4 CCACAGGTGCTAAAAATTCATGAAACAAGG ; end; BEGIN TRAITS; Dimensions NTRAITS=5; Format labels=yes missing=? separator=Comma; TraitLatitude 53 43.6811 5.4 -25.61 -0; TraitLongitude 16.75 87.3311 26.5 134.355 -76; TraitLabels Europe Asia Africa Australia America; Matrix seq1 10,5,0,6,0 seq2 0,0,5,0,0 seq3 4,0,10,0,0 seq4 0,0,0,4,2 ; END; |
DATA部分主要纪录单体型信息,比较好理解。
TRAITS部分主要纪录单体型来源的群体。如上所示,例子中取了来自5个大洲的样本,一共4种单体型,TRAITS纪录了每种单体型在不同大洲取样的个数,如seq1在Europe有10个,在Asia有5个等等。关键字TraitLatitude和TraitLongitude纪录5个群体取样地点的经纬度,该信息在单体型网络构建中可以不用,当需要用地图展示单体型地理分布时,需要填该信息。
NEXUS文件生成后,打开PopART,通过File -> Open输入NEXUS文件,然后通过菜单栏Network选择单体型网络构建算法,如常用的Median Joining Network。选择Median Joining Network后,会提示填写Epsilon参数,该参数用来控制推断中间单体型的细节程度,该值越大,会展示更多推断的中间单体型,一般选择默认的0就好。填好该参数后,点击OK,就生成了我们需要的单体型网络。然后通过菜单栏Edit下的选项,对图中群体的颜色、字体、图例等进行调整。
数据准备
PopART的输入文件是.nex格式文件,该文件主要包含两个部分内容:DATA和TRAITS,也就是序列信息和分组信息。
DATA准备
当然是准备我们要用来建立单体型网络图的序列文件,画图前要先把所有样本的该序列都做alignment(ClustalW、Muscle等软件)。
拿到序列文件后,就开始构建.nex文件中的DATA部分,把这个复杂的步骤交给DnaSP
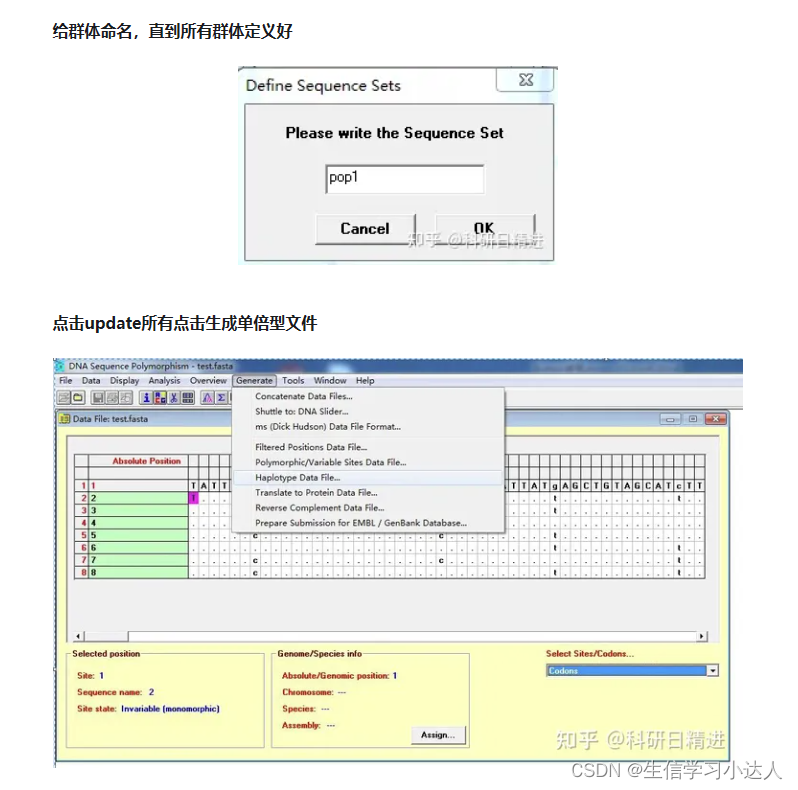
打开DnaSP->载入比对好的序列文件.fas->Generate->Haplotype file->储存为.nex文件
TRAITS准备
这个部分如字面意思,告诉软件数据的分组信息,直接人工添加到上一步生成的.nex文件的最下方(用记事本打开.nex文件编辑)。格式如下图所示:

TRAITS
红框所示部分为样本总分组数;黄框所示部分为样本分组名称;蓝框所示部分为单体型名称和每个分组在该单体型中的样本数量。(注意:分隔符号是Tab键)
画单体型网络图
打开PopART->导入上述.nex文件->选择Network->TCSnetwork
可通过菜单栏各个选项调整群体颜色、字体、比例等。
画单体型地图
选择View->Switch to map view
补充
上面提到,如果我们用来分析的样品不是某基因,而是基因组SNP序列,那么,我们需要找到强连锁区。
#用plink过滤低频位点
plink --vcf xxxx.vcf --maf 0.05 --geno 0.2 --recode vcf-iid -out xxxx-maf0.05 --allow-extra-chr
#用plink筛选连锁区
plink --vcf xxxx-maf0.05.vcf --indep-pairwise 100 50 0.2 -out xxxx-maf0.05-LD --allow-extra-chr --make-bed
#用plink找到强连锁区
plink --noweb --bfile xxxx-maf0.05-LD --blocks no-pheno-req --allow-extra-chr
强连锁区的结果储存在.blocks.det文件中,该文件第一列为染色体/contig名称;第二列为连锁区的开始位置;第三列为连锁区结束位置。找到感兴趣的区域,用beagle软件提出该区域构建DATA文件。
java -Xss5m -Xmx4g -jar YourWayToBeagle/beagle.jar gt=xxxx-maf0.05.vcf out=phased chrom=Chr:start-end
#VCF转换phy
vcf2phylip.py -i phased.vcf.gz
#有了phy文件拿到Win操作系统下,用Bioedit就可以变成fasta格式
上述VCF转换fasta脚本来源:vcf2phylip.py

进化分析Arlequin
Arlequin软件也可以进单倍型分析
参考来源:
PopART构建单倍型网络图 - 知乎 (zhihu.com)
单体型网络的构建——Haplotype Network - 简书 (jianshu.com)
单倍型地理图绘制
PoPART下载
DnaSP下载
beagle下载
进化分析Arlequin全教程,不来看看吗? - 知乎 (zhihu.com)