说在前面
两样本孟德尔随机化应该大伙都了解的不少,不过今天看到一篇,有点“料”的文章,一句话总结:Cytoscape乱拳打死老师傅,通篇除了WGCNA、差异分析是作为常规的转录组分析方法,剩下的几乎都是ClueGO的功劳
今天给大家分享一篇JCR一区,MR的文章:Exploration of the core pathway of inflammatory bowel disease complicated with metabolic fatty liver and two-sample Mendelian randomization study of the causal relationships behind the disease

- 标题:炎症性肠病合并代谢性脂肪肝的核心通路探索及疾病因果关系的二样本孟德尔随机研究
- 期刊名称:Frontiers in Immunology
- 影响因子:7.3
- JCR分区:Q1
- 中科院分区:医学2区
- 小类:免疫学2区
摘要
背景: 炎症性肠病(IBD)经常与复杂的肠外表现相关联。炎症性肠病患者中非酒精性脂肪肝病(NAFLD)的发病率每年都在增加。然而,NAFLD和IBD之间的相互作用机制尚不清楚。因此,本研究旨在探讨IBD和NAFLD的共同遗传特征,并确定潜在的治疗靶点。
材料和方法:从基因表达 Omnibus(GEO)数据库获取了IBD和NAFLD的基因芯片数据集。进行了加权基因共表达网络分析(WGCNA)以识别与IBD和NAFLD相关的数据集中的模块。使用ClueGO对IBD和NAFLD之间共享的基因进行生物学分析。根据Human MicroRNA Disease Database(HMDD),获取了与NAFLD和IBD共同的microRNAs(miRNAs)。使用miRTarbase、miRDB和TargetScan数据库预测miRNAs的潜在靶基因。使用从开放数据库检索的GWAS数据进行了两样本Mendelian随机化(MR)和双向MR,以探讨IL-17与IBD和NAFLD风险之间的因果关系。
结果:
通过WGCNA,确定了感兴趣的基因模块。使用ClueGO进行的GO富集分析表明,趋化因子的异常分泌可能是IBD和NAFLD的共同病理生理特征,并且与IL-17相关的途径可能是IBD和NAFLD中发生的病理变化的共同关键途径。在IBD和NAFLD中确定了核心差异表达基因(DEGs),其中包括COL1A1、LUM、CCL22、CCL2、THBS2、COL1A2、MMP9和CXCL8。使用另一个队列进行验证。最后,通过对miRNAs的分析确定了潜在的治疗靶点。MR的结果表明,虽然IBD和NAFLD之间没有因果关系,但IL-17与IBD和NAFLD之间存在因果关系。
结论:
我们建立了一个共病模型来解释IBD与NAFLD的潜在机制,并确定了由细胞因子IL-17介导的趋化因子相关途径作为IBD与NAFLD中的核心途径,其中miRNA也发挥作用,从而提供了潜在的治疗靶点。
关键词:炎症性肠病,非酒精性脂肪肝病,IL-17,趋化因子,治疗
结果

图1 加权基因共表达网络分析(WGCNA)。
- (A)IBD共表达基因的聚类树。
- (B)NAFLD共表达基因的聚类树。
- (C)IBD中的模块-特征关系。 每个单元格包含相应的相关性和p值。
- (D)NAFLD中的模块-特征关系。 每个单元格包含相应的相关性和p值。 IBD,炎症性肠病;NAFLD,非酒精性脂肪肝病。

图2 ClueGO富集分析。
- (A)由Cytoscape插件ClueGO生成的GO术语组的交互网络,以及(B)每个GO术语组在总体中的比例。 GO,基因本体。 ** p<0.05。

图3 ClueGO富集分析。
- (A)由Cytoscape插件ClueGO生成的KEGG术语组的交互网络,以及(B)每个KEGG术语组在总体中的比例。 KEGG,京都基因与基因组百科全书。 ** p<0.05。

图 4 PPI网络。
- (A-C) 从IBD的黑色模块中获得的1-3簇,(D-F) 从NAFLD患者的蓝色模块中获得的1-3簇。 PPI,蛋白质-蛋白质网络;IBD,炎症性肠病;NAFLD,非酒精性脂肪肝病。

图5 GO富集的生物过程分析。
- (A-C)IBD中三个基因簇的GO生物过程分析,(D-F)NAFLD中三个基因簇的GO生物过程分析。 GO,基因本体;IBD,炎症性肠病;NAFLD,非酒精性脂肪肝病。 ** p<0.05。
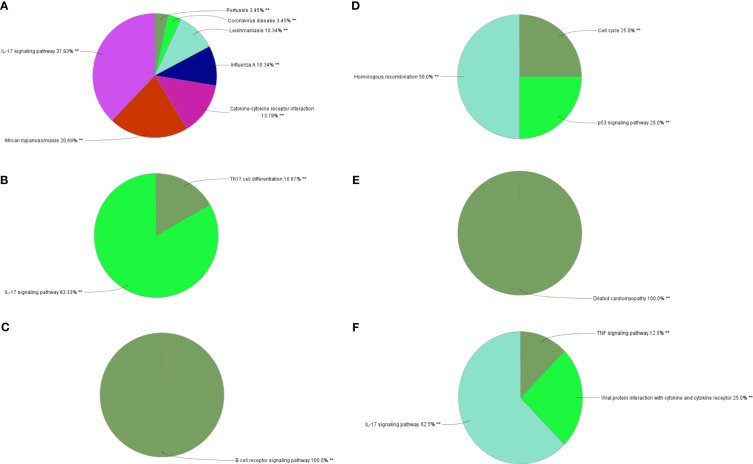
图 6 KEGG富集的生物过程分析。
- (A-C)IBD中三个基因簇的KEGG富集分析。
- (D-F)NAFLD中三个基因簇的KEGG富集分析。 KEGG:京都基因与基因组百科全书;IBD,炎症性肠病;NAFLD,非酒精性脂肪肝病。 ** p<0.05。

图 7 共同差异表达基因(DEGs)的鉴定和ClueGO富集分析。
- (A)IBD和NAFLD中上调和下调基因的维恩图,(B)由Cytoscape插件ClueGO生成的KEGG术语的交互网络及其在总体中的比例。 KEGG:京都基因与基因组百科全书;IBD,炎症性肠病;NAFLD,非酒精性脂肪肝病。 ** p<0.05。
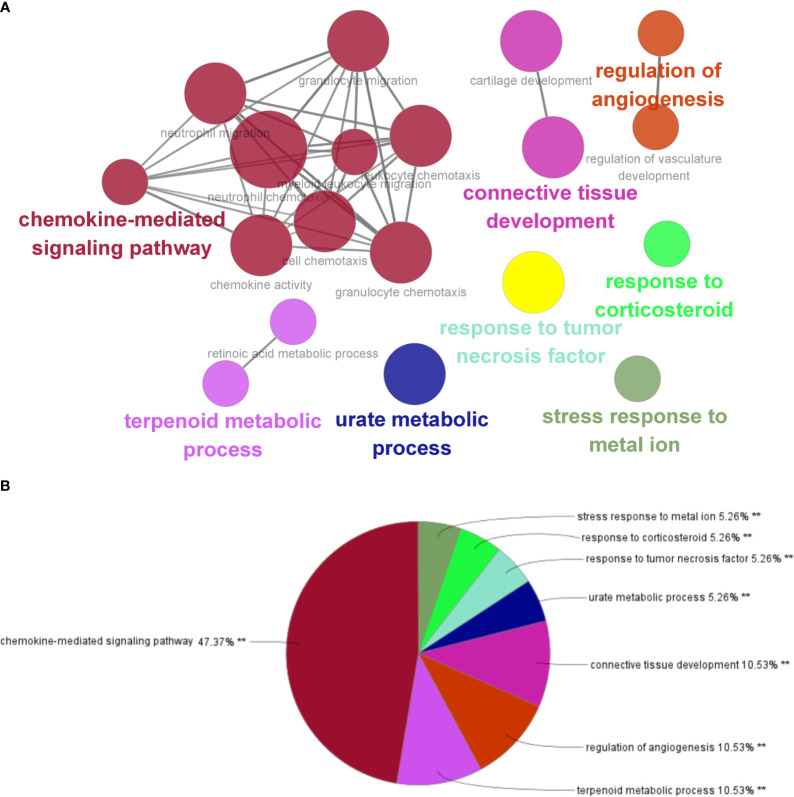
图 8 由Cytoscape插件ClueGO生成的KEGG术语的交互网络及其在总体中的比例。 GO,基因本体;IBD,炎症性肠病;NAFLD,非酒精性脂肪肝病。 ** p<0.05。
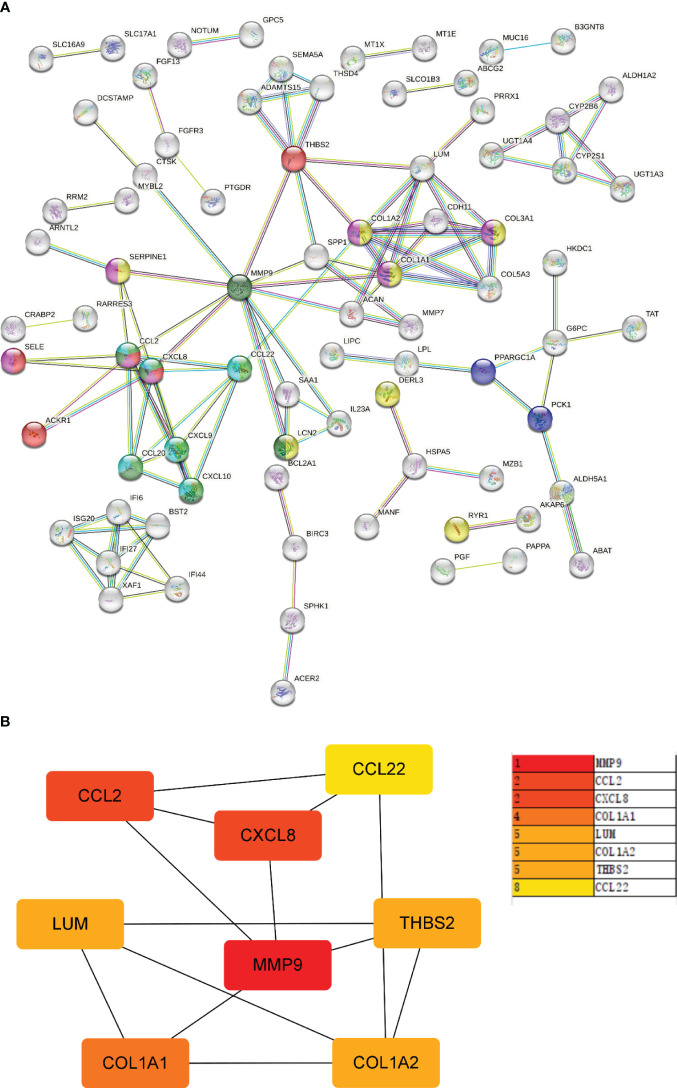
图 9 PPI网络和核心基因网络。
- (A) PPI网络;
- (B) 核心基因网络和排名。

图 10 GO和KEGG富集分析。
- (A) GO富集分析(生物过程),
- (B) KEGG富集分析。

图 11 在IBD进展的不同阶段中,每个组的基因表达。
- (A) GSE89632基因表达,
- (B) GSE130970基因表达,
- © GSE179285基因表达。 ***:p< 0.001, **:p<0.01, *:p<0.05, ns:p>0.05。
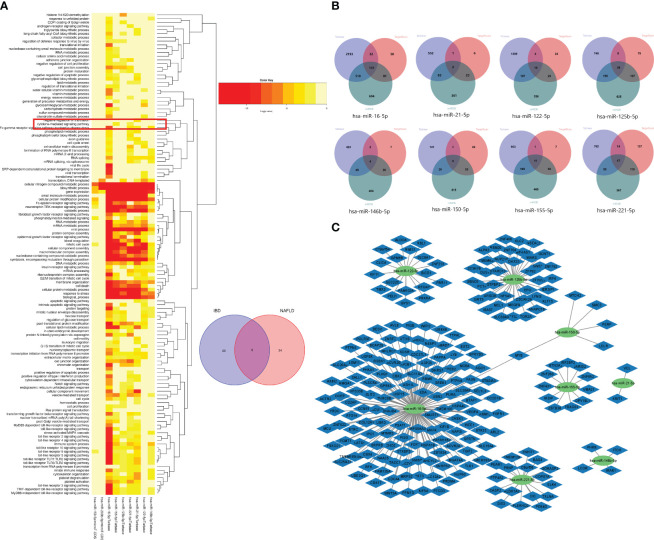
图 12 九个共同miRNA的GO功能富集分析。
- (A) 显示了趋化因子介导的信号转导途径。
- (B) 从miRTarbase、miRDB和TargetScan数据库预测的miRNA靶基因的维恩图。
- © MiRNAs和微RNA的miRNA-mRNA网络。

图 13 与IBD和NAFLD相关的关联。 NAFLD与IL-17F相关。 IBD与IL-17A和IL-17B相关,并且IL-17A和IL-17F之间存在双向相关关系。 IBD和NAFLD之间没有因果关系。
小结
- MR数据:
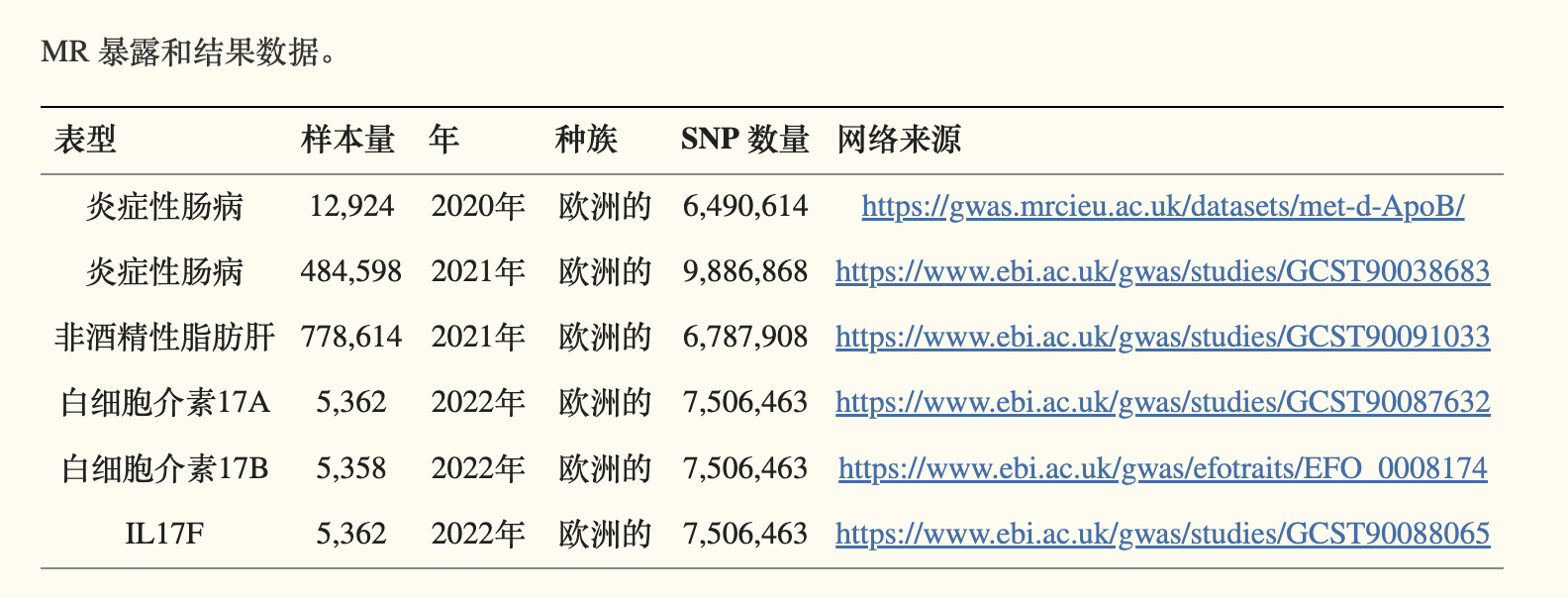
- 主要数据及方法:
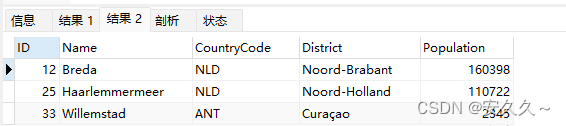
| Types | Notes |
|---|---|
| 分析数据 | NAFLD训练集:GSE126848;NAFLD验证集:GSE89632、GSE130970;IBD训练集:GSE193677、IBD验证集:GSE89632、GSE130970 |
| 分析方法 | WGCNA分析;Cytoscape+ClueGO做富集分析;limma差异分析;MR |