目录
一、PCA主成分分析
二、PCA人脸识别
三、结果
一、PCA主成分分析
PCA(主成分分析)是一种非常常用的数据降维技术。它通过线性变换将原始数据变换到一个新的坐标系统中,使得在这个新坐标系统的第一个坐标轴上的数据方差最大,第二个坐标轴上的数据方差次之,依此类推,以达到降维的目的。下面是更详细的步骤和解释:
中心化数据:首先将数据的每一维度都减去其均值,这样每一维的均值变为0。
计算协方差矩阵:在中心化后的数据基础上,计算协方差矩阵。协方差矩阵反映了数据中各维度间的相关性。
求解特征值和特征向量:计算协方差矩阵的特征值和对应的特征向量。这些特征向量代表了数据中的主要变化方向,而特征值的大小表示了各个方向上变化的程度。
选择主成分:一般会根据特征值的大小,从大到小选择前几个特征向量,这些特征向量称为“主成分”。通常选择的主成分数量取决于所需保留的原始数据信息量的百分比。
投影到新的空间:将原始数据投影到这些主成分构成的空间中,即可实现数据的降维。
二、PCA人脸识别
import numpy as np
import matplotlib.pyplot as plt
import scipy.io as scio
import matplotlib.image as mpimg # 读入图片
import mpl_toolkits.mplot3d as Axes3D # 用来画三维图
'''============================part1 数据导入及可视化========================='''
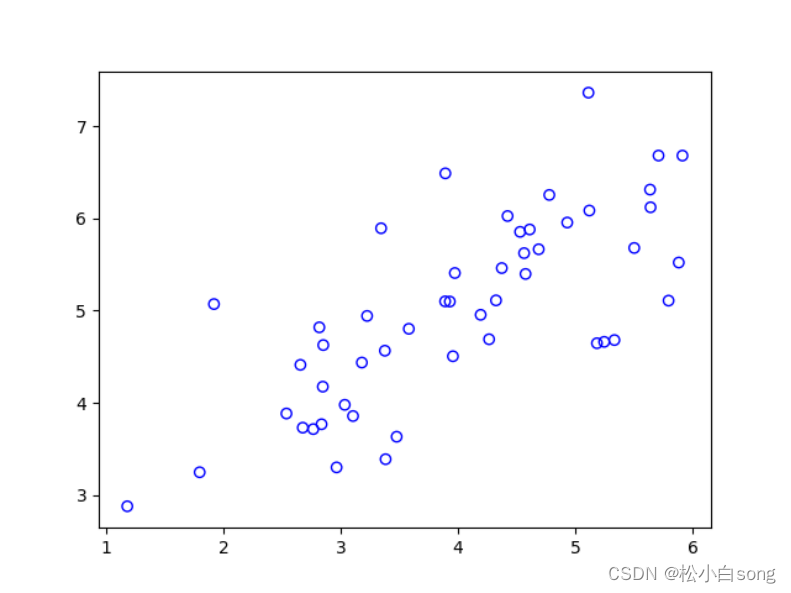
data = scio.loadmat('data_sets/ex7data1.mat')
x = data['X']
plt.scatter(x[:, 0], x[:, 1], marker='o', facecolors='none', edgecolors='b')
'''============================part2 PCA===================================='''
'''数据标准化'''
def featureNormalize(x):
mu = x.mean(axis=0) # 求每列的
sigma = x.std(axis=0, ddof=1) # 无偏的标准差,自由度为n-1
x_norm = (x - mu) / sigma
return x_norm, mu, sigma
x_norm, mu, sigma = featureNormalize(x)
'''得到特征向量和特征值'''
def pca(x):
m, n = x.shape
Sigma = (x.T @ x) / m # 协方差矩阵
U, S, V = np.linalg.svd(Sigma) # SVD奇异值分解
return U, S
U, S = pca(x_norm)
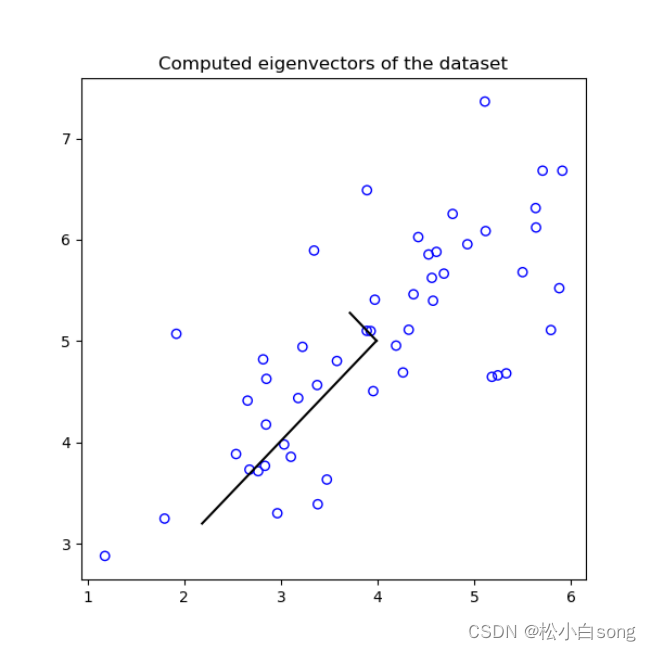
# 画出特征向量
plt.figure(figsize=(6, 6))
plt.scatter(x[:, 0], x[:, 1], marker='o', facecolors='none', edgecolors='b')
plt.plot([mu[0], mu[0] + 1.5 * S[0] * U[0, 0]], [mu[1], mu[1] + 1.5 * S[0] * U[1, 0]], 'k') # 两个点的连线
plt.plot([mu[0], mu[0] + 1.5 * S[1] * U[0, 1]], [mu[1], mu[1] + 1.5 * S[1] * U[1, 1]], 'k') # 这里的1.5和S表示特征向量的长度
plt.title('Computed eigenvectors of the dataset')
'''============================part3 降维========================='''
'''降维,数据投影到特征向量上'''
def projectData(x, U, K):
U_reduce = U[:, 0:K]
return x @ U_reduce
# 降维后的数据,(50, 1)
z = projectData(x_norm, U, 1)
z[0] # 1.48127391
'''重建数据'''
def recoverData(z, U, K):
U_reduce = U[:, 0:K]
return z @ U_reduce.T # 因为U是特征向量矩阵,由标准正交基组成,U.T@U为单位矩阵
# 重建后的数据
x_rec = recoverData(z, U, 1)
x_rec[0, :] # [-1.04741883, -1.04741883]
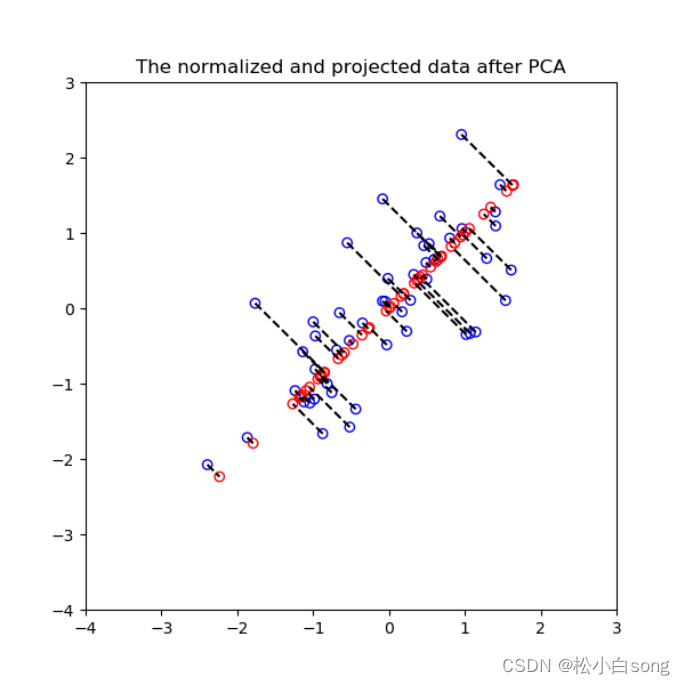
# 可视化投影
plt.figure(figsize=(6, 6))
plt.xlim((-4, 3))
plt.ylim((-4, 3))
plt.scatter(x_norm[:, 0], x_norm[:, 1], marker='o', facecolors='none', edgecolors='b')
plt.scatter(x_rec[:, 0], x_rec[:, 1], marker='o', facecolors='none', edgecolors='r')
for i in range(len(x_norm)):
plt.plot([x_norm[i, 0], x_rec[i, 0]], [x_norm[i, 1], x_rec[i, 1]], 'k--')
plt.title('The normalized and projected data after PCA')
'''保留了多少差异性'''
def retained_variance(S, K):
rv = np.sum(S[:K]) / np.sum(S)
return print('{:.2f}%'.format(rv * 100))
# 看看降维后保留了多少差异性
retained_variance(S, 1) # 86.78%
'''============================part4 人脸数据导入及可视化========================='''
data = scio.loadmat('data_sets/ex7faces.mat')
x = data['X']
'''人脸可视化'''
def displayData(x):
plt.figure()
n = np.round(np.sqrt(x.shape[0])).astype(int)
# 定义n*n的子画布
fig, a = plt.subplots(nrows=n, ncols=n, sharex=True, sharey=True, figsize=(6, 6))
# 在每个子画布中画出一个图像
for row in range(n):
for column in range(n):
a[row, column].imshow(x[n * row + column].reshape(32, 32).T, cmap='gray')
plt.xticks([]) # 去掉坐标轴
plt.yticks([])
# 可视化前100个人脸
displayData(x[0:100, :])
'''============================part5 在人脸数据上实施PCA========================='''
# 数据标准化
x_norm, mu, sigma = featureNormalize(x)
# 特征向量
U, S = pca(x_norm)
# 可视化前36个特征向量
displayData(U[:, 0:36].T)
'''============================part6 人脸数据降维以及可视化========================='''
# 将原始数据降至100维
K = 100
z = projectData(x_norm, U, K)
z.shape # (5000, 100)
# 重建后的数据
x_rec = recoverData(z, U, K) # (5000, 1024)
# 画图对比
displayData(x[0:100, :])
displayData(x_rec[0:100, :])
# 看看降维后保留了多少差异性
retained_variance(S, K) # 93.19%
'''============================part7 PCA用在数据可视化========================='''
# 读取图片
A = mpimg.imread('data_sets/bird_small.png') # 读取图片
'''k-means的代码'''
def findClosestCentroids(x, centroids):
idx = np.zeros(len(x))
for i in range(len(x)):
c = np.sqrt(np.sum(np.square((x[i, :] - centroids)), axis=1)) # 行求和
idx[i] = np.argmin(c) + 1
return idx
def computeCentroids(x, idx, K):
mu = np.zeros((K, x.shape[1]))
for i in range(1, K + 1):
mu[i - 1] = x[idx == i].mean(axis=0) # 列求均值
return mu
def kMeansInitCentroids(x, K):
randidx = np.random.permutation(x) # 随机排列
centroids = randidx[:K, :] # 选前K个
return centroids
# 运行k-means
def runKmeans(x, centroids, max_iters):
for i in range(max_iters):
idx = findClosestCentroids(x, centroids) # 簇分配
centroids = computeCentroids(x, idx, len(centroids)) # 移动聚类中心
return centroids, idx
# k-means部分
X = A.reshape(A.shape[0] * A.shape[1], 3)
K = 16 # 聚类数量
max_iters = 10 # 最大迭代次数
initial_centroids = kMeansInitCentroids(X, K) # 初始化聚类中心
centroids, idx = runKmeans(X, initial_centroids, max_iters) # 得到聚类中心和索引
sel = np.random.randint(X.shape[0], size=1000) # 随机选择1000个样本
cm = plt.cm.get_cmap('Accent') # 设置颜色
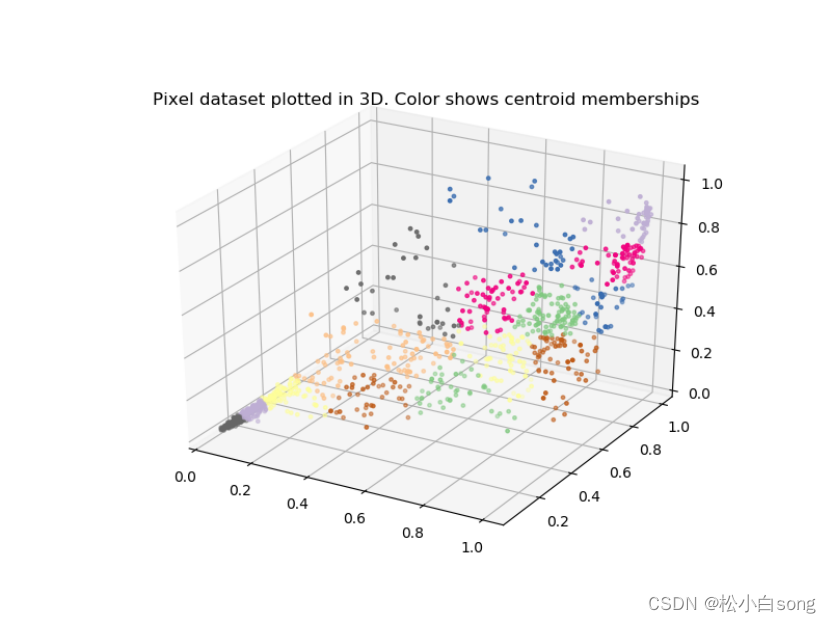
# 画三维图
fig = plt.figure(figsize=(8, 6))
ax = fig.add_subplot(111, projection='3d') # “111”表示“1×1网格,第一子图”
ax.scatter(X[sel, 0], X[sel, 1], X[sel, 2], c=idx[sel], cmap=cm, s=6)
plt.title('Pixel dataset plotted in 3D. Color shows centroid memberships')
# PCA部分
X_norm, mu, sigma = featureNormalize(X) # 标准化
U, S = pca(X_norm) # 特征向量
Z = projectData(X_norm, U, 2) # 投影降维
# 画二维图
plt.figure(figsize=(8, 6))
plt.scatter(Z[sel, 0], Z[sel, 1], c=idx[sel], cmap=cm, s=7)
plt.title('Pixel dataset plotted in 2D, using PCA for dimensionality reduction')
# 看看降维后保留了多少差异性
retained_variance(S, 2) # 99.34%
plt.show() #可视化三、结果