网状meta分析的制作步骤主要包括:
1. 绘制网状证据图
2. 普通Meta分析(两两之间的直接比较)
3. 网状Meta分析(整合直接比较和间接比较的结果,绘制相关图形)
4. 绘制累积概率排序图
5. 三个假设的检验(同质性、相似性和一致性)
6. 偏倚风险评估、文献质量评价、敏感性分析、亚组分析、meta回归等等
步骤1前面已经说过了,步骤2是普通meta分析,也没什么好说的,今天重点介绍步骤3,就是进行网状Meta分析。
今天的代码是接在上一篇文章的后面(网状Meta分析中网络证据图的绘制(R语言版)),用的plotdata1这个连续性变量数据(公众号后台回复“网络meta”获取数据),
首先我们需要准备好数据,数据跟上次演示的数据一样,在公众号后台回复“网络meta”获取数据。
连续性变量:有5种治疗方法,这里用1,2,3,4,5来代替
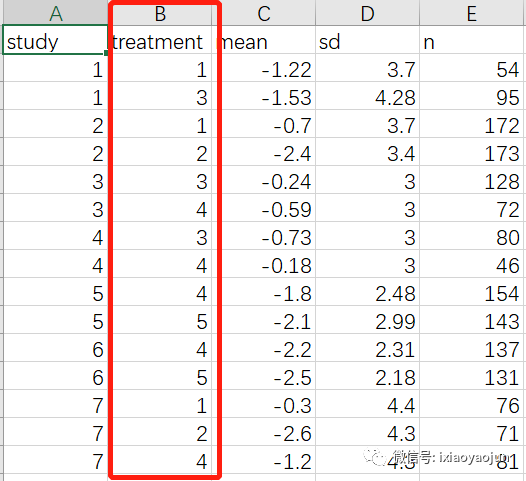
二分类变量:有4种干预措施,这里用1,2,3,4代替。
下面介绍用R绘制网状图的步骤,关于R和Rstudio的安装就不再介绍了,前面的文章都写过。
1. 我们打开Rstudio,第一步仍然是设置工作路径,然后把数据文件都放到路径文件夹下。
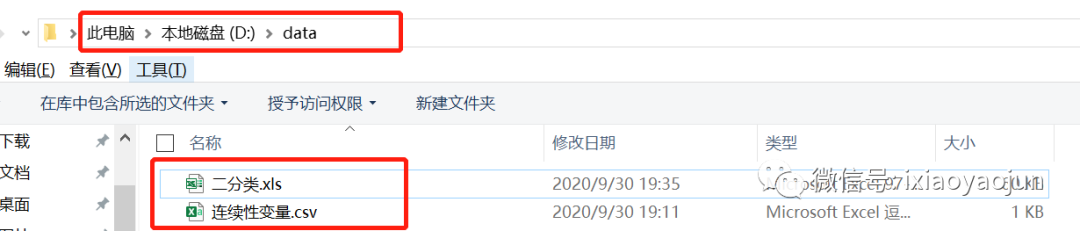
在R语言里设置工作路径为D盘data文件夹。

2. R语言进行网状Meta分析主要就是通过件gemtc 程序包调用对应的 rjags 程序包来执行的,所以第二步需要安装gemtc包,并且调用gemtc和rjags。

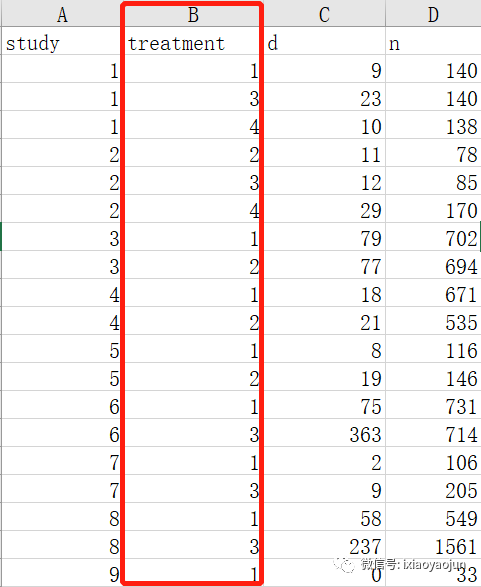
3. 导入数据,分别导入连续性变量和二分类变量,注意数据格式为csv格式。导入后分别赋值为plotdata1(连续性变量)和plotdata2(二分类变量)

导入后在data中可以看到数据。

4.设置标签,连续性变量有5种干预措施,分别定义为A,B,C,D,E五种治疗,赋值为treatments1。二分类变量有4种干预措施,分别定义为F,G,H,I四种治疗,赋值为treatments2。
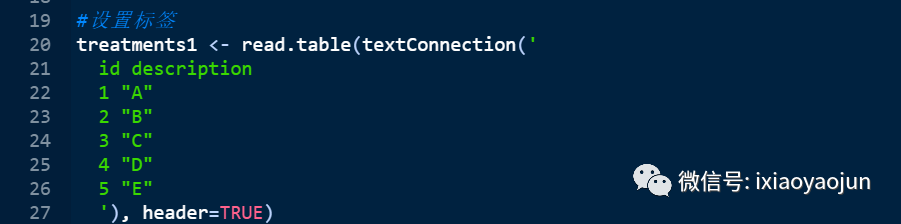
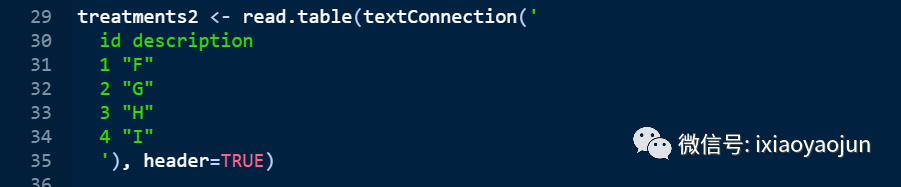
注意跟上篇文章数据的不同之处: 均值、标准差和样本量要设置为mean, std.dev, sampleSize
5.绘制网状图,分别绘制连续性变量和二分类变量的网状图。
连续性变量:注意红色标注的部分,注意和二分类变量区分。每一行都有对应的功能说明,可以自行修改。

执行后即可绘制出下面的网状图
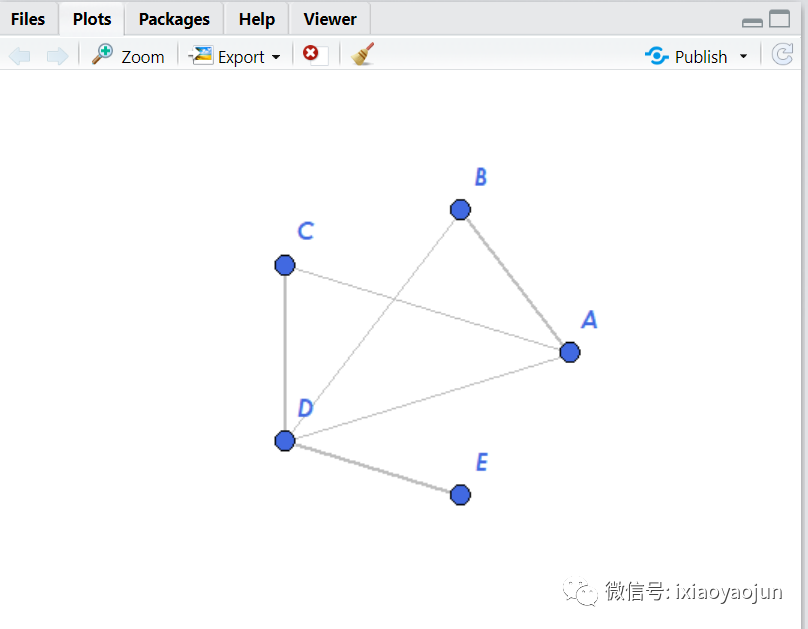
二分类变量:代码跟上面的一样。
执行后即可绘制出下面的网状图
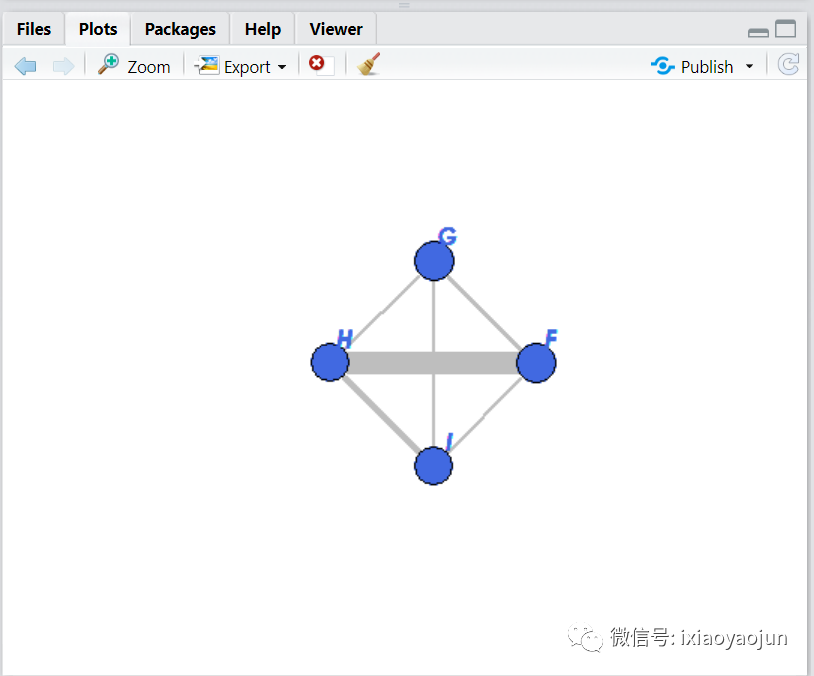
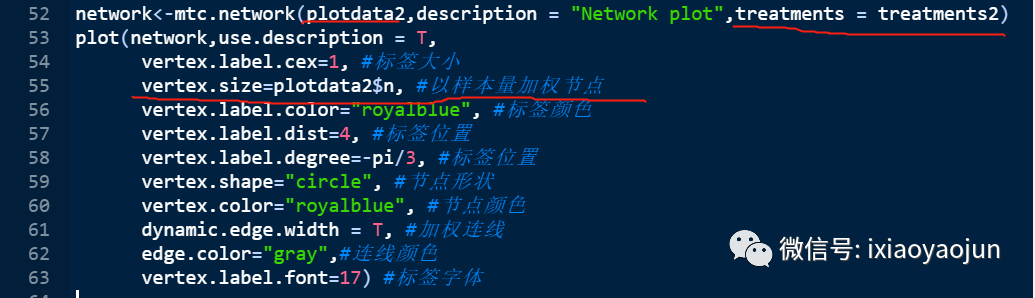
这里要提到的是,如果样本量非常大,建议把上面代码中的样本量加权节点代码(下图)删除掉,不然做出来会非常紧凑。节点加不加权不影响论文的发表。
![]()
通过修改代码里的数值可以进一步修正网状图,也可以导出为PDF后在Adobe illustrator里修改。
在AI软件中可以对线条、节点、标签进行各种修改。
前面的文章里我们构建了network这个数据集,然后制作了网状证据图,今天我们会用到这个数据集。

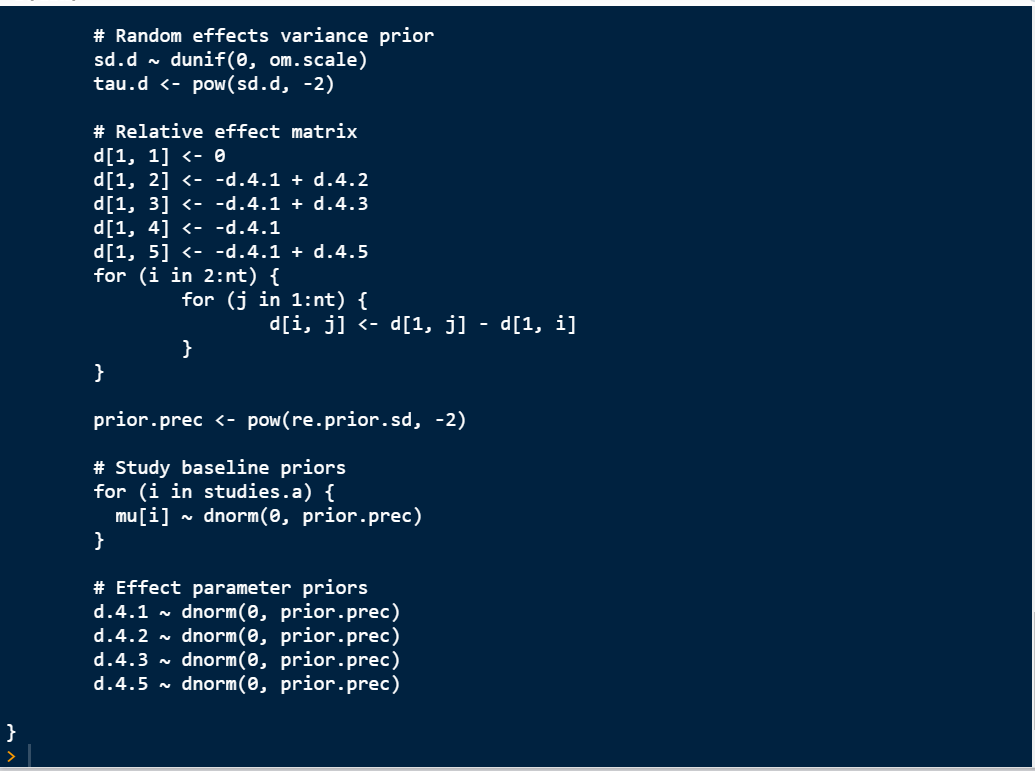
1. 首先利用上面的连续性变量plotdata1的network数据集去构建网状meta分析的随机效应模型。

如果是二分类变量数据需要进行以下修改:link代表链接函数,如果是二分类变量,把identity改成logit,意思就是进行对数转换;likelihood是似然函数,二分类变量改成binom。其他都用上面默认的。
R语言中关于上述代码函数的说明:

2. 使用Markov Chain & Monte Carlo(MCMC)方法进行抽样模拟和计算,参数为n.adapt=20000, n.iter=50000, thin=10。这里可以查看代码和结果。
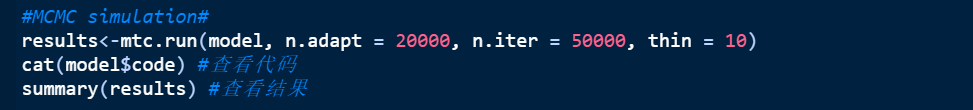
查看代码:这个是调用JAGS计算的贝叶斯代码(电脑要提前安装好JAGS软件,百度一搜一大把),跟WinBUGs这些软件代码一样。我们在文章投稿的时候需要把代码放在附录里,直接在这里copy就可以了。

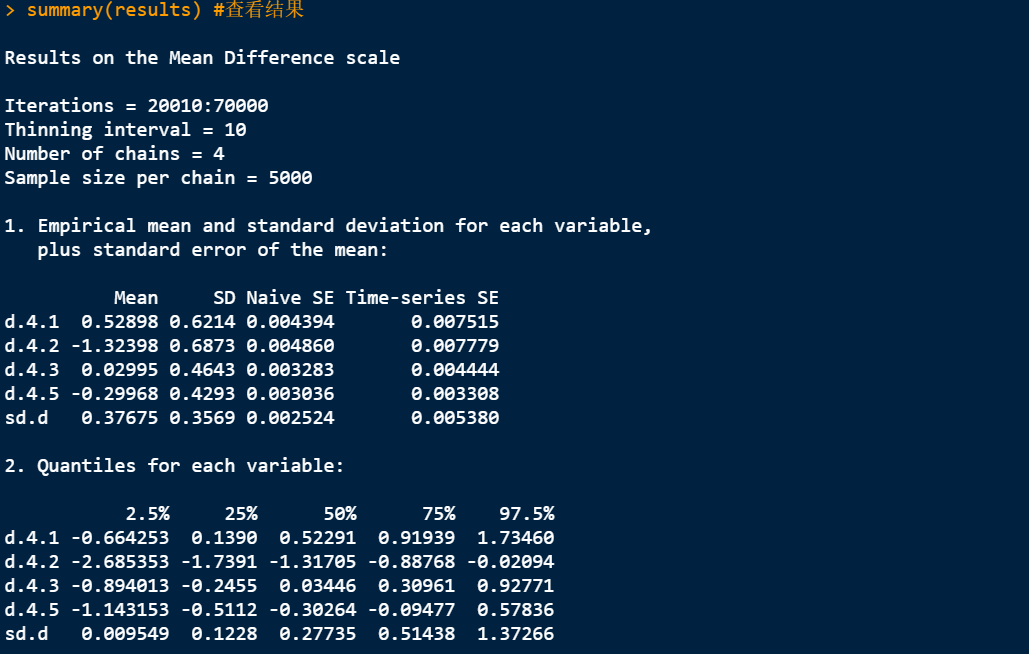
查看结果:里面给了两两比较的结果,红框框出来的结果给出了模型拟合的结果,ratio越接近1表示模型拟合程度越好,同时也给出了模型整体的异质性I2值,我们这个模型异质性等于0,是非常不错的。

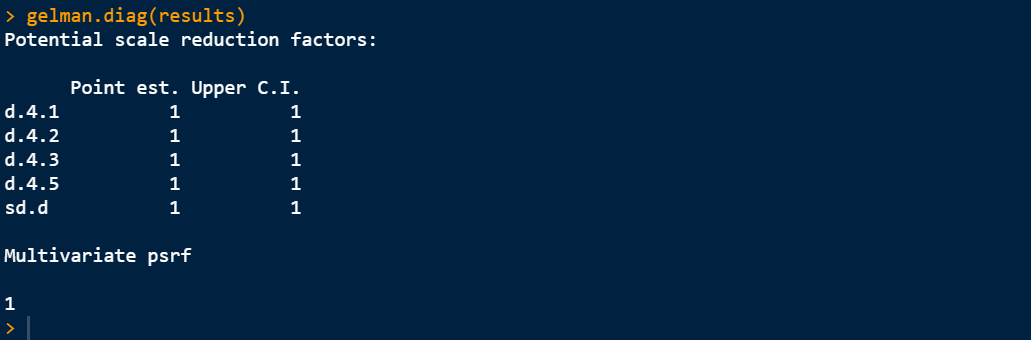
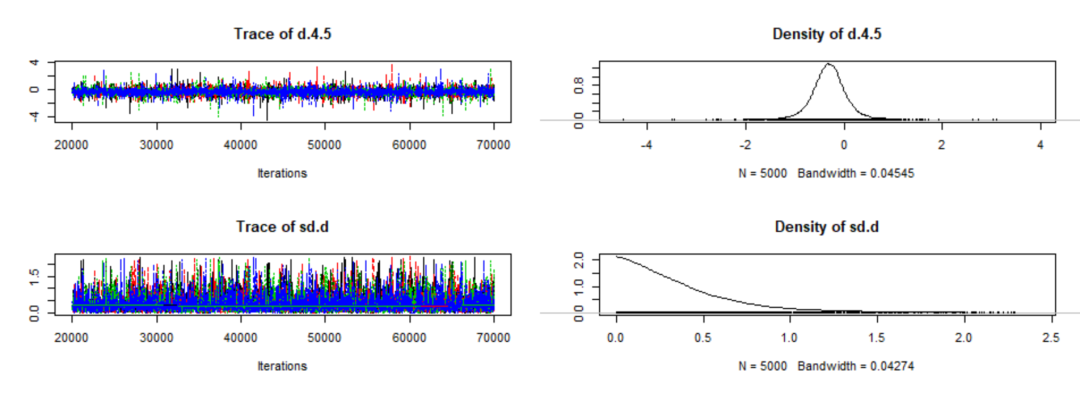
3. 查看收敛诊断结果,并绘制收敛诊断图、轨迹图和密度图

收敛诊断结果:
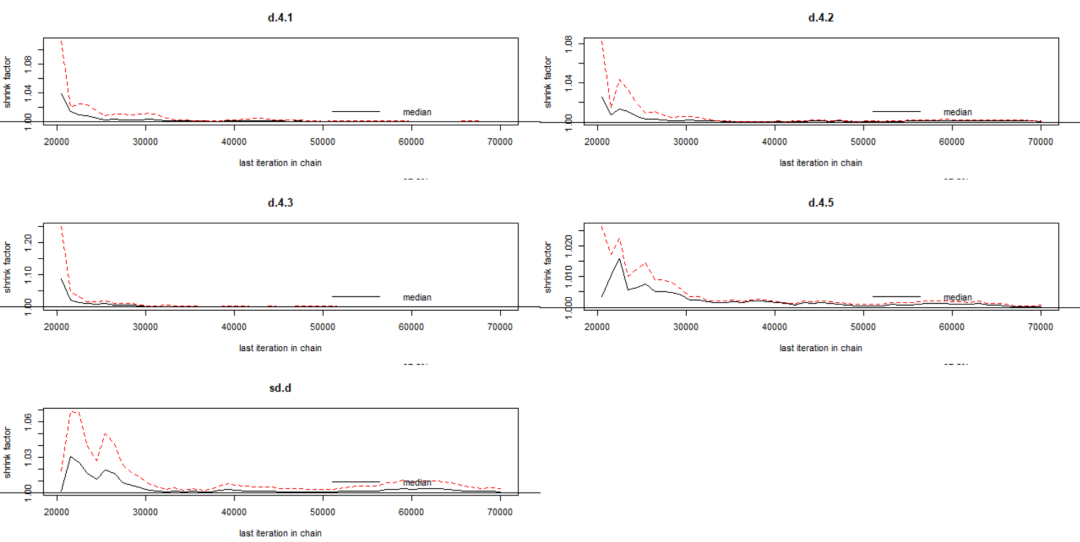
收敛诊断图:
轨迹图和密度图:
4. 绘制网状meta分析的联赛表,并导出为excel。

R中可以查看结果:
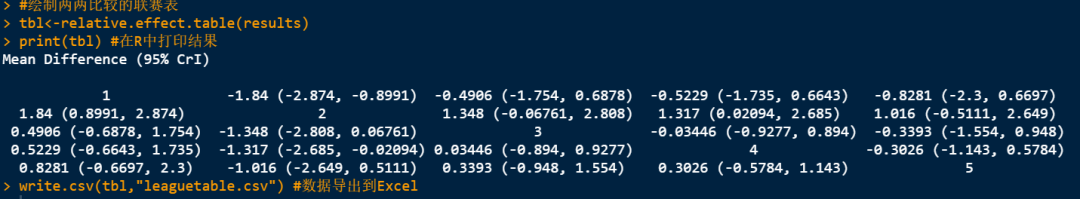
结果导出到你的R语言一开始设置的工作目录,在excel中可以进行进一步编辑
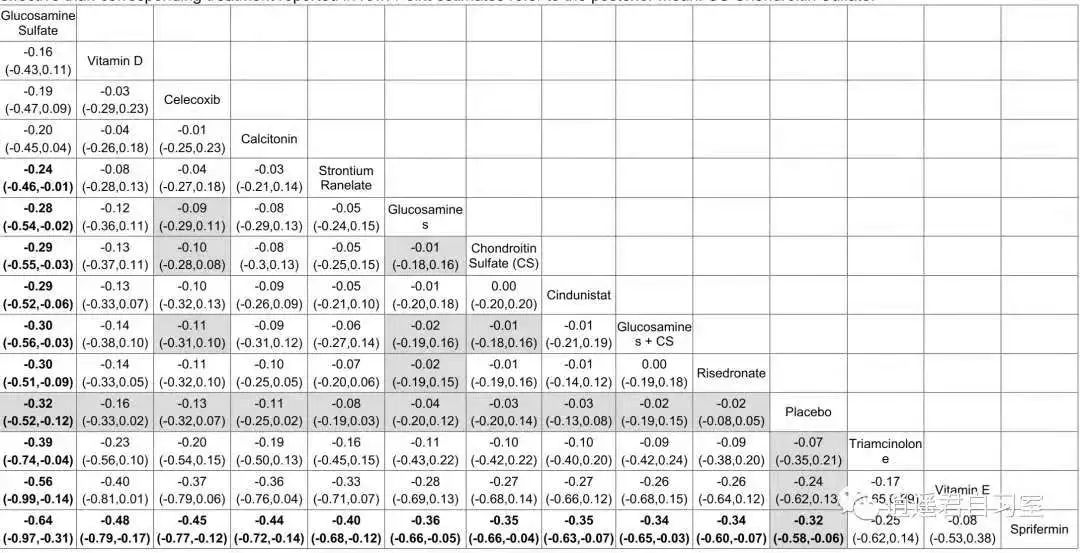
联赛表在前面的文章中有过展示,就是在网状meta分析文章中经常看到的下面的这个图,这个图展示的就是直接比较和间接比较的结果。我们把上面的Excel表格进行加工整理就可以做成下面的图。
5. 绘制网状meta分析的森林图
我们可以用自带的forest代码绘制森林图

森林图展示:
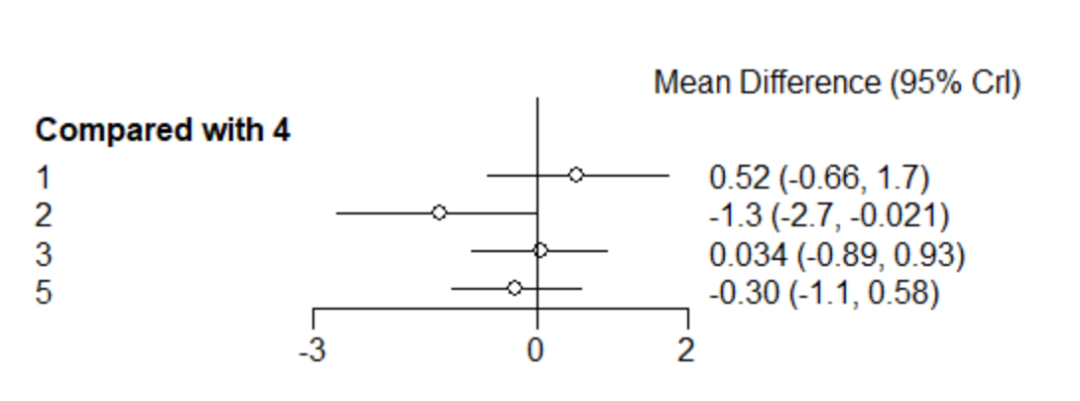
你也可以对数据格式进行重新整理,然后用forestplot包来绘制森林图,这个在前面的文章中有过介绍(用R来做一个临床研究亚组分析的森林图),就不再重复。
累积概率排序图的绘制:
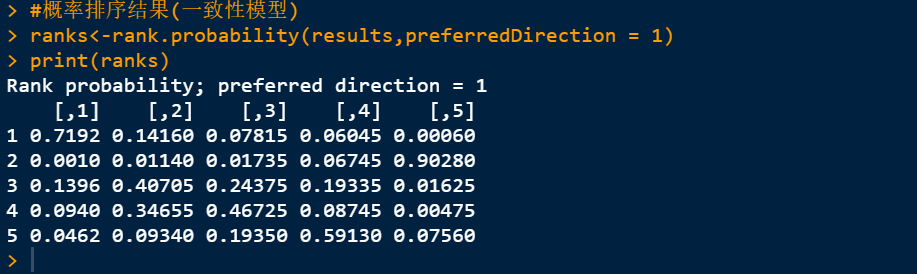
1. 计算排序结果,代码如下,这个主要是用ranks.probability代码来实现,括号里的results是上个帖子里面MCMC抽样预算的结果数据, 如果是比较的治疗方法,preferredDirection设置为1代表数值越大疗效越好,如果设置为-1代表数值越大疗效越差。print代表把排序结果展示出来。write.csv是把结果导出为excel文件。

2.展示的排序结果,从数据来看,如果是治疗方法,那么治疗效果从大到小的顺序依次为1,3,4,5,2,第1种治疗方法效果最好。
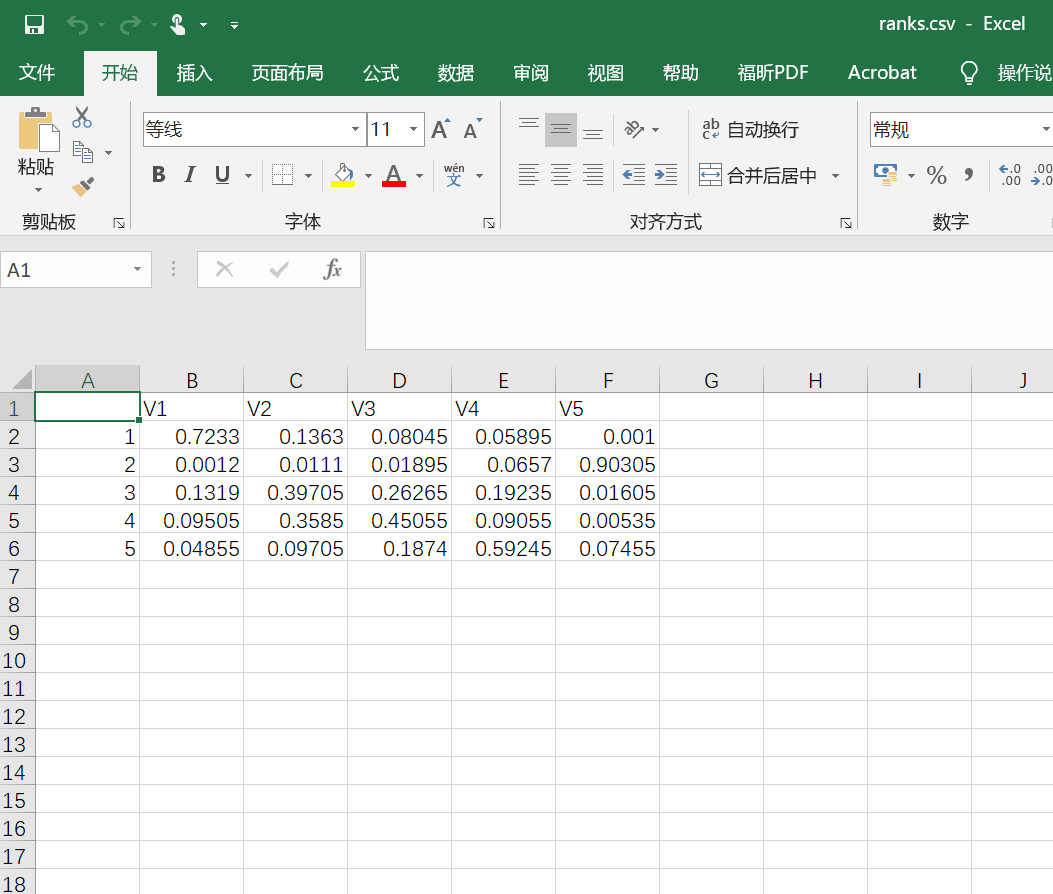
3.导出的excel文件
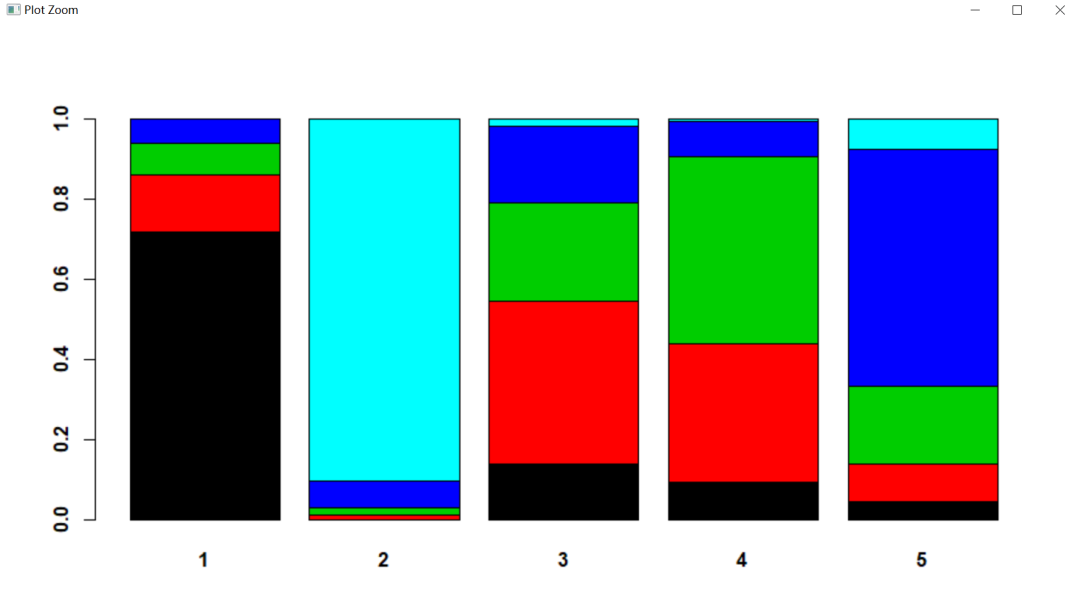
4. 绘制累积概率排序图,如果不需要修改标签和颜色,直接用简单的默认代码,非常简单。

出来的图形:

5.如果需要改标签和图形颜色,可以用下面的代码。

col是颜色函数,这里用的palette是基本颜色(默认是下面几种颜色)。

font.axis是设置字体,可以自己修改数值感受下。
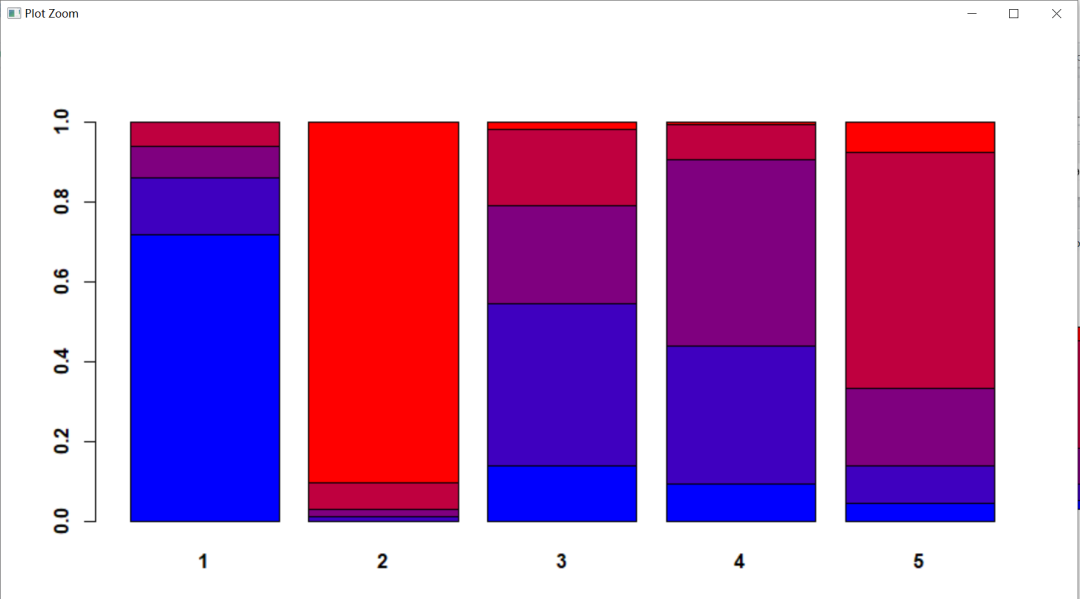
执行代码后是下面的图:
如果你就想用默认的灰白颜色搭配,就把这个代码删除就行了,你想自己修改颜色稍微麻烦点,具体可以参考下面网站的介绍:
网址:https://segmentfault.com/a/1190000006780090
比如我换成colorRampPalette这个函数

出来的就是下面的图:
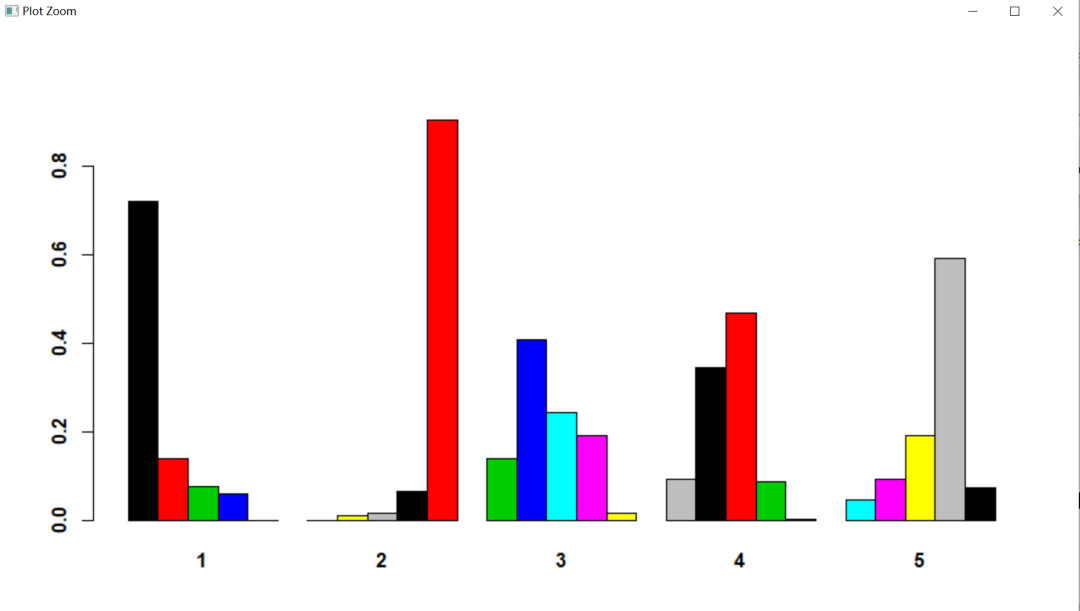
6. 上面的图是累积概率图,如果绘制不累积的概率图,可以用下面的代码。

加一个beside=T就可以分开了,执行后是下面的不累积概率图:
7. 然后我们还可以算sucra值,用的是sucra这个函数,代码如下。

print是展示结果,plot是作图
执行代码后出结果:SUCRA值越接近1,则排名越靠前,若越接近0则排名靠后。
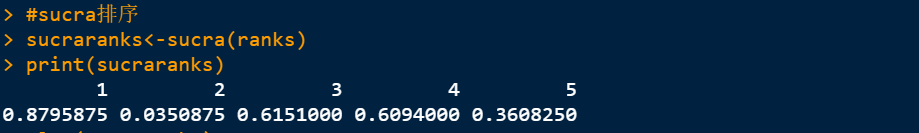
作图:
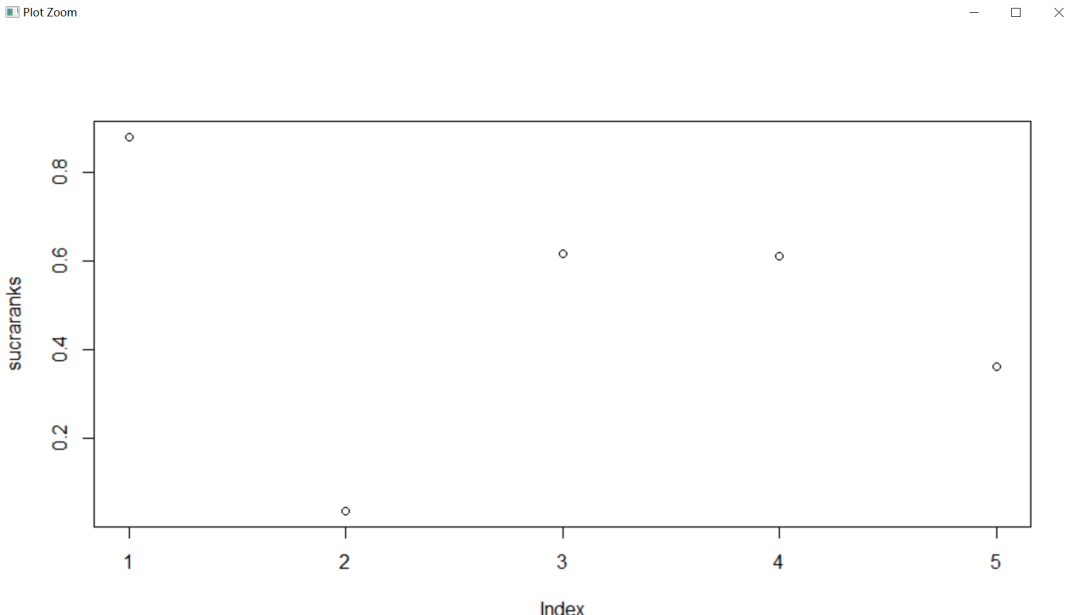
如果想画更好看的图,可以用R语言经典的ggplot2绘图包来绘制概率图,但是要重新整理数据,而且代码也相对比较复杂,这里就不介绍了,上面画的图直接就可以放到文章里,并不影响文章的发表。
5. 三个假设的检验(同质性、相似性和一致性)
6. 偏倚风险评估、文献质量评价、敏感性分析、亚组分析、meta回归等等
1. 首先我们来讲同质性:
网状Meta分析里提到的同质性检验实际上就是普通Meta分析常说的异质性检验。
举个例子,你纳入网状Meta分析有3种治疗措施A、B、C,纳入的文献中有A和B直接比较的文献10篇,B和C直接比较的文献13篇,A和C直接比较的文献12篇。那么你的网状Meta分析中就要包含A和B、B和C、A和C直接比较的普通Meta分析异质性分析结果。
普通Meta分析异质性分析前面的文章中已经专门具体写过,就是从临床异质性、方法学异质性和统计学异质性三个方面去分析,大家可以去看前面的文章(聊聊Meta分析异质性的检验和处理)。最后这个异质性定量分析的结果会在直接比较的森林图里展示出来,主要就是看I2值。
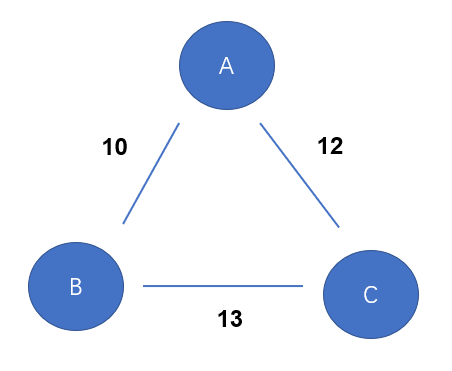
有的同学会提到网状Meta分析不是应该考虑整体同质性吗? 但实际上在做网状Meta分析的时候不需要做这个,你可以去看顶刊的网状Meta分析文章都是做的直接比较的异质性分析。为什么这么做就行了,因为如果直接比较的文献之间都没有同质性,那就谈不上整体的同质性,根本就不适合做网状Meta分析。
最后总结下网状Meta分析的同质性检验实际上就是普通Meta分析的异质性分析,定性用Q检验,定量用I2值,另外还可以比较随机效应模型和固定效应模型的结果(这个在前面提到的一篇JAMA范文里作者用Bland–Altman图示法来展示了随机效应模型和固定效应模型比较的结果),在此基础上还可以做亚组分析和Meta回归来找异质性来源,这个就是最后一步的事情了。



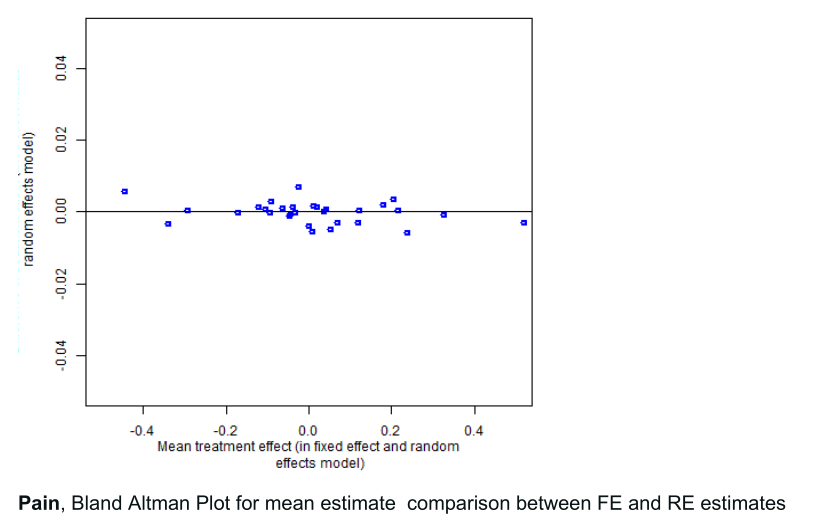
理论上讲,随机效应模型和固定效应模型两种方法一般不会获得完全相同的结果,但是会具有一定趋势的差异,啥意思呢?就是说一种方法的测量结果总是大于(或小于)另一种方法,这种系统误差就是我们常说的“偏倚”。
Bland-Altman法主要是计算出两种方法测量结果的“95%一致性界限(95% limits of agreement, 95% LoA)”,并用图形的方法直观地反映出这个一致性界限——通常以测量结果的差值为纵轴,以测量结果的均数为横轴,绘制散点图,并标注出95%一致性界限。最后得出两种方法是否具有同质性的结论。如果两个测量结果的差异位于95% LoA内,则可以认为这两种方法测量结果具有较好的同质性。
这个Bland-Altman图统计软件都可以做,最常用的傻瓜式软件是Medcalc,点点菜单就完成了,R语言来做当然是没有问题的。因为我们做网状Meta分析全程都是用R,所以这里我把R的代码给到大家,主要用到的就是BlandAltmanLeh这个R包,如果打算做这个图的可以试试。
(1)数据整理成下面的格式
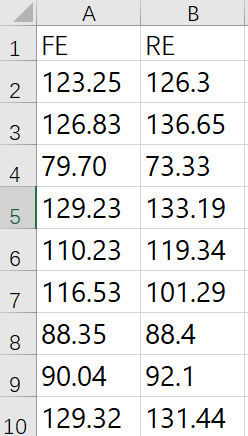
(2)设置工作路径并安装BlandAltmanLeh包
(3)导入数据

(4)作图
#FE指的是固定效应模型的均值,RE指的是随机效应模型的均值,注意顺序,用FE-RE, silente=F,同时给出统计量。默认的LoA计算是用差值mean±1.96SD,并且给出95%置信区间,作出来的图如下,所有的点都95%LoA范围内说明同质性比较好。

2. 我们再来说相似性:
所谓的相似性我们还是用上面的例子来举例,我们要做A和B的间接比较,共同对照组是C,那么相似性指的就是A和C直接比较和B和C直接比较两者之间的相似性。

相似性目前还没有公认的统计学方法,主要还是靠主观判断,可以从临床相似性和方法相似性来分析,这个其实跟普通Meta分析探讨异质性也是一样的思想。
临床相似性从PICO来分析:
-
P主要指的是病人特征,包括性别、年龄、并发症、样本来源、纳入排除标准。
-
I和C主要指干预措施的剂量、干预方法、疗程等等。
-
O指的是结局指标的随访时间、测量方法等等。
方法学相似性主要从S来分析:
也就是研究设计来分析,包括随机的方法、盲法、分配隐藏等等。
既然没有定量的统计学方法,那么如何主观判断是否相似呢?实际上只要会影响到试验效应的关键特征相似即可,一些不会影响到试验效应的特征不相似是没有关系的。
什么是一致性?
网状 Meta 分析中一致性是指直接与间接比较结果的相似度,或者不同路径之间间接比较结果的相似度。
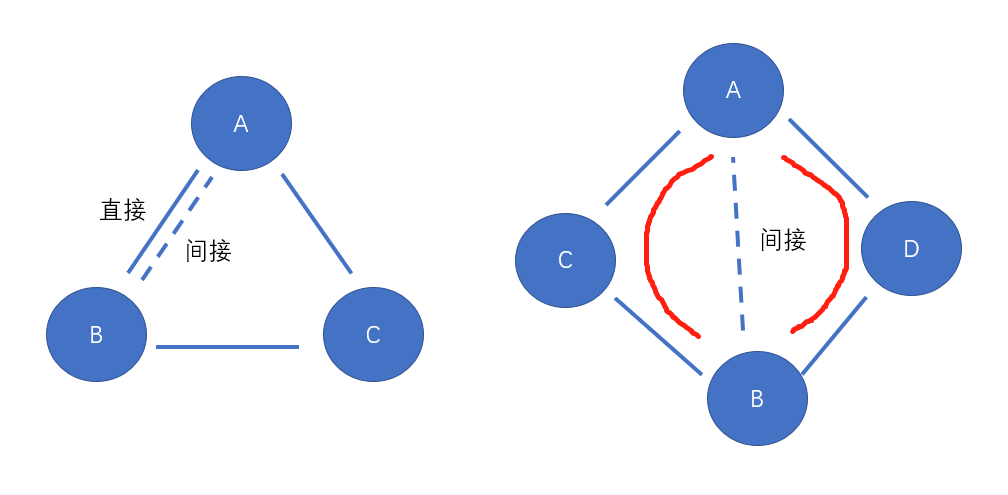
像下面左边的图A和B两种干预措施之间我们要进行比较,既有A和B直接比较的数据合并结果,也有以C为共同对照的A和B间接比较的结果,一致性就是看A和B直接比较和间接比较的结果是不是一致的。右边的图A和B没有直接比较的结果,但是有2种共同对照C或者D,那么一致性就是看以C为共同对照的A和B间接比较和以D为共同对照的A和B间接比较的结果是不是一致的。
关于一致性的检验,就是判断是否存在结果不一致,如果存在直接证据和间接证据的不一致,那么就会影响网状Meta分析结果的真实性。目前我们主要通过定量分析直接比较和间接比较的统计学差异来进行鉴定,常用的检验方法就是节点拆分法(Node-Splitting)(前面的范文里就是用的这个方法: 读一篇JAMA上的范文掌握网状Meta分析的制作)。
节点拆分法就是对网状Meta分析的每一个节点进行分析,比较直接证据和间接证据的统计学差异,如果没有差异说明结果是一致的,可以用一致性模型拟合网状Meta分析,如果有差异说明存在不一致性,那么我们就需要采用不一致性模型进行拟合,我们前面的帖子介绍的网状meta分析都是用的一致性模型拟合的,如果要用不一致性模型拟合,就需要把前面帖子(网状Meta分析进行模型构建及图形绘制)中下面代码的type=“consistency”改成“ume”即可。

在说明书中,可修改的模型如下,除了ume,还有回归模型和节点拆分模型。

扯了这么多,最重要的还是介绍怎么用软件进行一致性检验,我们还是用R语言进行检验,代码如下。
1. 执行节点拆分法。

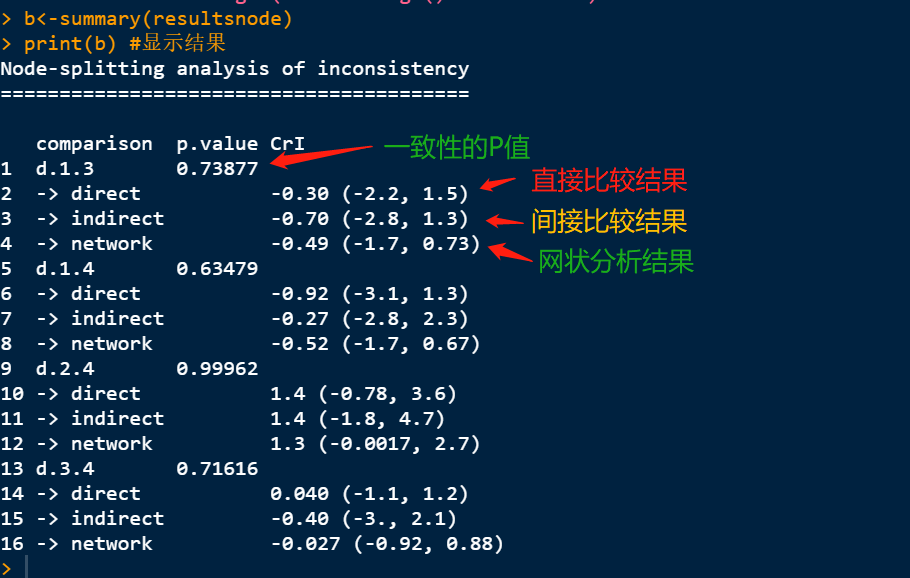
2.展示结果。

结果展示:P值大于0.05表明2个干预措施的直接比较和间接比较是一致的。d.1.3 代表的是1号干预措施和3号干预措施之间的比较,以下类推。
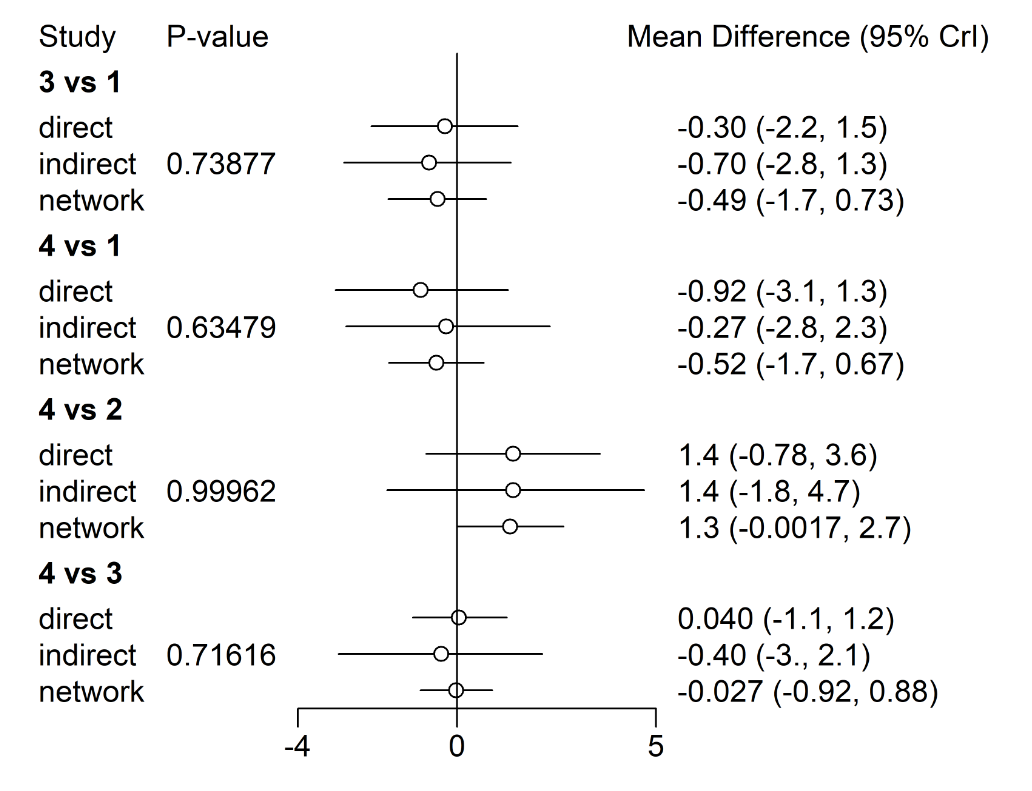
3.绘制森林图并导出

在完成一致性检验之后,如果发现存在不一致性,那么就需要找产生不一致性的原因,比如临床特征和方法学是否有差异,是否存在异质性,这里就顺便讲下如何进行异质性检验。
1.异质性检验的代码如下:
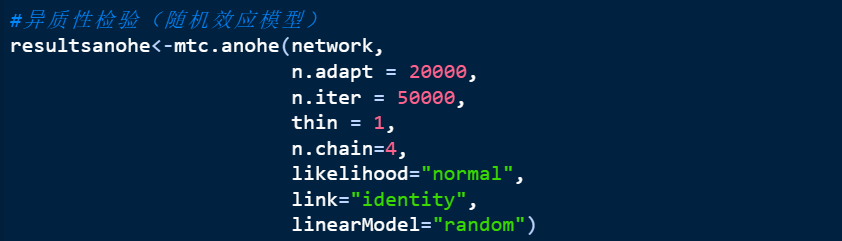
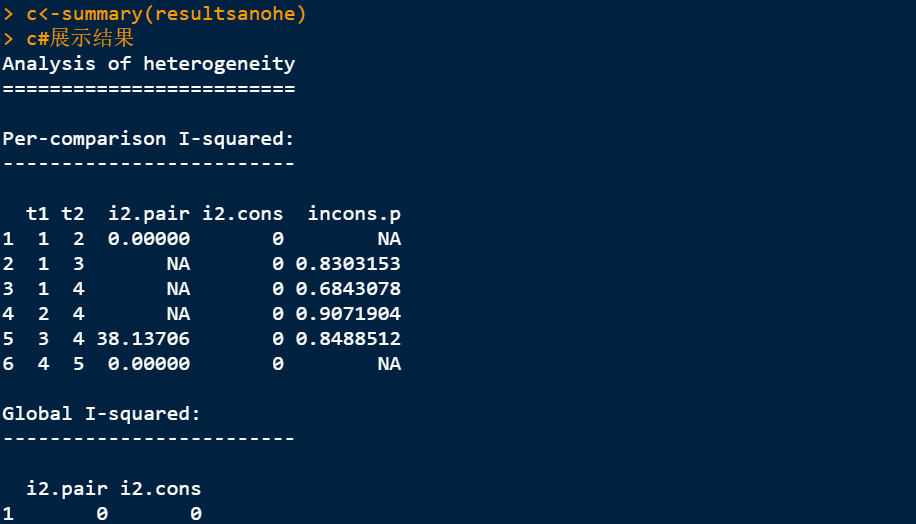
2. 展示结果

结果展示:i2.pair是直接比较的I2值,i2.cons是间接的I2值,incons.p是一致性检验的P值,从整个结果来看本数据没有明显的异质性。
3.绘制森林图并导出
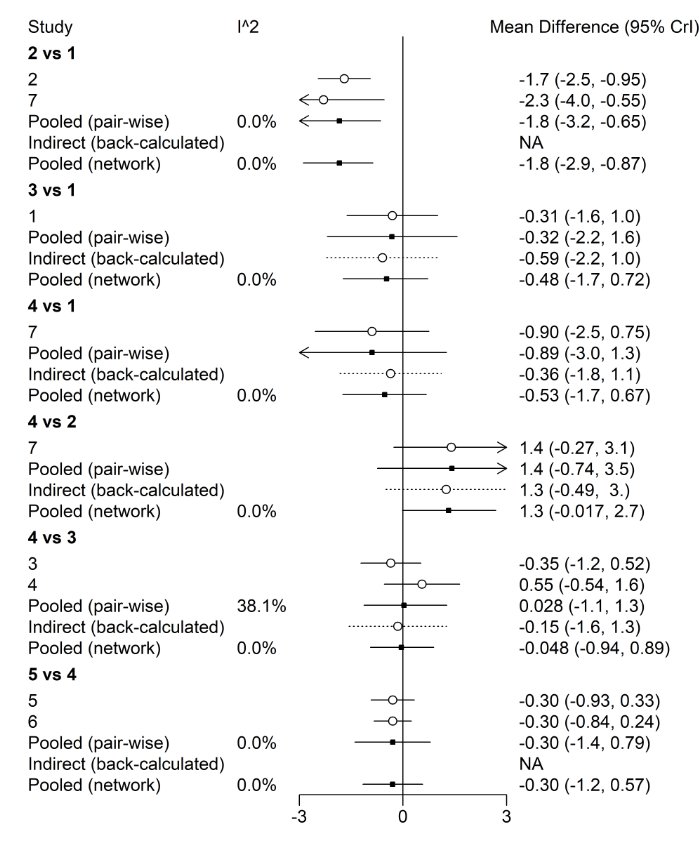
好了,关于网状Meta分析的内容基本就介绍完了,前面的步骤当中的最后一步也就是第六步,主要是进行偏倚风险评估、文献质量评价、敏感性分析、亚组分析、meta回归等等,这些跟普通Meta分析是一样的,这里就不再过多介绍了,我在《系统评价/Meta分析零基础从入门到精通》这门课里都有详细的讲解。










![[Qt] QString::fromLocal8Bit 的使用误区](https://img-blog.csdnimg.cn/direct/62600f6b003a467495d5f47db140b1b2.png)
