CRISPR-Cas9知识学习笔记
https://www.163.com/dy/article/FGCP58KC0532AN5N.html
https://crispr.dbcls.jp
CRISPR(clustered regularly interspaced short palindromic repeats,成簇的规律间隔短回文重复序列)和CRISPR-associated protein 9(Cas9)共同组成的这套CRISPR-Cas9系统是一种细菌抵抗噬菌体DNA注入和质粒转移的天然防御机制。该RNA引导的DNA靶向编辑工具已被科学家用在基因组编辑、转录干扰、表观遗传调控等多个领域。
一、天然情况下,细菌如何抵抗噬菌体入侵?

图1 细菌天然免疫系统:细菌适应性免疫中的CRISPR-Cas9介导的DNA干扰
从图1可以看到,CRISPR locus由这些元件构成:一开始是个反式激活的RNA基因,编码特异的非编码RNA(trancrRNA,trans-activating CRISPR RNA,橙色矩形),与重复序列具有同源性,后面是各种cas基因(多种颜色的箭头),接着是CRISPR array(棕色的菱形是重复序列,彩色的是间隔)。而这些间隔序列是细菌从噬菌体DNA中获得的遗传元件:当噬菌体感染细菌,细菌激活相关的cas基因——Cas1,Cas2,和Csn2,将新的间隔序列(暗绿色)整合到自身的CRISPR array中。一旦整合,新的间隔序列(spacer)会与其他间隔序列共同转录到一个长的CRISPR RNA前体(pre-crRNA)中,此pre-crRNA含有重复序列(棕色线)和间隔序列(深绿色、蓝色、浅绿色和黄色线)。tracrRNA是分开转录的,之后重组进入pre-crRNA的重复序列(tracrRNA与重复序列互补),经RNA酶III酶切处理成成熟的crRNA。进一步,在其他未知的核酸酶的作用下,剪切crRNA的5’端,使得引导序列长为20nt。在干扰过程中,成熟的crRNA-tracrRNA结构指引Cas9核酸内切酶对这个在PAM(protospacer adjacent motif,原间隔序列临近基序;特征为NGG,这里N可为任意碱基)前携带20nt的与crRNA互补序列的外源DNA进行切割。如果噬菌体再次注入DNA,那么这个免疫系统将被激活,来干扰噬菌体DNA。

补充说明:关于tracrRNA基因的定位有少部分资料显示在Cas基因后,CRISPR array前(图2);另外,有些公众号文章说tracrRNA由重复序列转录而来。个人目前水平优先,所以觉得这两个点须要进一步核实。
二、CRISPR/Cas9在其它细胞中的应用
2.1 DNA剪切与修复

图3Type II Cas9(最上面一个 SpCas9最常见)
目前主要使用II型CRISPR系统(图3),它和其他类型的区别是,只需要一个DNA内切酶Cas9来对与sgRNA 20个碱基互补的带有PAM结构的双链DNA进行剪切。剪切后的DNA产生平末端的DSB(double strand break, 双链断裂)。之后,要么发生易出错的非同源末端连接(NHEJ),导致切割位点容易发生小的随机插入和/或缺失(Indels: insert/delete的合成词,目前貌似还是非正规用词 );要么进行高保真同源定向修复(HDR),这可利用同源修复模板在DSB位点进行精确修饰(图4)。

图4Cas9剪切及突变产生: ins, del, sub, knock in
2.2 CRISPR-Cas9效应复合物组装与sgRNA结合后的构象重排
这里的例子是化脓链球菌(S. pyogenes)的Cas9(即SpyCas9或SpCas9),是一个含有1368个氨基酸的多结构和多功能的DNA核酸内切酶。它的切割位点在PAM上游的第三个碱基,通过HNH(sgRNA互补的目标序列)和RuvC核酸酶结构域(非目标序列)。要识别特定序列并进行剪切,sgRNA与Cas9组合成一个复合体,其中sgRNA与Cas9结合起着关键的作用,能够使Cas9重构,变得具有活性。crRNA前20碱基使得Cas9具有靶序列特异性,tracrRNA来招募Cas9蛋白。在这个系统中,有一个所谓的 种子序列 , 20碱基的间隔序列的3’端10-12个核苷酸 。在种子序列的错配以及本身同源性都会严重影响系统特异性和脱靶效率。

图5 sgRNA二级结构,灰色矩形区域代表全长sgRNA支架中的额外的重复序列与其重复反义序列,在基因组工程设计sgRNA时候通常是被去掉的。黄色区域代表sgRNA的3’尾巴,这对于Cas9功能不是必要的,在sgRNA-bound结构中是被省略掉的。
PAM序列非常关键,能够起到识别自身和外来的序列。如果PAM发生单一突变(原单词是single mutation,应该可以理解为点突变),那么就能够让噬菌体入侵宿主。在sgRNA互补之前,首先是寻找PAM序列,如果没有合适的PAM,那么通过蛋白三维结构的坍塌,CRISPR-Cas9复合体会离开DNA,直到找到合适的PAM。一旦找到PAM,Cas9就使DNA局部解链,RNA进入,与DNA互补,形成RNA-DNA结构。sg种子区域序列与靶DNA的完美互补是很重要的。
2.4 CRISPR–Cas9介导的DNA靶定与剪切模型
- 首先,guide RNA的结合,使得Cas9从一个未激活的构象变成具有DNA识别能力的构象。RNA种子序列先形成A型构象,为目标结合和链入侵,PAM识别位点预先形成用来PAM识别。
- 然后,Cas9 结合到PAM序列,使得酶能够去识别附近的潜在的DNA靶序列。
- 一旦Cas9 在PAM附近找到了潜在的靶序列,会开始解双螺旋并继续检查剩余的靶序列。磷酸锁环稳定解旋的目标DNA,且第一个碱基开始翻转向上,与guide RNA碱基配对。而Cas9继续与非靶链上的翻转碱基作用,促进双螺旋解开。
- 接着,碱基配对伴随着Cas9构象改变,促进种子序列前面的guide RNA从限制中释放出来,也形成配对,这个过程促使Cas9构象持续变化,直到到达有活性的状态。
- 最终,guide RNA与目标DNA完全互补使得HNH具有稳定的,具有活性的构象,来剪切目标链DNA。与此同时,引起更大的构象变化,使得非目标链DNA进入RuvC催化中心被剪切,这种转态下,Cas9中牢牢结合在靶点序列上,直到其他的细胞因子过来替代它。

图6剪切示意图
三、其它资料:CRISPR-Cas9向导RNA的类型

图7CRISPR-Cas9向导RNA的类型
(A) 野生型化脓性链球菌(S. pyogenes)CRISPR系统使用的是分开的crRNA与tracrRNA。 Alt-R CRISPR-Cas9系统使用的crRNA和tracrRNA序列模拟的正是化脓性链球菌的序列。为了提高效率和便于生产,已经对其长度进行优化。
(B)另一个通用方法是用一个短的连接序列将crRNA与tracrRNA连接成一个融合的sgRNA。
对于两种向导类型,crRNA(粗绿色条)中的原间隔序列元件都赋予了剪切位点特异性。crRNA和tracrRNA与Cas9酸内切酶形成复合物,指导并激活Cas9对基因组DNA进行切割。除了原间隔元件,所有SpyCas9(或SpCas9)的靶序列需要一个相反链上紧接着的短的NGG PAM序列。在缺少PAM序列的位点剪切不会发生。双链切割一般发生在靶序列上距离PAM位点3个碱基的位置。
文:什么是sgRNA
https://www.zhihu.com/question/67234694
https://www.bilibili.com/video/BV15x411Z7kf/?spm_id_from=333.337.search-card.all.click
sgRNA(single guide RNA)是向导RNA(guide RNA,gRNA),在RNA编辑的过程中引导尿苷残基插入或缺失到动质体(kinetoplastid)中,属于一种小型非编码RNA,可与pre-mRNA配对。gRNA编辑RNA分子,长度大约60-80个核苷酸,由单独的基因转录。
文: sgRNA设计工具,让基因编辑不再高冷!
https://zhuanlan.zhihu.com/p/89496017
CRISPR/Cas9 最常用基因编辑系统。CRISPR/Cas9包括2部分:Cas9核酸内切酶和sgRNA(single guide RNA),sgRNA由天然的tracrRNA (transactivating crRNA)和crRNA (CRISPR RNA)融合而来。
使用CRISPR/Cas9工具进行基因敲除等基因编辑时,首先要进行sgRNA设计。理论上只要根据PAM序列(SpCas9识别的最佳PAM是5-NGG-3)对所需靶向的物种基因组进行扫描,即可设计所有可能的sgRNA。但如何设计合理有效的sgRNA则需要谨慎考虑,因为这将决定其基因编辑结果。
文:CRISPR中sgRNA的设计
https://zhuanlan.zhihu.com/p/539819746
CRISPR-Cas9系统可用位置更多。理论上基因组中每8个碱基就能找到一个可以用CRISPR-Cas9进行编辑的位置,这一技术能对任一基因操作。此外还可将Cas9蛋白连接其他功能蛋白,在特定DNA序列上研究这些蛋白对细胞的影响。CRISPR-Cas9系统的使用极为方便,只需要替换一段核酸序列。
gRNA是什么?
CRISPR/Cas9系统的工作原理是 crRNA( CRISPR-derived RNA )通过碱基配对与tracrRNA(trans-activating RNA)结合形成 tracrRNA/crRNA复合物,此复合物引导核酸酶Cas9蛋白在与crRNA 配对的序列靶位点剪切双链DNA。而通过人工设计crRNA和tracrRNA这两种RNA,可以改造形成具有引导作用的sgRNA(single guide RNA),从而引导Cas9对DNA定点切割。
CRISPR系统包含两个组件,一个是sgRNA,另一个就是Cas 9蛋白。sgRNA是一个短的合成RNA,只有20bp大小,可以与Cas9蛋白结合,所以在设计sgRNA之前,应在基因组上寻找PAM序列(PAM:Protospacer Adjacent Motif),PAM序列是有固定形式的,来自于不同菌种属的Cas,它的PAM序列形式是不一样的。目前主流是SpCas 9(Cas 9蛋白来源于S. pyogenes (化脓链球菌)II型CRISPR系统)。所以,我们的PAM序列为**-NGG的形式**,“N”可以是A,T,C,G中的任何一个。Cas 9相当于是限制性核酸内切酶,使PAM序列前形成DSB(Double Strand Break)。所以,sgRNA其实相当于是向导,告诉Cas 9蛋白在哪进行切割。
为了提高CRISPR/Cas9 的特异性,使用Cas9切口酶和一对sgRNA,两个相近的切口造成DNA双链断裂,诱导细胞发生非同源末端连接修复,造成目的基因的突变。
利用CRISPR/Cas9进行基因的编辑,首先要构建有效的sgRNA。一般地,基因特异的sgRNA模板序列为位于PAM序列(Protospacer Adjacent Motif)前间区序列邻近基序。这是一种见于crRNA分子的短核苷酸基序,可以被Cas9蛋白特异性识别并切割的 20 个nt。而 PAM 序列的特征为 NGG。
sgRNA的设计
sgRNA的设计原则
(1)sgRNA的长度:S. pyogenes II型CRISPR系统(SpCas 9)一般为20nt。
(2)sgRNA序列的碱基组成:基因特异的sgRNA 模板序列为位于PAM序列前,PAM序列的特征为NGG(N可以为任意核苷酸),所以选择3’末端含有GG的sgRNA,这样可以构成PAM序列。同时,sgRNA的序列应避免以4个以上的T结尾,GC%含量最佳为30%-70%(40%-60%)。
(3)sgRNA的序列与On-target和Off-target的匹配数都应尽可能的高,一般大于60,认为是可用的?
(4)如果构建U6启动子或T7启动子驱动sgRNA的表达载体,需要考虑sgRNA的5’碱基为G或GG,来提高其转录效率?
(5)全基因的脱靶效应分析,需要考虑脱靶位点的错配碱基数,最多不超过5个。
(6)如果想要造成基因移码突变,需要尽量靠近基因编码区的ATG下游,最好位于第一或第二外显子上。

2.2 sgRNA的设计步骤
sgRNA通常通过网站在线设计,下面介绍介个常用网站
网站1:通过网址:https://www.deskgen.com/landing/cloud进行sgRNA的设计,进入网址后点击KNOCK OUT进入设计页面,输入想要设计sgRNA的名称后,进行sgRNA的选择。由于内含子在基因表达的过程中,不会进行表达且会被删掉,所以我们在设计sgRNA时一般在靠近启动子的第一个CDS区(也就是外显子的保守结构域)进行sgRNA的选择。在网站上,On-Target和Off-target都是有评分的,一般来说,评分越高,则sgRNA越好。On-Target其实就是sgRNA对目标位点识别后与DNA模板进行识别切割的的效率,所以准确性越高,On-Target的评分越高。Off-Target也就是脱靶效应,由于CRISPR技术的切割是由sgRNA根据PAM序列定位识别位点,从而进行切割,但是如果sgRNA识别的位点是错误的,就产生了错误切割,这样就产生了脱靶效应。所以说,在进行sgRNA选择的时候,要尽可能地选择脱靶效率比较低的,也就是Off-target分值比较高的。另外,符合sgRNA的其它设计原则就可以了。
网站2:打开网站 http://crispr.mit.edu,将基因名称,邮箱,第二外显子序列输入到对话框,点击 Agree and submit,等待网站设计成对的 sgRNA 序列。一般推荐网站设计,因为网站可以预测脱靶位点数,避免基因脱靶产生。
网站3:http://crispr.dfci.harvard.edu/SSC/
这也是一个很常用且好用的网站~
cas9 基因敲除
CRISPR/Cas9 系统作为细菌和古细菌的获得性免疫系统, 通过 RNA 介导特异性的切割外源遗传物质, 用以对抗入侵的病毒和质粒。利用 Cas9 摧毁入侵 DNA 的 Type Ⅱ CRISPR/Cas 系统, 可以在体外进行基因编辑。
为了提高CRISPR/Cas9 的特异性,使用 Cas9 切口酶和一对 sgRNA,两个相近的切口造成 DNA 双链断裂, 诱导细胞发生非同源末端连接修复, 造成目的基因的突变。
利用 CRISPR/Cas9 进行基因的编辑,首先要构建有效的 sgRNA。一般地,基因特异的 sgRNA 模板序列为位于 PAM 序列(Protospacer Adjacent Motif)前间区序列邻近基序。这是一种见于 crRNA 分子的短核苷酸基序,可以被 Cas9 蛋白特异性识别并切割的 20 个 nt。 而 PAM 序列的特征为 NGG(其中 N 为任意核苷酸)
- 找敲除目的基因的外显子
根据目的基因选择待敲除靶基因位点找出敲除目的基因的外显子。 首先在第一个起始密码子 ATG 之后的外显子中找出特异性高的上下游序列。以小鼠基因 Th 为例。在 pubmed 上找到基因的 mRNA 的 CDS 区,选择第二个外显子作为敲除位点。
- 设计成对 sgRNA 序列
打开网站 http://crispr.mit.edu,将基因名称,邮箱,第二外显子序列输入到对话框,点击 Agree and submit,等待网站设计成对的 sgRNA 序列。一般推荐网站设计,因为网站可以预测脱靶位点数,避免基因脱靶产生。当然也可以手动选择特异的 sgRNA 序列。
设计成对的 sgRNA 序列。打开 Nickase analysis,如图所示,网站会给出多组成对 sgRNA 序列,如箭头所示,并且预测可能脱靶数。
03
分析成对 sgRNA 序列
打开 http://www.oligoevaluator.com,将 sgRNA 输入,点击 Calcute,得到基因分析。综合考虑 Tm(56~62)、GC content (45~60%) 及 secondary structure 来最终确定适宜的 sgRNA 序列,因此,高 score 的序列不一定是最佳序列。
如图,第一对 81 分的序列,Tm 值大于 62 度,而且存在稳定二级结构,所以并不推荐。所以应该从上述成对序列中继续寻找合适成对 sgRNA 序列。
04
根据找到的合适的 sgRNA 订购 oligo
05
关于 cas9 的几个注意事项
CRISPR-cas9 最主要的要求:PAM 序列为 NGG。
sgRNA,即 cas9 guide RNA,是引导 cas9 蛋白在基因编辑位点进行定向切割,所以一般是 20 个碱基,不含 PAM 序列。
设计的 sgRNA 一定有效吗?一般设计好的 sgRNA 会在细胞水平验证下 DNA 水平的编辑效率,通过对细胞 DNA 序列测序以及 T7EN1 酶切验证敲减效率。对于目的基因,更主要的是验证蛋白基因 mRNA 水平进行验证。
cas9 的相关应用:
(1)敲减细胞,可以选择 lenti-crisprv2 系统,通过包装慢病毒,可对目的细胞进行转染,并不断药筛得到稳定敲减细胞系。
(2)构建基因敲除编辑鼠。根据染色体修复方式又可分为 knockout 与 knockin 两种形式。
文:sgRNA设计,需注意问题
https://www.sohu.com/a/480422948_100001538
CRISPR-Cas9是一项可对基因组特定靶基因进行编辑的DNA操控技术,该系统由sgRNA和Cas9蛋白组成,Cas9蛋白在sgRNA的引导下对靶位点处的DNA双链进行剪切,并产生一个平末端的双链DNA缺口,进而启动DNA损伤修复机制,通过非同源末端链接(Non-homologous end joining,NHEJ)或同源重组(Homologous recombination,HR)的方式将断裂上下游两端的序列连接起来。
目前,CRISPR-Cas9基因编辑技术在疾病基础研究、靶点验证、药物分子的高通量筛选、以及遗传性疾病的治疗等领域得到越来越广泛的应用。sgRNA在CRISPR-Cas9基因编辑系统中具有准确识别靶基因序列的作用,其效果可影响编辑的效率、是否发生脱靶等,甚至对最终基因编辑的效果产生决定性作用。因此,设计合理有效的sgRNA是实现基因编辑的重要基础。
sgRNA的设计流程
靶基因信息的分析
数据库有NCBI、Ensembl等。找到目的基因,需进一步关注其所在基因座上下游基因情况、转录本数量、外显子数量及长度、翻译起始位点与终止位点等信息。然后再综合考量上述信息进行下一步的sgRNA 设计。
以查询人类Rag1基因为例。点击查询目的基因人类Rag1基因,显示出该基因的基本信息。转录本相关信息,外显子等相关信息,转录本基本结构…
靶区域的选择原则
以基因敲除(KO)为例,基因敲除可采用2种不同策略——移码突变和片段敲除,虽然不同的策略对于靶区域选择的参考标准有差异,但也需遵循以下原则:
1. 不影响其他基因,尤其是编码蛋白的基因。挑选靶区域时避免选择与其他基因重叠的区域(图a)。
2. 尽可能影响所有的转录本,敲除位点最好在编码区的前50%,但避免敲除ATG所在的位置(图b)。
3. 能影响蛋白的功能结构域。
对于片段敲除,需考虑更多因素:如片段敲除所敲除的外显子编码序列之和为非3的倍数(图3c),这样可使靶区域后面的序列发生移码,无法翻译出功能蛋白,从而使敲除更彻底。片段敲除所选定的敲除区域不超过10Kb,超过10Kb后编辑效率会降低。片段敲除的gRNA设计在内含子,这样能敲除整个外显子区,避免翻译出残留蛋白。并且gRNA的设计位点需靠近所敲除的外显子,这样可避免产生不可控的剪切信号而导致形成新的转录本。敲除区域前后序列尽量简单,方便后续的PCR鉴定。

图. 靶区域选择示意图
(a)基因A与基因B 共有一个外显子(标蓝的外显子),因此选择靶区域时应遵循不影响其他基因的原则,不以共有的外显子作为靶区域。(b)存在多个转录本时,为敲除所有转录本,应选择在多个转录本均存在,且在编码区的前50%的外显子(此处选择标红的外显子)作为靶区域。(c)片段敲除时,因基因片段过大不能全部敲除而选择敲除部分外显子,敲除片段的编码序列之和应为非3的倍数,使后面的蛋白发生移码突变。
gRNA的设计
目前有较多gRNA设计在线工具,如张锋的CRISPOR(http://crispor.tefor.net/),只需输入目标序列,选定好种属基因组与相应的PAM,则可以得出多个 gRNA,以及每个gRNA对应的特异性、切割效率和潜在脱靶位点,一般选择特异性、切割效率得分高的gRNA作后续实验(图4)。
如果需要手动设计gRNA,则需要考虑其特异性与切割效率。在靶点设计时要综合考虑所有候选靶点的序列、位置、正负链、GC含量、潜在的脱靶位点等信息。


脱靶分析
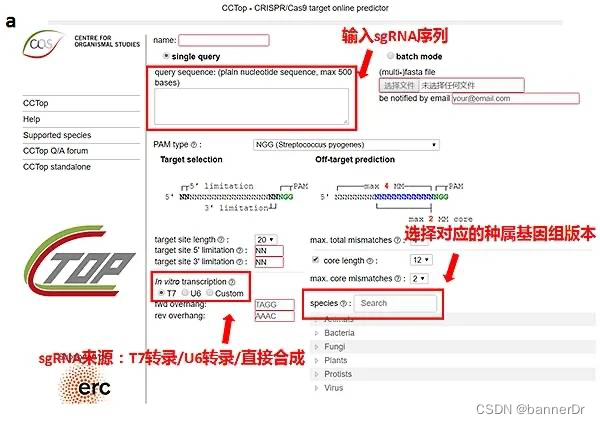
根据选择的sgRNA,通过生物信息学方法,对sgRNA进行脱靶分析。推荐使用CCTop(https://cctop.cos.uni-heidelberg.de:8043/)在线网页预测。将sgRNA序列输入,选定相应种属基因组进行分析(图5)。并且对获得的遗传材料进行检测。挑选前10个潜在脱靶位点,通过PCR测序验证是否脱靶。如果实验要求较为严格的,则需要通过全基因组测序鉴定脱靶情况。
sgRNA设计仅是基因编辑方案设计中的一环,基因编辑方案还需考虑目的基因转录本分析、转染方法、细胞克隆形成能力等多种因素