文章目录
- 权重初始化方法
- Xavier初始化(Xavier initialization)
- Kaiming初始化,也称为He初始化
- LeCun 初始化
- 正态分布与均匀分布
- Orthogonal Initialization
- Sparse Initialization
- n_in和n_out
- 代码实现
权重初始化方法
Xavier初始化(Xavier initialization)
是一种用于初始化神经网络权重的方法,也称为Glorot初始化。更有效地传播信号并减少梯度消失或梯度爆炸的问题。适用于激活函数为tanh或sigmoid的情况。
Xavier初始化的计算方法如下:
- Glorot(或 Xavier)初始化:
- 适用于激活函数如sigmoid和tanh。
- 初始化公式:
σ
=
2
n
in
+
n
out
\sigma = \sqrt{\frac{2}{n_{\text{in}} + n_{\text{out}}}}
σ=nin+nout2
其中, n in n_{\text{in}} nin 是输入单元数, n out n_{\text{out}} nout 是输出单元数。
对于单个神经元的权重w,从均匀分布或正态分布中随机采样,具体取决于所选择的激活函数:
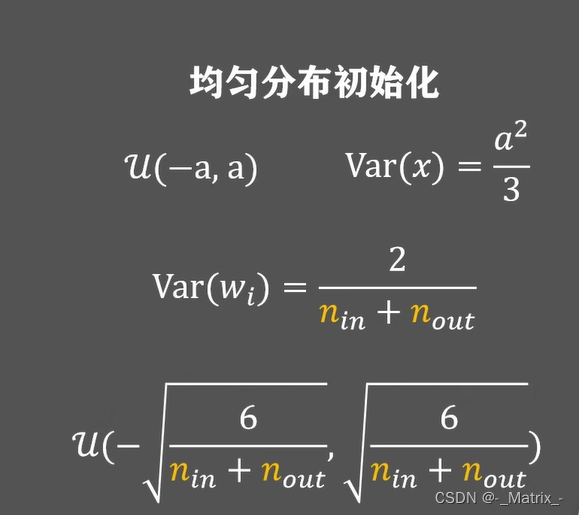
- 如果使用
tanh激活函数,从均匀分布采样:- 采样范围:
[-sqrt(6 / (n_in + n_out)), sqrt(6 / (n_in + n_out))] - 其中
n_in是上一层的输入节点数量,n_out是当前层的输出节点数量。
- 采样范围:
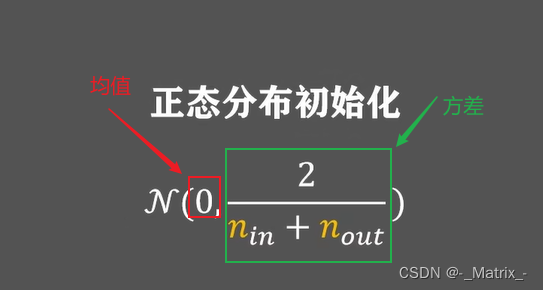
- 如果使用
sigmoid激活函数,从正态分布采样:- 均值:0
- 方差:
sqrt(2 / (n_in + n_out)) - 其中n_in是上一层的输入节点数量,n_out是当前层的输出节点数量。
Kaiming初始化,也称为He初始化
- He 初始化:
- 适用于ReLU及其变种(如LeakyReLU)激活函数。
- 初始化公式: σ = 2 n in \sigma = \sqrt{\frac{2}{n_{\text{in}}}} σ=nin2
这种初始化方法主要用于修正线性单元(Rectified Linear Units,ReLU)激活函数的神经网络。
与Xavier初始化适用于tanh和sigmoid等S型激活函数不同,Kaiming初始化专门针对ReLU激活函数的特性进行优化。ReLU是一个常用的非线性激活函数,它在输入大于零时保持不变,在输入小于等于零时输出为零。
Kaiming初始化的计算方法如下:
对于单个神经元的权重w,从均匀分布或正态分布中随机采样,具体取决于所选择的激活函数:
-
如果使用
ReLU激活函数,从正态分布采样:- 均值:0
- 方差:
sqrt(2 / n_in) - 其中n_in是上一层的输入节点数量。
-
对于带有ReLU激活的卷积层,可以使用相同的初始化方法,只是需要考虑卷积层的输入通道数量(即n_in)。
LeCun 初始化
- 适用于Sigmoid激活函数。
- 初始化公式: σ = 1 n in \sigma = \sqrt{\frac{1}{n_{\text{in}}}} σ=nin1
正态分布与均匀分布
- 使用较小的标准差(如0.01)从正态分布中采样权重。
- 使用较小的范围(如-0.01到0.01)从均匀分布中采样权重。
Orthogonal Initialization
- 使用正交矩阵初始化权重。这种初始化方法对于某些任务和模型架构可能很有益。
Sparse Initialization
- 将大部分权重初始化为0,只初始化一小部分非零的权重。
n_in和n_out
n_in和n_out分别表示神经网络层的输入节点数量和输出节点数量。这些节点也称为神经元,它们是网络的基本组成部分。
-
n_in:代表上一层(前一层)的节点数量,也就是当前层的输入数量。在神经网络中,每个神经元都会接收来自上一层所有节点的输入,这些输入被加权和后传递给当前神经元的激活函数。因此,n_in指的是上一层与当前层之间的连接数量。
-
n_out:代表当前层的节点数量,也就是当前层的输出数量。每个神经元会将经过激活函数处理后的结果传递给下一层所有节点,形成下一层的输入。因此,n_out指的是当前层与下一层之间的连接数量。
代码实现
#include <iostream>
#include <Eigen/Dense>
#include <random>
#include <cmath>
Eigen::MatrixXd glorotInitialize(int rows, int cols);
Eigen::MatrixXd heInitialize(int rows, int cols);
Eigen::MatrixXd lecunInitialize(int rows, int cols);
Eigen::MatrixXd normalDistributionInitialize(int rows, int cols, double std_dev=0.01);
Eigen::MatrixXd uniformDistributionInitialize(int rows, int cols, double limit=0.01);
Eigen::MatrixXd orthogonalInitialize(int rows, int cols);
// Sparse Initialization需要额外参数来确定稀疏度,这里我们使用一个简化版本,指定一个非零的权重数。
Eigen::MatrixXd sparseInitialize(int rows, int cols, int nonZeroCount);
//1. **Glorot (Xavier) Initialization**:
Eigen::MatrixXd glorotInitialize(int rows, int cols) {
std::random_device rd;
std::mt19937 gen(rd());
double limit = sqrt(6.0 / (rows + cols));
std::uniform_real_distribution<> dis(-limit, limit);
Eigen::MatrixXd matrix(rows, cols);
for(int i = 0; i < rows; i++) {
for(int j = 0; j < cols; j++) {
matrix(i, j) = dis(gen);
}
}
return matrix;
}
//**He Initialization**:
Eigen::MatrixXd heInitialize(int rows, int cols) {
std::random_device rd;
std::mt19937 gen(rd());
double std_dev = sqrt(2.0 / rows);
std::normal_distribution<> dis(0, std_dev);
Eigen::MatrixXd matrix(rows, cols);
for(int i = 0; i < rows; i++) {
for(int j = 0; j < cols; j++) {
matrix(i, j) = dis(gen);
}
}
return matrix;
}
//3. **LeCun Initialization**:
Eigen::MatrixXd lecunInitialize(int rows, int cols) {
std::random_device rd;
std::mt19937 gen(rd());
double std_dev = sqrt(1.0 / rows);
std::normal_distribution<> dis(0, std_dev);
Eigen::MatrixXd matrix(rows, cols);
for(int i = 0; i < rows; i++) {
for(int j = 0; j < cols; j++) {
matrix(i, j) = dis(gen);
}
}
return matrix;
}
//4. **Normal Distribution Initialization**:
Eigen::MatrixXd normalDistributionInitialize(int rows, int cols, double std_dev) {
std::random_device rd;
std::mt19937 gen(rd());
std::normal_distribution<> dis(0, std_dev);
Eigen::MatrixXd matrix(rows, cols);
for(int i = 0; i < rows; i++) {
for(int j = 0; j < cols; j++) {
matrix(i, j) = dis(gen);
}
}
return matrix;
}
//5. **Uniform Distribution Initialization**:
Eigen::MatrixXd uniformDistributionInitialize(int rows, int cols, double limit) {
std::random_device rd;
std::mt19937 gen(rd());
std::uniform_real_distribution<> dis(-limit, limit);
Eigen::MatrixXd matrix(rows, cols);
for(int i = 0; i < rows; i++) {
for(int j = 0; j < cols; j++) {
matrix(i, j) = dis(gen);
}
}
return matrix;
}
//6. **Orthogonal Initialization**:
Eigen::MatrixXd orthogonalInitialize(int rows, int cols) {
// 创建一个随机矩阵
std::random_device rd;
std::mt19937 gen(rd());
std::normal_distribution<> dis(0, 1);
Eigen::MatrixXd randomMatrix(rows, cols);
for(int i = 0; i < rows; i++) {
for(int j = 0; j < cols; j++) {
randomMatrix(i, j) = dis(gen);
}
}
// 使用QR分解获得正交矩阵
Eigen::HouseholderQR<Eigen::MatrixXd> qr(randomMatrix);
Eigen::MatrixXd orthogonalMatrix = qr.householderQ();
// 如果您需要一个具有特定维度的正交矩阵(例如rows != cols),您可以选择一个子矩阵
return orthogonalMatrix.block(0, 0, rows, cols);
}
//7. **Sparse Initialization**:
Eigen::MatrixXd sparseInitialize(int rows, int cols, int nonZeroCount) {
Eigen::MatrixXd matrix = Eigen::MatrixXd::Zero(rows, cols);
std::random_device rd;
std::mt19937 gen(rd());
std::uniform_real_distribution<> dis(-1, 1);
for(int i = 0; i < nonZeroCount; i++) {
int r = rand() % rows;
int c = rand() % cols;
matrix(r, c) = dis(gen);
}
return matrix;
}
int main() {
int rows = 5;
int cols = 5;
// Glorot Initialization
Eigen::MatrixXd weights_glorot = glorotInitialize(rows, cols);
std::cout << "Glorot Initialized Weights:" << std::endl << weights_glorot << std::endl << std::endl;
// He Initialization
Eigen::MatrixXd weights_he = heInitialize(rows, cols);
std::cout << "He Initialized Weights:" << std::endl << weights_he << std::endl << std::endl;
// LeCun Initialization
Eigen::MatrixXd weights_lecun = lecunInitialize(rows, cols);
std::cout << "LeCun Initialized Weights:" << std::endl << weights_lecun << std::endl << std::endl;
// Normal Distribution Initialization
Eigen::MatrixXd weights_normal = normalDistributionInitialize(rows, cols);
std::cout << "Normal Distribution Initialized Weights:" << std::endl << weights_normal << std::endl << std::endl;
// Uniform Distribution Initialization
Eigen::MatrixXd weights_uniform = uniformDistributionInitialize(rows, cols);
std::cout << "Uniform Distribution Initialized Weights:" << std::endl << weights_uniform << std::endl << std::endl;
// Sparse Initialization
int nonZeroCount = 10; // As an example, set 10 weights to non-zero values
Eigen::MatrixXd weights_sparse = sparseInitialize(rows, cols, nonZeroCount);
std::cout << "Sparse Initialized Weights with " << nonZeroCount << " non-zero values:" << std::endl << weights_sparse << std::endl;
return 0;
}