FADE软件参考文献
参考文献:片段化酶诱导的双链伪影的表征和缓解 - PMC (nih.gov)
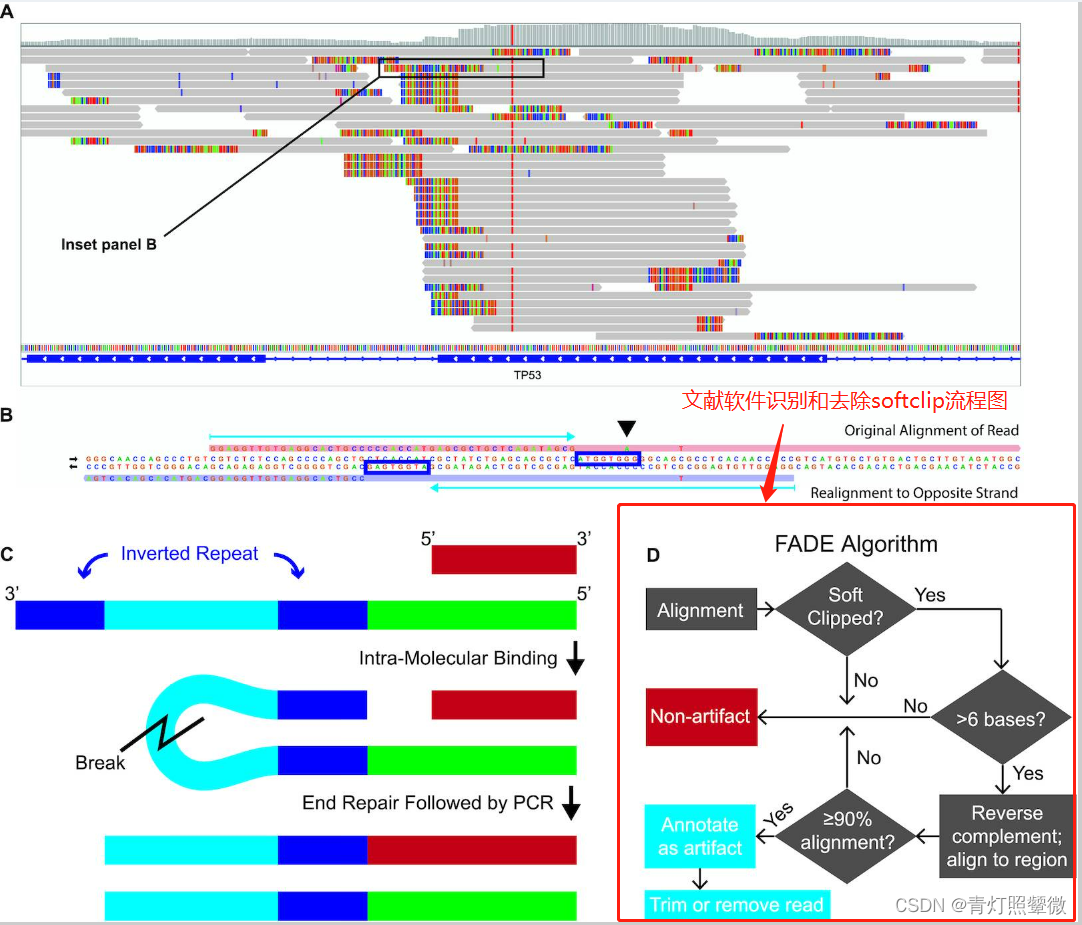
文献提供的酶切产生的错误识别和去除软件FADE:软件git地址
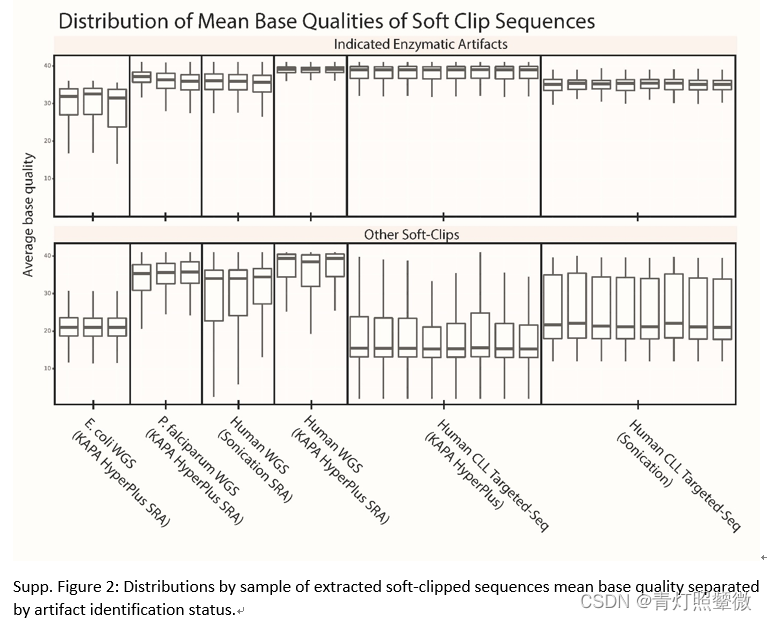
文献补充材料图:由酶切产生的人工错误序列的碱基质量值偏高,相对其他softclip的质量值。
FADE软件下载
下载linux版FADE
wget https://github.com/blachlylab/fade/releases/download/v0.6.0/fade.many-linux-x86_64.tar.gz
软件使用
./fade annotate ${yourbam} > anno.bam # 先增加bam注释
./fade stats anno.bam > stats.out # 统计是否是artifact softclip(包含比对信息)
./fade stats-clip anno.bam > stats-clip.out # 统计是否是artifact softclip(质量值和是否是artifact)
stats-clip.out结果文件示例:
| qname | sc_q_scores | sc_seq | sc_avg_bq | avg_bq | art_status |
|---|---|---|---|---|---|
| A1:2:3:4 | F,FF,:F:F,FFFFFFF | TCGGGCCGGTCGTCTTT | 31.4444 | 31.8671 | false |
| A1:2:3:5 | F,FF,:F:F,FFFF::F:FF:F | TCGGGTTCGTGACCTCAGCGGC | 31.4444 | 31.8671 | ture |
各列说明:
| 表头名 | 说明 |
|---|---|
| qname | read名称 |
| sc_q_scores | softclip序列质量值 |
| sc_seq | softclip序列 |
| sc_avg_bq | softclip序列质量值均值 |
| avg_bq | 序列的质量值均值 |
| art_status | softclip是否是artifact [value: true/false] |
更多了解:
酶切片段化建库,奇怪的知识增加了
酶切文库构建试剂盒 v2 升级款来袭
** FFPE 样本的融合基因检测:DNA or RNA
超声法与酶切法随机打断基因组方法的比较