Overview
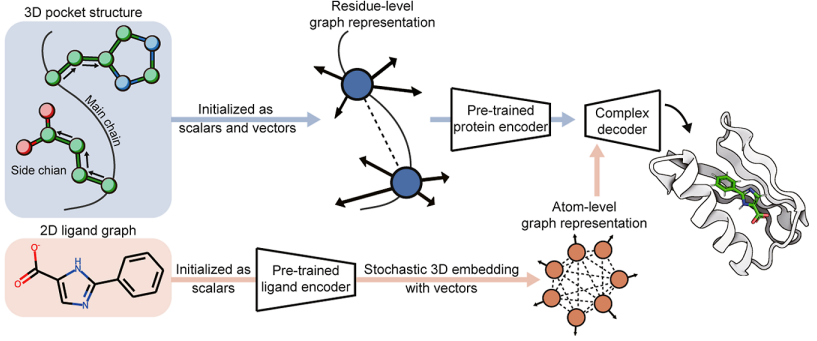
该论文解决了药物开发中蛋白质-配体复合结构灵活建模的挑战。作者提出了一种名为FlexPose的新型深度学习框架,它可以直接对复杂结构进行建模,而不需要传统的采样和评分策略。 该模型结合了标量-向量双特征表示和 SE(3)等变网络设计来处理动态结构变化。 它还利用构象感知预训练和弱监督学习策略来提高模型在看不见的化学空间中的通用性。 作者证明 FlexPose 的性能优于流行的对接工具和最新的深度学习方法,特别是在涉及蛋白质构象变化的任务中。 该模型还为后期分析提供亲和力估计和模型置信度。 该研究的主要发现包括 FlexPose 在预测配体结合构象方面的卓越性能以及蛋白质和配体相似性对模型性能的影响。
Highlights
- FlexPose通过将蛋白质-配体对表示为带有标量和向量特征的图(节点和边),然后通过编码器-解码器风格的网络处理。它能够直接预测和优化复合物结构,而不需要预先生成的构象。(实现的功能类似于**“柔性对接”**)
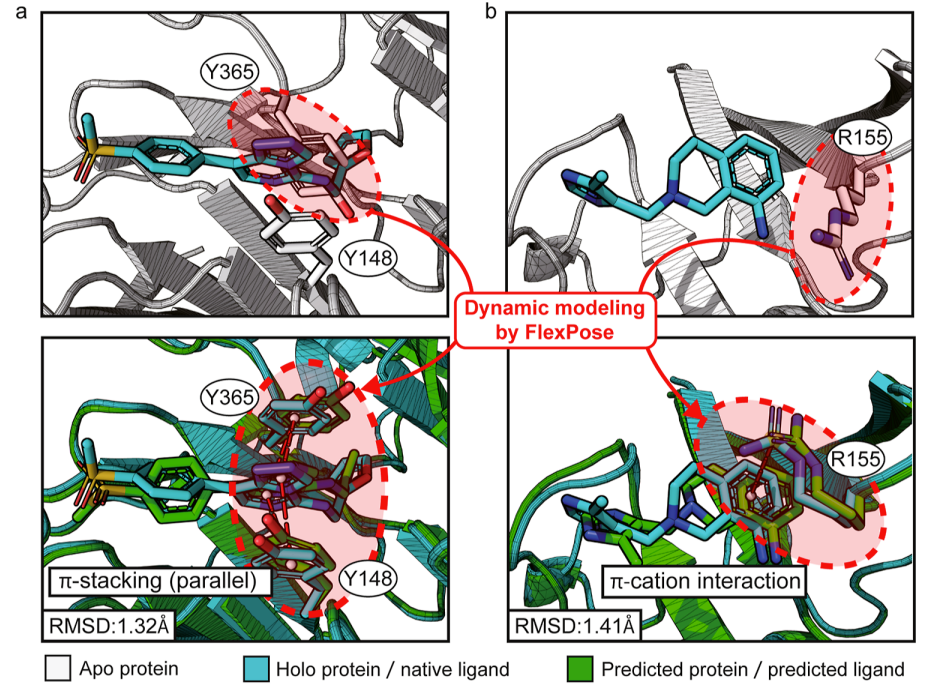
- FlexPose在预测蛋白质构象变化的任务中,特别是在涉及蛋白质构象变化的任务中,显著优于所有测试的流行对接工具和最近的深度学习方法。它在某些任务中的准确率比第二佳方法高出37.58%。
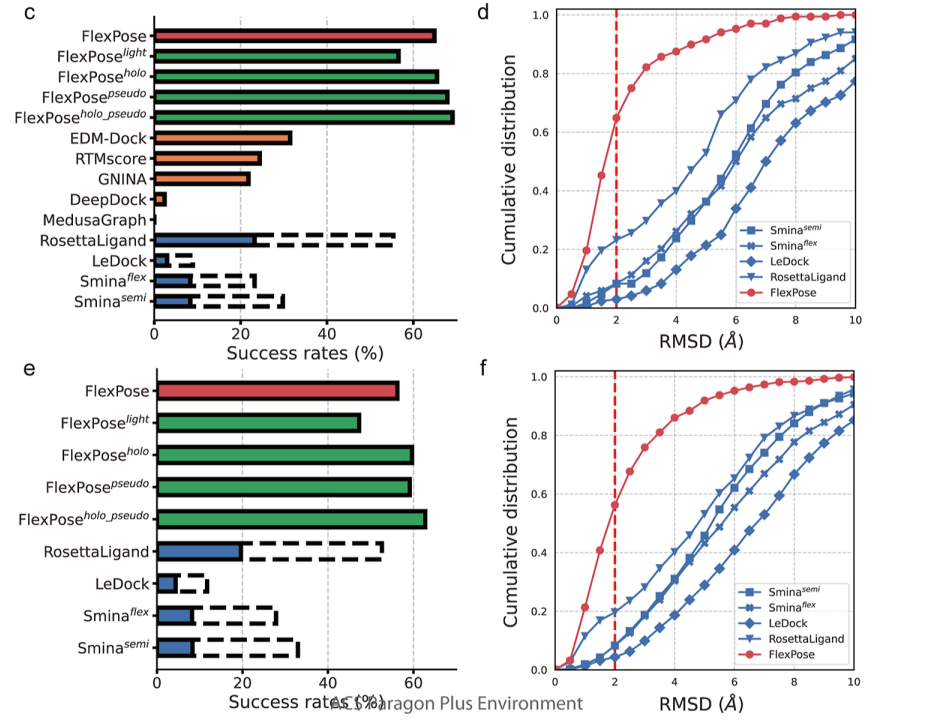
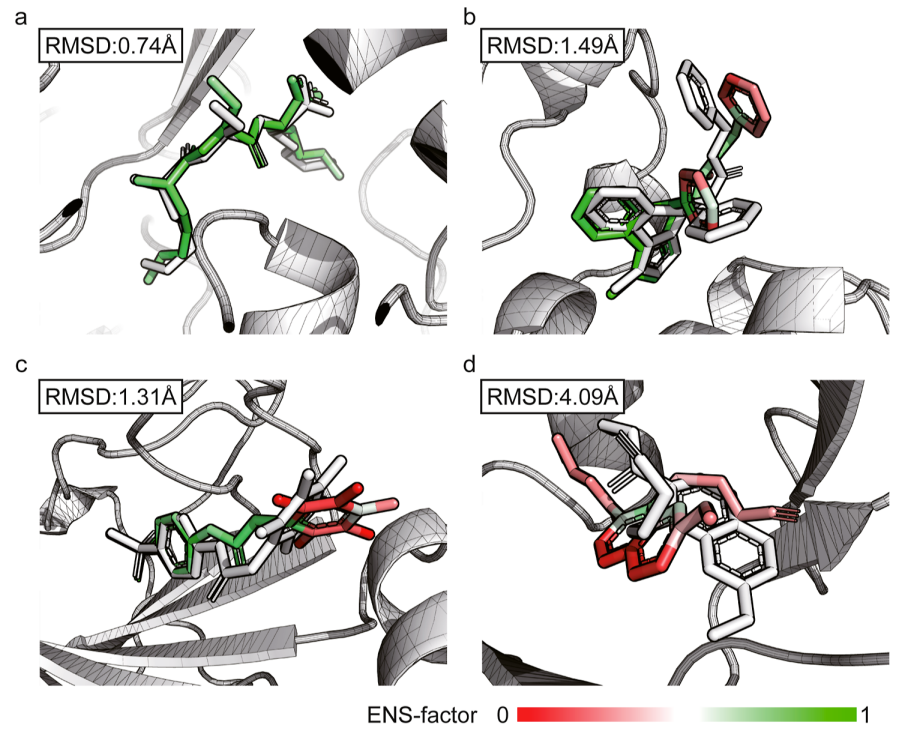
- 通过构象感知预训练和弱监督学习,FlexPose能够在数据稀缺的情况下,通过使用低置信度结构(例如,由对接工具采样的结构)来扩展学习到的化学空间。此外,FlexPose提供了亲和力估计和模型置信度,这对于蛋白质-配体相互作用的后期分析非常有用。模型置信度(ENS因子)可以帮助专家在结构预测中识别潜在的错误。
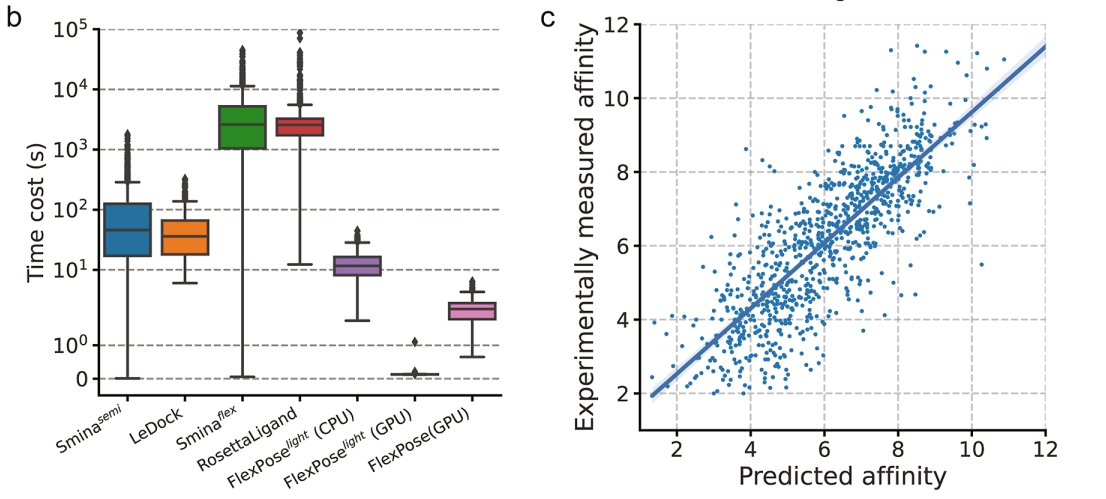
- 在多种测试集上,FlexPose展示了其在预测配体结合构象、蛋白质构象变化以及亲和力估计方面的高效性和准确性。它在处理蛋白质构象变化时的效率尤其突出,比传统的对接工具快得多。
总之这份工作展示了FlexPose在药物发现和设计领域的潜力,特别是在处理蛋白质-配体相互作用和结构预测方面的应用。
项目地址:https://github.com/tiejundong/FlexPose







![[UI5 常用控件] 05.FlexBox, VBox,HBox,HorizontalLayout,VerticalLayout](https://img-blog.csdnimg.cn/direct/9382617aa00640019700dce227df4fa5.png)