新系列 —— R高级绘图,准备整理所有曾经绘制过的图图和未来需要的图图们的代码!预计这个系列会囊括所有常见图形,只提供高级绘图代码,基础绘图主要在 R语言绘图 系列中进行介绍,这个系列咱们主打:需要XX图?没问题!来这里!找到你要画的图!点进去!直接复制粘贴RUN!
更新顺序随机,小伙伴们有急需想要的图图也可以后台告诉我!我给它优先更新!
带边缘分布散点图
带边缘分布散点图(Scatter Plot with Distribution Plots),用于同时显示两个变量的散点分布以及每个变量的边缘分布。它将散点图与边缘直方图或核密度估计图结合起来,提供了更全面的数据分析视角。在这种图中,通常沿着图的边缘或周围绘制边缘直方图或核密度估计图,以显示每个变量的分布情况,而散点图则展示了两个变量之间的关系。
带边缘分布散点图经常用于展示两个生物学特征之间的关系,并且可以提供关于每个特征的单独分布的信息。以下是一些在生物信息学领域中使用带边缘分布散点图的常见应用:
- 基因表达和关联分析: 通过在横轴和纵轴上分别表示两个基因的表达水平,可以使用带边缘分布散点图来展示它们之间的相关性。这有助于发现共表达基因、检测共调控模式以及了解基因表达的整体分布。
- 疾病与基因关联: 用于研究基因与特定疾病之间的关联。例如,可以将基因的变异情况与患病状态绘制在散点图上,同时在边缘显示基因变异和患病状态的分布情况。
- 蛋白质-蛋白质相互作用网络: 在蛋白质组学研究中,可以使用带边缘分布散点图来可视化蛋白质之间的相互作用关系。横轴和纵轴上的变量可能分别表示两个蛋白质的特定性质,而散点图的模式和趋势有助于了解它们之间的相互作用。
- 突变和表达关系: 当研究基因突变与基因表达之间的关系时,带边缘分布散点图可以用于同时显示突变信息和基因表达水平,例如,可以将基因表达量在横轴上表示,基因突变情况在纵轴上表示,并在边缘绘制基因表达和突变的分布,这有助于发现突变与表达之间的关联。
- 单细胞: 在单细胞研究中,带边缘分布散点图可以用于可视化单细胞表达谱中不同细胞群体之间的关系,同时提供每个基因的边缘表达分布信息。
- ……(让我们聪明的小脑瓜子自由发挥吧!!!)
总的来说,带边缘分布散点图提供了一个直观的方式来同时观察两个变量之间的关系及其各自的分布情况,可用于多种分析,包括基因表达、相互作用网络、突变与表达关系等,有助于我们发现两个生物学特征之间潜在的关联和模式。
绘图代码
这里,我们使用了 ggplot2 包来创建带边缘分布的散点图,所用数据集为R自带的iris数据集(大家替换为自己的数据即可,记得与示例数据格式保持一致),其中展示了Sepal.Length与Sepal.Width之间的关系,同时通过颜色区分了不同物种,也就是Species。
首先绘制散点图
# 带边缘分布散点图(Scatter Plot with Distribution Plots)
# 我们使用R自带的iris数据集
head(iris)
# Sepal.Length Sepal.Width Petal.Length Petal.Width Species
# 1 5.1 3.5 1.4 0.2 setosa
# 2 4.9 3.0 1.4 0.2 setosa
# 3 4.7 3.2 1.3 0.2 setosa
# 4 4.6 3.1 1.5 0.2 setosa
# 5 5.0 3.6 1.4 0.2 setosa
# 6 5.4 3.9 1.7 0.4 setosa
# 其中包含3个物种,每个物种包含50个样本
table(iris$Species)
# setosa versicolor virginica
# 50 50 50
# 首先绘制散点图
p <-
# 指定数据集为iris,x轴为Sepal.Length,y轴为Sepal.Width,同时用颜色表示不同的物种
ggplot(iris, aes(Sepal.Length, Sepal.Width, color = Species)) +
# 添加散点图层,设置点的大小为4
geom_point(size = 4) +
# 添加图表标签
labs(
title = 'Scatterplot with Edge Distribution', # 标题,自己给图片起个名!
subtitle = 'IRIS: Sepal.Length vs Sepal.Width', # 副标题,展示两个变量是什么!
caption = 'source: IRIS' # 节标题,这里我们展示了图表来源!
) +
# 设置颜色,不同颜色表示不同物种
scale_color_manual(values = c("#025259", "#d94f04", "#f29325")) +
# 设置主题为极简主题,并指定基础字体大小为14
theme_minimal(base_size = 14) +
# 设置绘图区域背景颜色为白色,并将图例放在底部,当然,咱想放哪放哪!随心所欲!
theme(plot.background = element_rect(fill='white', color='white'),
legend.position = 'bottom')
p
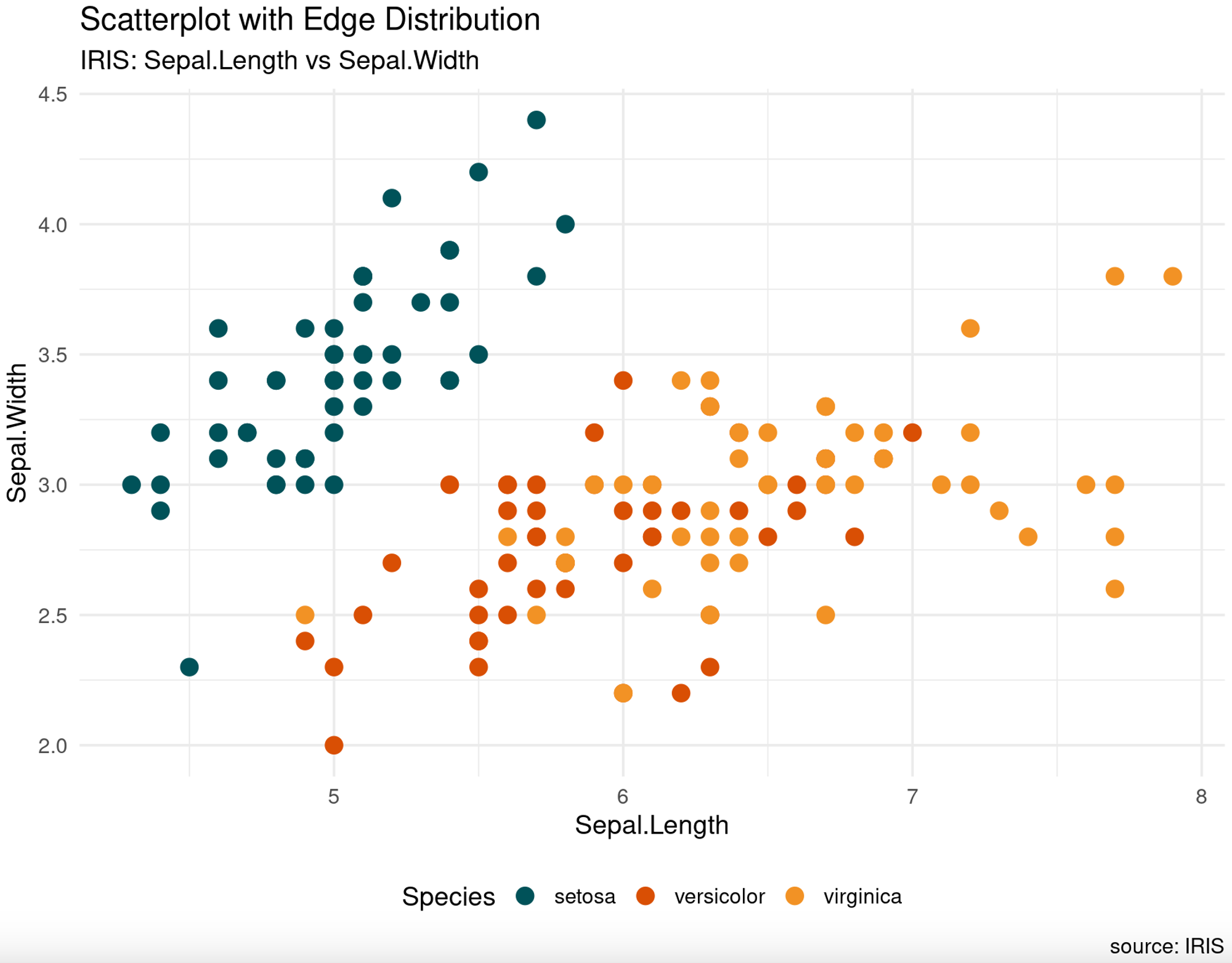
图形解读:
- 散点分布: 散点图展示了Sepal.Length和Sepal.Width两个变量之间的关系。每个点代表iris数据集中的一个观测值,横轴是Sepal.Length,纵轴是Sepal.Width。通过观察散点的分布,可以初步判断这两个变量之间是否存在某种趋势或者模式。
- 颜色区分: 每个点通过颜色进行分类,颜色表示不同的物种。通过
scale_color_manual函数设置了颜色映射,不同的颜色分别代表不同的物种。这样的颜色映射可以帮助我们直观地辨认出每个点所属的类别。 - 点的大小: 所有的点都使用了相同的大小,这可能是为了确保所有点在图中都有相似的可见性。点的大小通常可以用来突出重要的观测值或者显示数据的某些特性。
添加边缘分布图形
# 添加边缘分布图形
# 使用ggMarginal在散点图p上添加边缘分布图形
# 加载包,没安装的记得装一下哟!
library(ggExtra)
# 添加直方图类型的边缘分布图
p1 <- ggMarginal(p, type = "histogram", groupColour = TRUE, groupFill = TRUE, alpha = 0.7)
p1
# 函数解读:
# - `ggMarginal`: 用于在原始图上添加边缘分布图形的函数。
# - `type = "histogram"`: 指定了边缘分布图的类型为直方图。
# - `type = "density"`: 指定了边缘分布图的类型为核密度估计图。
# - `type = "boxplot"`: 指定了边缘分布图的类型为箱线图。
# - `type = "densigram"`: 指定了边缘分布图的类型为联合密度图。
# - `groupColour = TRUE`: 设置边缘分布图中的颜色与散点图中的颜色一致
# - `groupFill = TRUE`: 设置边缘分布图中的颜色与散点图中的颜色一致,并且填充颜色
# - `alpha = 0.7`: 设置图形的透明度,增加图形的可读性。
# 添加核密度估计图类型的边缘分布图
p2 <- ggMarginal(p, type = "density", groupColour = TRUE, groupFill = TRUE, alpha = 0.7)
p2
# 添加箱线图类型的边缘分布图
p3 <- ggMarginal(p, type = "boxplot", groupColour = TRUE, groupFill = TRUE, alpha = 0.7)
p3
# 添加联合密度图类型的边缘分布图
p4 <- ggMarginal(p, type = "densigram", groupColour = TRUE, groupFill = TRUE, alpha = 0.7)
p4
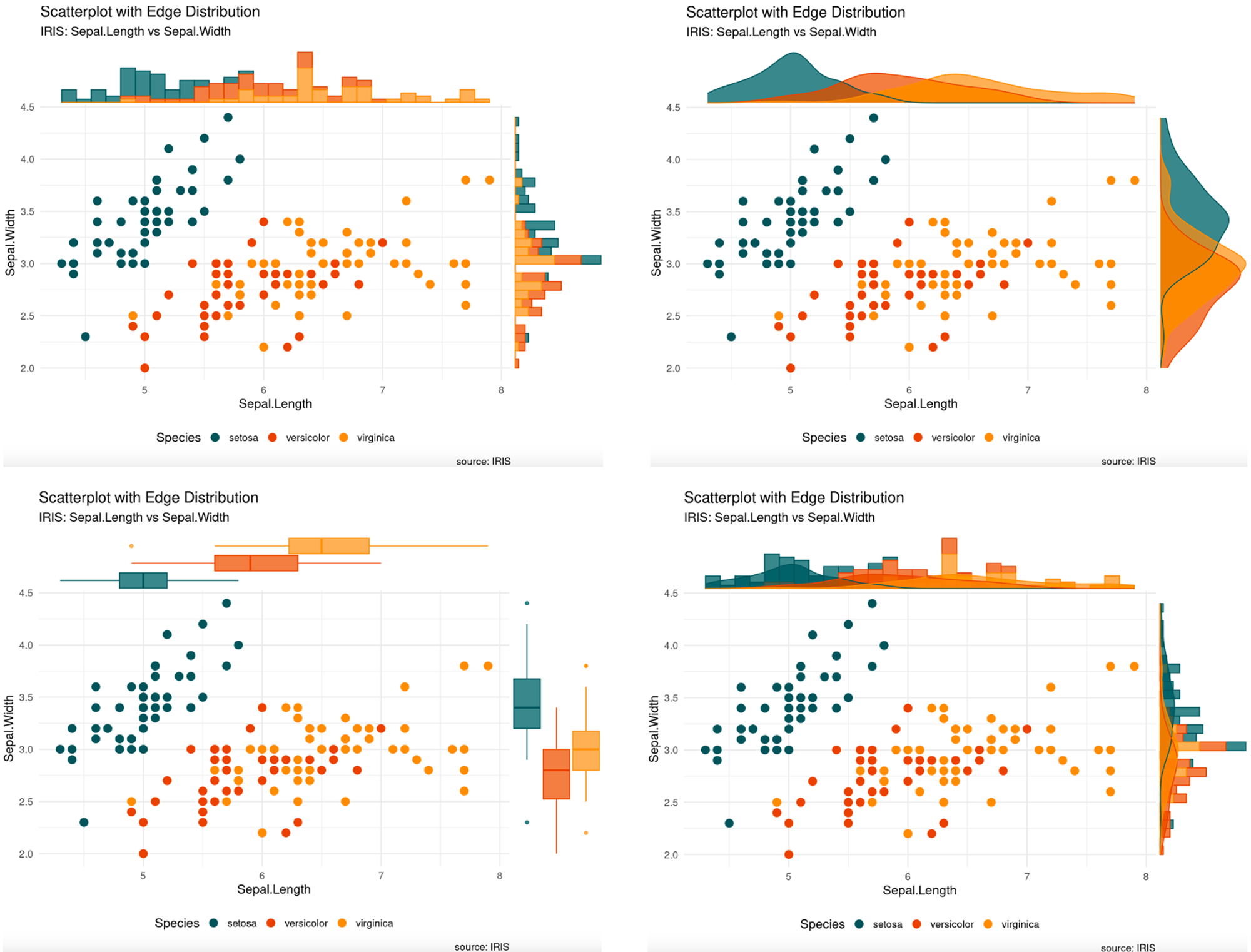
漂亮!!!
添加拟合线
如果你关注的是两个变量之间的相关性,比如两个基因表达水平之间或者某基因与疾病状态之间,这样就需要在散点图中添加拟合线,使用stat_smooth(method = lm, level = 0.99)即可实现。
添加拟合线时要注意是否需要不同组别(我们所用的数据中指不同物种)分别进行拟合,我们这里给大家演示一下。
# 添加拟合线
p <-
# 指定数据集为iris,x轴为Sepal.Length,y轴为Sepal.Width,同时用颜色表示不同的物种
ggplot(iris, aes(Sepal.Length, Sepal.Width, color = Species)) +
# 添加散点图层,设置点的大小为4
geom_point(size = 4) +
# 添加拟合线,
stat_smooth(method = lm, level = 0.99) +
# `method = lm`: 设置平滑曲线的拟合方法为线性回归,我们也可以选择其他方法!
# `se = F`: 设置不显示平滑曲线的标准误差带。
# `level = 0.99`: 当采用线性回归方法拟合平滑曲线时,`level` 参数表示置信区间的水平。
# 添加图表标签
labs(
title = 'Scatterplot with Edge Distribution', # 标题,自己给图片起个名!
subtitle = 'IRIS: Sepal.Length vs Sepal.Width', # 副标题,展示两个变量是什么!
caption = 'source: IRIS' # 节标题,这里我们展示了图表来源!
) +
# 设置颜色,不同颜色表示不同物种
scale_color_manual(values = c("#025259", "#d94f04", "#f29325")) +
# 设置主题为极简主题,并指定基础字体大小为14
theme_minimal(base_size = 14) +
# 设置绘图区域背景颜色为白色,并将图例放在底部
theme(plot.background = element_rect(fill='white', color='white'),
legend.position = 'bottom')
p
p4 <- ggMarginal(p, type = "densigram", groupColour = TRUE, groupFill = TRUE, alpha = 0.7)
p4
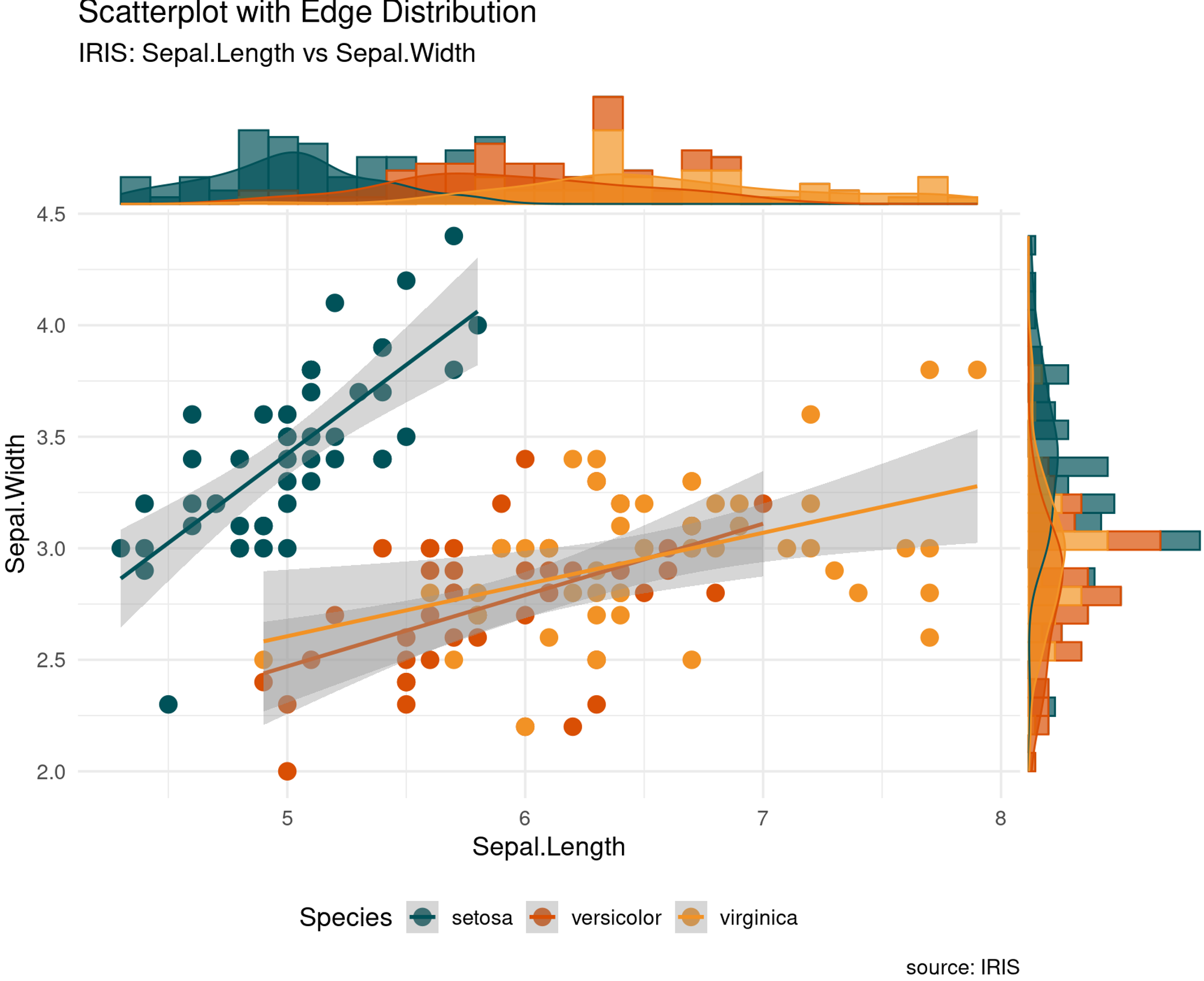
哒哒哒!每个组别都拟合就会是这样子!所以如果你只有一个组,那图图肯定就很简单明了!快去试试吧!
但其实,还有一种更简单的方法!一个函数就能搞定!
为了方便大家复现,我们还是用iris数据集进行演示,大家可以将Sepal.Length和Sepal.Width两个变量想象为你关注的两个基因或你关注的某基因与可能相关的疾病。
# 加载包,没安装的记得装一下哟!
library(ggstatsplot)
# 使用ggscatterstats创建带有边缘分布图的散点图
ggscatterstats(
data = iris, # 使用iris数据集
x = Sepal.Length, # 设置x轴为Sepal.Length列的数据
y = Sepal.Width, # 设置y轴为Sepal.Width列的数据
xlab = "Sepal Length", # 设置x轴标签为"Sepal Length"
ylab = "Sepal Width", # 设置y轴标签为"Sepal Width"
marginal = TRUE, # 在图上添加边缘分布图
marginal.type = "histogram", # 边缘分布图的类型为直方图
centrality.para = "mean", # 在边缘分布图上显示中心趋势,使用均值
margins = "both", # 显示上下和左右两侧的边缘分布图
title = "Relationship between Sepal Length and Sepal Width", # 设置整个图的标题
messages = FALSE, # 禁用输出关于平均值和置信区间的消息
)

哇!是不是你想要的嘞!
那今天的分享就到这里啦!我们下期再见哟!
最后顺便给自己推荐一下嘿嘿嘿!
如果我的分享对你有用的话,欢迎关注点赞在看转发分享阿巴阿巴阿巴阿巴巴巴!这可是我的第一原动力!
蟹蟹你们的喜欢和支持!!!
啊对!如果小伙伴们有需求的话,也可以加入我们的交流群:一定要知道 | 我们的生信交流群终于来啦!
还有兴趣的话,也可以看看我掏心掏肺的干货满满 | 给生信小白的入门小建议 | 掏心掏肺版!绝对干货满满!
后续这个链接可能会更新,如果小伙伴点开它之后发现,咦,怎么失效啦!不要慌!咱们辛苦一下动动小手去公众号主页的作者精选那里,会有一篇同名的文章,点进去就是啦!
参考资料
- https://zhuanlan.zhihu.com/p/654728525
- https://zhuanlan.zhihu.com/p/627088942
![[Java 并发基础]多线程编程](https://img-blog.csdnimg.cn/img_convert/70042d40e2f78a901ae6c14c8c751615.webp?x-oss-process=image/format,png)