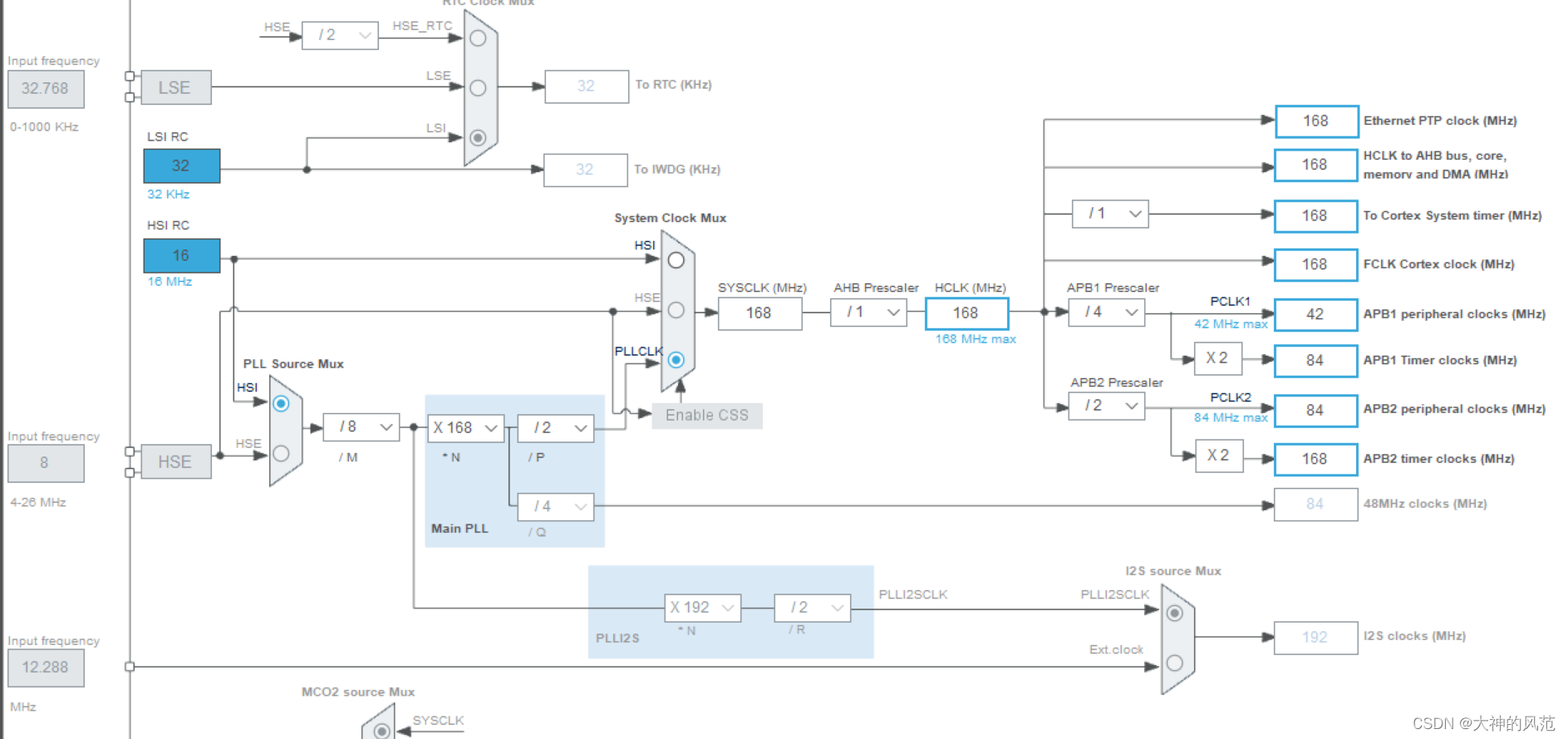
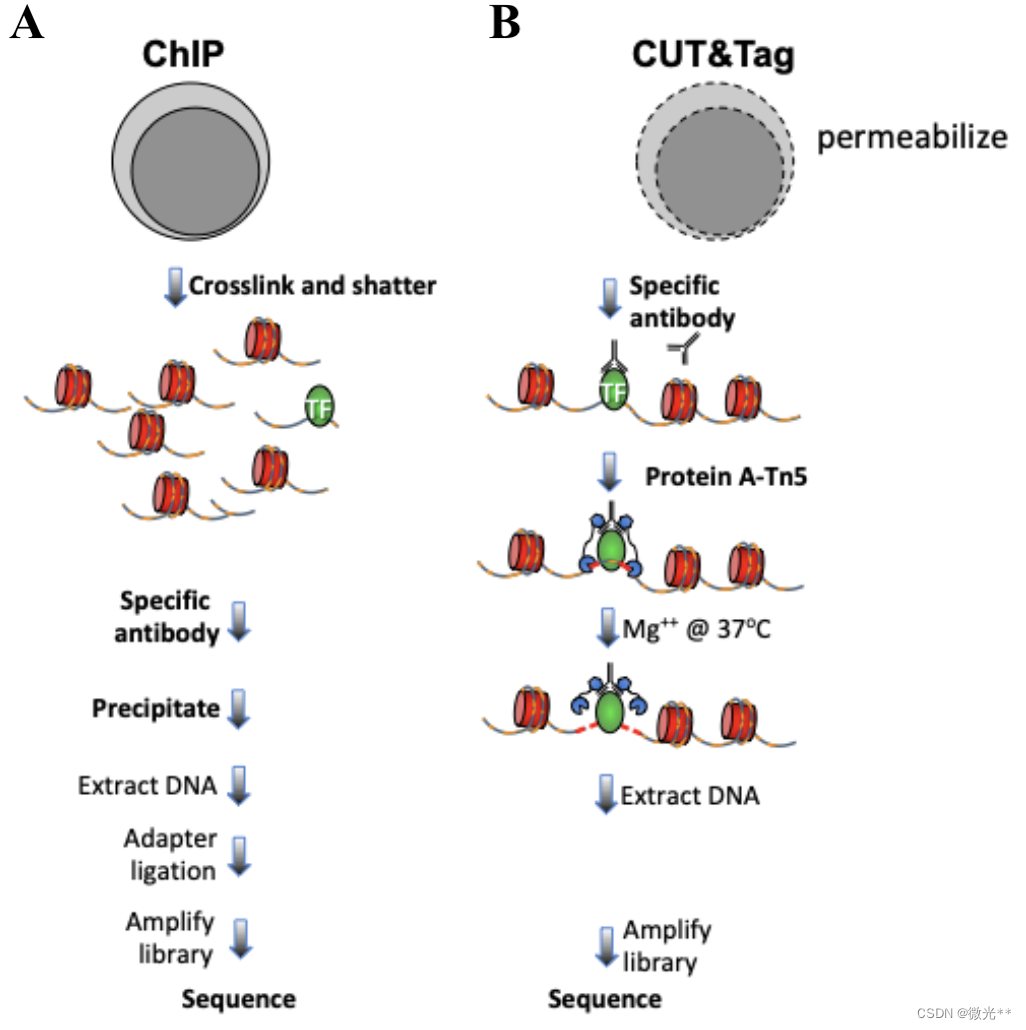
Cut&Tag(Cleavage Under Targets and Tagmentation)和ChIP-Seq(Chromatin Immunoprecipitation Sequencing)都是用于研究蛋白质与DNA相互作用的生物技术。它们在技术原理和应用方面有一些关键的区别。
1.ChIP-Seq测序
1.1 基本原理
利用特异性抗体免疫沉淀(immunoprecipitation,IP)蛋白质-DNA复合物,然后使用DNA酶切割,对沉淀的DNA片段进行纯化和测序。这个过程涉及到对固定(cross-linking)的细胞样本的处理,其中蛋白质和DNA通过形成共价键被固定在一起。
1.2 操作步骤
步骤包括细胞固定、染色质剪切、免疫沉淀、DNA提取、文库构建和测序。它是一个相对复杂且耗时的过程。
- 染色质交联(使用形式醛或其他交联剂)。
- 抗体免疫沉淀。
- 逆交联,提取DNA。
- 制备测序文库。
1.3 特点
通常需要较大数量的输入材料(成千上万的细胞),并且其灵敏度受限于抗体的亲和力和特异性。广泛用于研究组蛋白修饰、转录因子和其他DNA结合蛋白的基因组范围内的结合模式。通常成本较高,需要更多的试剂和更长的时间。
- 适用于研究广泛类型的蛋白-DNA相互作用。
- 需要相对较多的起始材料。
- 比较成熟和广泛使用的技术。
2.Cut&Tag测序
2.1 基本原理
使用特异性抗体来靶向蛋白质-DNA复合物,但是在细胞或细胞核的原位条件下使用一种叫做Tn5转座酶的酶直接在结合位点进行切割并插入测序接头(tagmentation)。这个过程不需要固定(cross-linking)样本。
2.2 操作步骤
操作更为简便,通常可以在一天内完成。这一技术减少了多个步骤,例如不需要染色质剪切和复杂的DNA纯化过程。
- 活细胞与特异性抗体孵育。
- Tn5转座酶介导的标记(Tagmentation)。
- 收集细胞,提取DNA。
- 直接制备测序文库。
2.3 特点
通常比ChIP-Seq更灵敏,可以使用更少的输入材料(甚至单细胞水平)。它能更精确地确定蛋白质结合位点,因为没有交联和解交联的步骤,这减少了背景噪声和假阳性。由于其高灵敏度和较少的输入材料要求,特别适用于稀有样本或单细胞层面的研究。由于步骤更简化,通常比ChIP-Seq更经济,需要的试剂和时间较少。
- 更适合于低丰度或单细胞分析。
- 无需交联和逆交联步骤,整个过程更快、更简单。
- 可能有更高的信号到噪声比。
总的来说,Cut&Tag是一种相对较新且高效的技术,适用于资源有限和需要高灵敏度的研究。而ChIP-Seq则是一种更传统且被广泛验证的技术,适用于大多数基因组范围的蛋白质-DNA交互研究。选择哪种技术取决于研究的具体目的、样本的类型和可用量,以及所需的数据质量和分辨率。