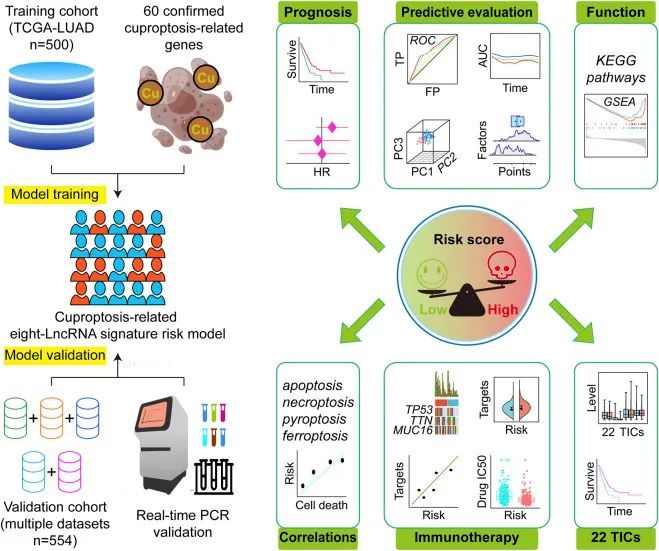
今天给同学们分享一篇生信文章“A novel defined risk signature of cuproptosis-related long non-coding RNA for predicting prognosis, immune infiltration, and immunotherapy response in lung adenocarcinoma”,这篇文章发表在Front Pharmacol期刊上,影响因子为5.6。

结果解读:
患者特征
本研究共纳入了1054例具有可用生存数据的LUAD病例。本研究的流程图如图1所示。作者从TCGA项目中纳入了500名患者用于风险模型的构建。作者从GEO下载了五个验证数据集(GSE29013、GSE30219、GSE31210、GSE37745和GSE50081)。根据作者的选择标准,这五个GEO数据集中包含了554个可用的LUAD信息,用于验证作者构建的基因特征。本研究使用的患者信息列于表1中。作者将五个验证数据集合并为一个队列,以更好地进行研究。批次效应是两个或多个数据集之间的非生物差异。作者根据之前提到的方法(图2A、B)消除了批次效应引起的偏差,并使转录谱更加相似。
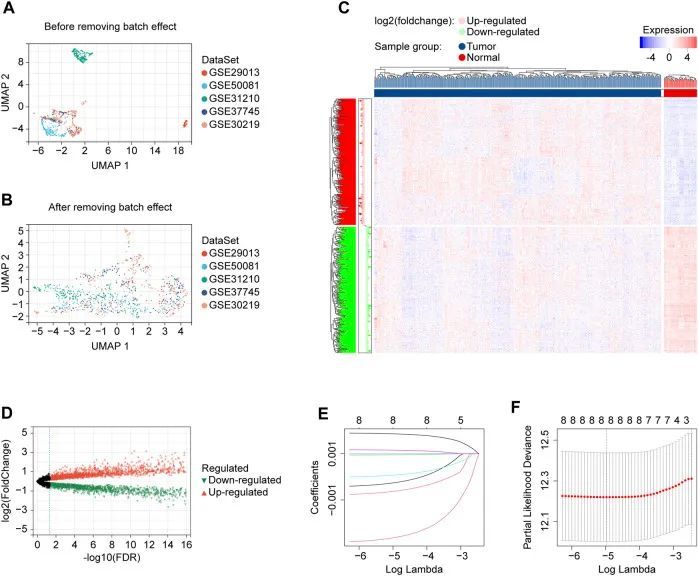
生成的与死亡相关的八个长链非编码RNA标记
根据作者的标准,作者得到了60个与铜死亡相关的基因,如表2所示。作者使用Pearson算法在训练队列样本中检测与铜死亡相关基因显著相关的lncRNA,最终确定了8277个独特的lncRNA。从这8277个lncRNA中,"limma" R语言包确定了其中3261个在正常和肿瘤组织中表达差异显著(图2C、D)。然后,作者使用这3261个lncRNA在训练队列中的表达谱来评估具有预后能力的候选基因。鉴定出9个具有潜在预后能力的lncRNA。通过使用"glmnet" R包(版本4.1.2,新西兰),进行LASSO Cox回归分析,缩小了申请基因的范围并开发了一个预测模型。最终,最佳lambda值选择为0.006898922,保留了8个lncRNA及其系数(图2E、F)。此外,作者展示了这8个lncRNA与这60个与铜死亡相关的基因之间的相关性。
已证实具有稳定预后能力的八个lncRNA基因特征
上部分显示了按风险评分递增排序的患者,中间的散点图显示了LUAD的生存状态,下部分的热图显示了八个关键lncRNA的相对表达水平。根据作者开发的风险评分计算器,为每个LUAD病例计算了一个风险评分。Kaplan-Meier估计器表明,在TCGA队列(log-rank检验,p < 0.001,图3A)和验证队列(log-rank检验,p < 0.001,图3B)中,高风险患者的生存率较低于低风险患者。此外,肿瘤分期亚型分析表明,作者的风险评分可以准确预测患者的预后,其中较高的风险评分预示着较差的疾病结局(图3A、B)。作者使用两个队列的数据以Kaplan-Meier曲线的形式展示了每个八个lncRNA的预后能力,结果显示MIR4435-2HG和NKILA对LUAD患者有稳定的不利影响,而TSPOAP1-AS1、AC107464.3、AC006449.7、LINC00324、COLCA1和HAGLR有助于LUAD的预后改善。
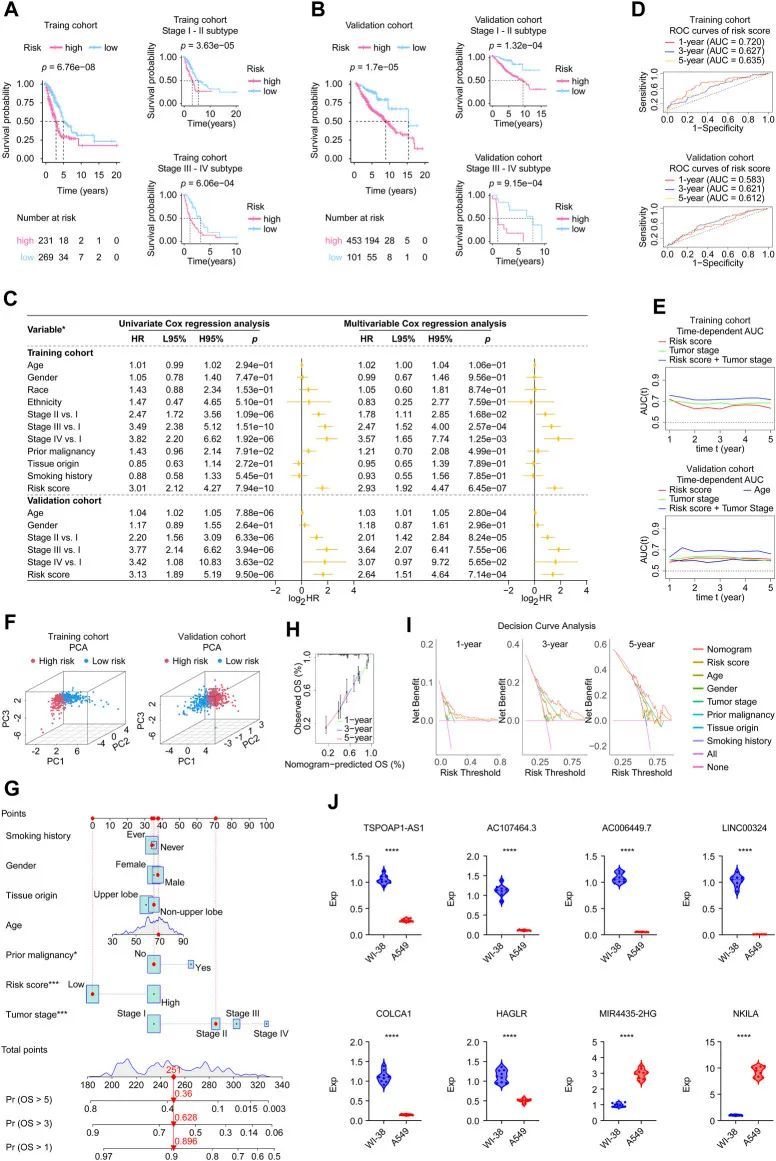
通过实时聚合酶链反应验证了基因特征中每个基因的表达水平
为了更好地了解基因基因特征中每个基因在真实世界中的表达模式,作者应用实时聚合酶链反应(real-time PCR)检测了人类LUAD细胞系(n = 9)和人类正常细胞系(n = 9)中上述基因的表达差异。表3显示了8个基因的引物序列,分别为TSPOAP1-AS1、AC107464.3、AC006449.7、LINC00324、COLCA1、HAGLR、MIR4435-2HG和NKILA。值得注意的是,所有基因在肿瘤和正常细胞系中的表达均存在差异(图3J)。具体而言,MIR4435-2HG和NKILA基因在LUAD细胞系中高度表达,而其余基因在LUAD细胞系中表达不足。LUAD中上调的基因(如MIR4435-2HG和NKILA),而LUAD中下调的基因(如TSPOAP1-AS1、AC107464.3、AC006449.7、LINC00324、COLCA1和HAGLR)则显示出对LUAD结果具有保护功能,这进一步证明了作者发现的基因基因特征的有效性,并为深入研究提供了线索。
GSEA确定了预后标志的机制
基于风险评分进行的GSEA分析发现,lncRNA基因特征中存在显著富集的基因集,其中前十个项目主要与移植物排斥、脂肪酸代谢、肌醇磷酸代谢、花生四烯酸代谢、PPAR信号通路、肠道免疫网络对IgA产生、血管平滑肌收缩、α-亚麻酸代谢和非同源末端连接相关(图4)。

八个lncRNA基因特征与凋亡、坏死程序性细胞死亡、火凤凰病和铁死亡的关系
根据作者的标准,作者找到了凋亡、坏死程序化、火凤凰和铁死亡基因。皮尔逊系数分析了作者的预后模型与凋亡、坏死程序化、火凤凰和铁死亡相关基因之间的关系。分析结果显示,SELENBP1、METTL7A、ANLN、SARM1、GGA2、GAPDH、MOAP1、CTSH、RBP5和CEP55是与凋亡相关的前十个相关基因,总体上,2321/3681(63.05%)个基因与长链非编码RNA特征显著相关。RIPK3、FADD、TRPM7、CYLD、TLR3、IPMK、ZBP1、TNF、FASLG和PGLYRP1被发现是与八个长链非编码RNA特征显著相关的前十个坏死程序化相关基因。总体而言,11/20(55.00%)个坏死程序化相关基因与该特征显著相关。此外,皮尔逊检验发现与作者的特征显著相关的前十个火凤凰相关基因是NLRP1、CARD8、GSDMB、CYCS、IRF2、GSDMD、IL1A、NLRP6、CASP1和CHMP2B。总体而言,34/50(68.00%)个火凤凰相关基因与作者的特征显著相关。检查发现SLC2A1、RRM2、AURKA、MDM4、PLA2G6、DUOX1、GLS2、NRAS、HIF1A和SIRT3是与作者的标志相关的顶级铁死亡相关基因。总结起来,222/380(58.42%)与作者的标志相关的铁死亡相关基因。
风险评分在免疫疗法中的作用
关于相关的免疫功能,CCR、检查点、细胞溶解活性、HLA、促炎、T细胞共抑制、T细胞共刺激和II型干扰素反应的评分在高风险组中低于低风险组(图5A)。这些发现表明,这八个长链非编码RNA的特征可能与LUAD的免疫状态有关。作者全面探索了TCGA-LUAD队列中所有肿瘤样本的突变特征。如图5B所示,前20个最常突变的基因中,TP53基因突变最频繁,约占队列的53.3%,其次是TTN(50.6%)和MUC16(43.8%)。在这些突变中,错义突变是最常见的变异类型。有趣的是,作者注意到这20个基因在高风险组和低风险组的突变分布在统计学上具有显著差异(p < 0.05)。
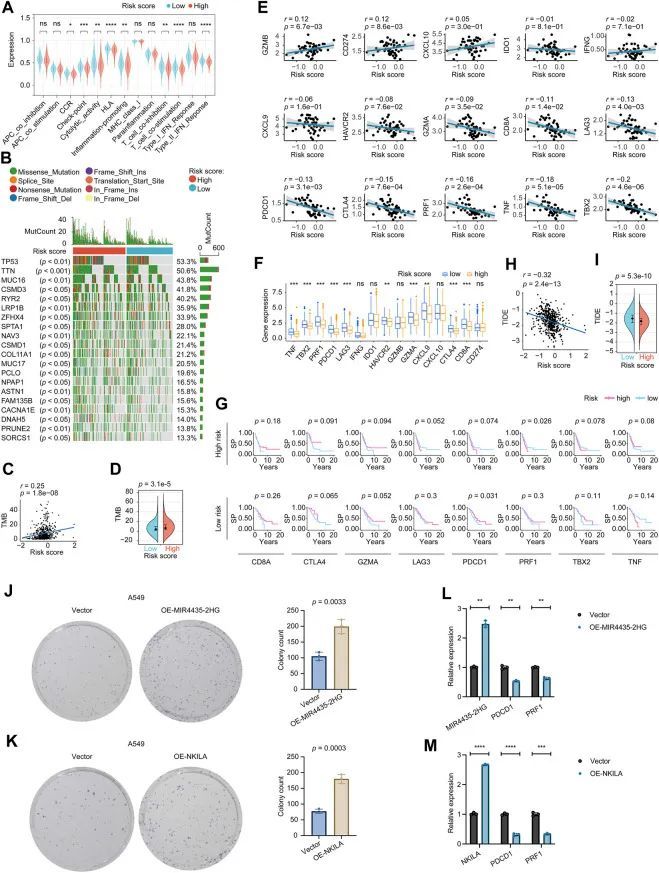
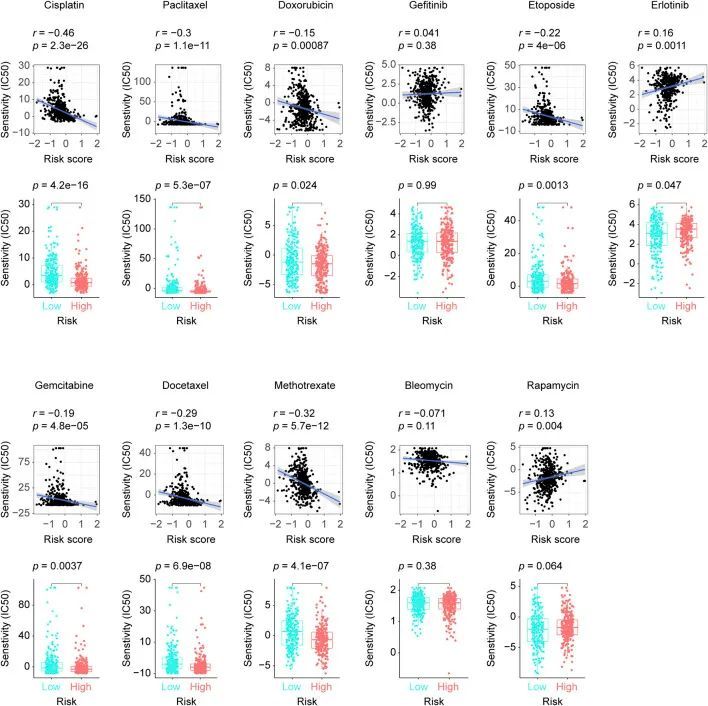
风险评分和抗癌药物敏感性
作者从GDSC数据库中筛选出了8种化疗药物(顺铂、紫杉醇、多柔比星、依托泊苷、吉西他滨、多西他赛、甲氨蝶呤和博来霉素),2种表皮生长因子受体(EGFR)抑制剂(吉非替尼、厄洛替尼)和1种免疫抑制剂(雷帕霉素),这些药物在LUAD临床实践中被使用。通过采用“pRRophetic”软件包,作者发现7种化疗药物(顺铂、紫杉醇、多柔比星、依托泊苷、吉西他滨、多西他赛和甲氨蝶呤)和1种EGFR抑制剂(厄洛替尼)在高风险和低风险患者中显示出显著的敏感性差异,而博来霉素、吉非替尼和雷帕霉素与作者的风险评分模型没有相关性。如图6所示,顺铂、紫杉醇、多柔比星、依托泊苷、吉西他滨、多西他赛和甲氨蝶呤在高风险患者中得到了更多的反馈,而厄洛替尼在低风险LUAD中得到了更强烈的反应,这暗示了八个长链非编码RNA标志在临床实践中的潜在用途。
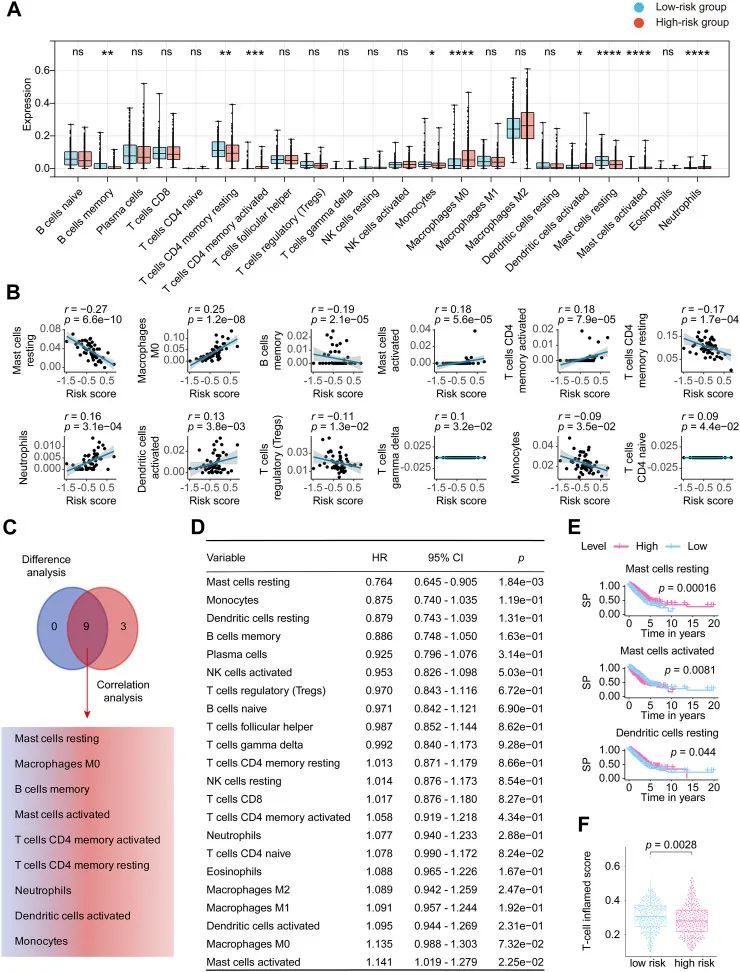
肥大细胞在八个长链非编码RNA标记的预测能力中的重要作用
如图7A所示,Wilcoxon秩和检验确定了与风险评分相关的9个TICs。皮尔逊系数(图7B)发现了与作者的标志密切相关的12个TICs。总结如下(图7C),共有9个TICs与lncRNA标志显著相关,包括静息型肥大细胞、M0型巨噬细胞、记忆型B细胞、活化型肥大细胞、激活型CD4记忆T细胞、静息型CD4记忆T细胞、中性粒细胞、激活型树突状细胞和单核细胞。具体而言,作者的标志与M0型巨噬细胞、活化型肥大细胞、激活型CD4记忆T细胞、中性粒细胞、激活型树突状细胞呈正相关,与其他细胞呈负相关。
总结
本研究构建了一种新的铜死亡相关的8 lncRNA LUAD预后标志。通过将基因特征应用于独立队列,验证了其稳定性和广泛适用性。该基因特征具有承担精确免疫治疗和抗癌药物选择作用的潜在能力。免疫浸润分析表明,肥大细胞可能有助于信号保持其预测能力。作者的工作可能会促进LUAD诊断和治疗的发展。