博客已同步微信公众号:GIS不要炒茄子

01 前言
最近打算认真从头开始学习深度学习和遥感结合的相关内容,主要通过Python进行处理。此前用深度学习进行遥感相关的学习一直都是用 tensorflow3.0 框架,但是考虑很多因素我后面打算换用 Pytorch 进行学习。好在tensorflow我只是浅尝辄止,不然还真有点不舍。
02 如何读取TIFF文件
2.1 安装GDAL
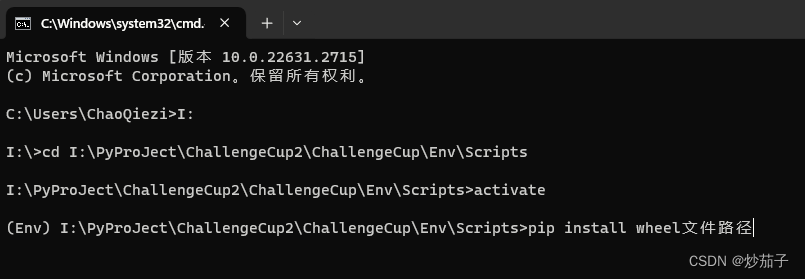
读取TIFF文件我们就用GDAL吧,安装稍麻烦。如果通过pip方式无法安装模块,可通过下方链接下载Wheel文件:
https://www.lfd.uci.edu/~gohlke/pythonlibs/#
下载后通过pip install wheel文件绝对路径即可安装。如果想要安装在虚拟环境,需要先进入对应虚拟环境路径activate再进行pip安装。
2.2 读取TIFF文件
这个就仅仅给出代码吧,其它时间有限自行理解。
一个波段一个波段的读取:
def read_img(img_path):
"""
该函数用于读取TIFF文件中各个波段数据集
:param img_path: TIFF文件路径
:return: 多波段数据集(ndarray, [波段数, 行数, 列数])
"""
# 打开文件
ds = gdal.Open(img_path)
if not ds:
return False
# 读取
bands = []
for band_ix in range(1, ds.RasterCount + 1):
band = ds.GetRasterBand(band_ix)
band_data = band.ReadAsArray()
bands.append(band_data)
bands = np.asarray(bands)
return bands
当然,一下全部读取也可以:
def read_img(img_path):
"""
该函数用于读取TIFF文件中各个波段数据集
:param img_path: TIFF文件路径
:return: 多波段数据集(ndarray, [波段数, 行数, 列数])
"""
# 打开文件
ds = gdal.Open(img_path)
if not ds:
return False
bands = ds.ReadAsArray()
return bands
03 如何切片?
切片你可以用numpy的 from numpy.lib.stride_tricks import as_strided 自己写(谨慎使用该函数),当然或者完全自己写。但是运行的效率自然无法保证。
这里我们用 skimage 模块的 view_as_windows 函数进行切片,不仅支持高维度切片并且效率非常高。
import numpy as np
from skimage.util import view_as_windows
def make_chips(ds, window_shape, stride, stacking=False):
"""
对数据集进行切片处理
:param ds:
:param window_shape: 滑动窗口大小
:param stride: 步幅大小
:return: 返回切片后的数据
"""
chips = view_as_windows(ds, window_shape, step=stride)
chips = np.squeeze(chips) # 去除维度上数值为1的所有维度
if stacking:
chips = np.reshape(chips.shape[0] * chips.shape[1], -1)
return chips
使用:
img_paths = glob.glob(os.path.join(in_dir, '**', 'GF2*MSS*.tiff'), recursive=True)
for img_path in img_paths:
# 读取
data = read_img(img_path)
# img_transform = get_transform(img_path)
# 切片
chips = make_chips(data, (4, 256, 256), 256, False)
data的shape为(4, 6908, 7300),我需要切片为(4,256, 256),那么滑动窗口大小设置为(4, 256, 256),窗口在各个维度上移动的步幅大小这里设置为256(当然你也可以设置为(4, 256,256),即具体指定各个维度上移动的步幅)。