挖掘GEO公共单细胞数据集时,会遇到常见各种单细胞测序数据格式。现总结如下,方便自己日后调用,以创建Seurat对象
(1)barcodes.tsv.gz、features.tsv.gz、matrix.mtx.gz
(2)表达矩阵
(3)h5
(4)h5ad
格式一:barcodes.tsv.gz、features.tsv.gz、matrix.mtx.gz【☆】
- 这是cellranger上游比对分析产生的3个文件,分别代表细胞标签(barcode)、基因ID(feature)、表达数据(matrix)
- 一般先使用
read10X()对这三个文件进行整合,得到行为基因、列为细胞的表达矩阵(为稀疏矩阵dgCMatrix格式,节约内存);然后再配合CreateSeuratObject()函数创建Seurat对象 - 示例数据集:GSE166635,创建代码如下----

dir="./data/HCC2/filtered_feature_bc_matrix/"
list.files(dir)
#[1] "barcodes.tsv.gz" "features.tsv.gz" "matrix.mtx.gz"
counts <- Read10X(data.dir = dir)
class(counts)
#[1] "dgCMatrix"
#attr(,"package")
#[1] "Matrix"
scRNA <- CreateSeuratObject(counts = counts)
scRNA
#An object of class Seurat
#33694 features across 9112 samples within 1 assay
#Active assay: RNA (33694 features, 0 variable features)
- 如上
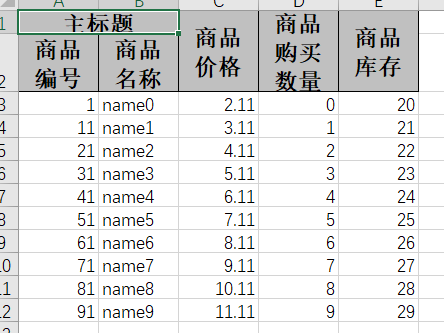
Read10X()函数接受的参数为目录名,该目录包含了所需的三个配套文件;值得注意的是三个文件名只能分别是barcodes.tsv.gz、features.tsv.gz、matrix.mtx.gz,然后read10X函数可以自动加载。如上截图那样就是需要修改的~
格式二:直接提供表达矩阵,使用seurat读取
- 这种是最方便的,直接创建Seurat即可
- 示例数据:GSE144320

格式2.1:直接提供表达矩阵,使用seurat读取
对于GSE104154这个数据集,比较费工夫,需要duplicated去重

library(dplyr)
#1 读取rawdata-----
raw_counts=read.csv("~/ipf/GSE104154_scRNA-seq_fibrotic MC_bleomycin/GSE104154_d0_d21_sma_tm_Expr_raw/GSE104154_d0_d21_sma_tm_Expr_raw.csv")
head(raw_counts)[1:4,1:4]
table(raw_counts$symbol) %>%head()
head(raw_counts)[1:4,1:4]
#1.2 去重复----
tmp=raw_counts[!duplicated(raw_counts$symbol) ,]
head(tmp)[1:4,1:4]
rownames(tmp)=tmp$symbol
head(tmp)[1:4,1:4]
#2 获取counts----
counts=tmp[,c(-1,-2)]
head(counts)[,1:9]
library(Seurat)
#https://zhuanlan.zhihu.com/p/385206713
#2 创建seruat对象------
rawdata=CreateSeuratObject(counts = counts,project = "blem",assay = "RNA")
hp_sce=rawdata
hp_sce@assays$RNA@counts[1:5,1:6]
格式三:h5格式文件
- 使用
Read10X_h5()函数,读入表达矩阵,在创建Seurat对象 - 示例数据:GSE138433

sce <- Read10X_h5(filename = GSM4107899_LH16.3814_raw_gene_bc_matrices_h5.h5")
sce <- CreateSeuratObject(counts = sce)
格式四:h5ad格式
- 需要安装,使用
SeuratDisk包的两个函数; - 先将后
h5ad格式转换为h5seurat格式,再使用LoadH5Seurat()函数读取Seurat对象。 - 示例数据集:GSE153643
#remotes::install_github("mojaveazure/seurat-disk")
library(SeuratDisk)
Convert("GSE153643_RAW/GSM4648565_liver_raw_counts.h5ad", "h5seurat",
overwrite = TRUE,assay = "RNA")
scRNA <- LoadH5Seurat("GSE153643_RAW/GSM4648565_liver_raw_counts.h5seurat")
#注意一下,我之前载入时,表达矩阵被转置了,需要处理一下~

以上是我目前了解到的针对不同数据来源,创建Seurat对象的几种方式。如遇新的方法,会继续补充~~