真核生物基因包含一系列外显子和内含子,内含子必须在转录过程中被移除以便成熟的 mRNA 被翻译成蛋白质,RNA 剪接则是这一过程中至关重要的一步。RNA 剪接包含两类剪接事件。
组成型剪接 (constitutive splicing): RNA 剪接的一种基本方式,这种情况下每个转录单位只产生一种成熟的 mRNA。
选择性剪接 (alternative splicing): 一个 pre-mRNA 可形成多种成熟的 mRNA。可变剪接与疾病密切相关,最近的研究就发现 RNA 剪接在肿瘤细胞中发生频率远超过正常细胞,RNA 剪接过程及其调控可以促进癌症的发生和发展。
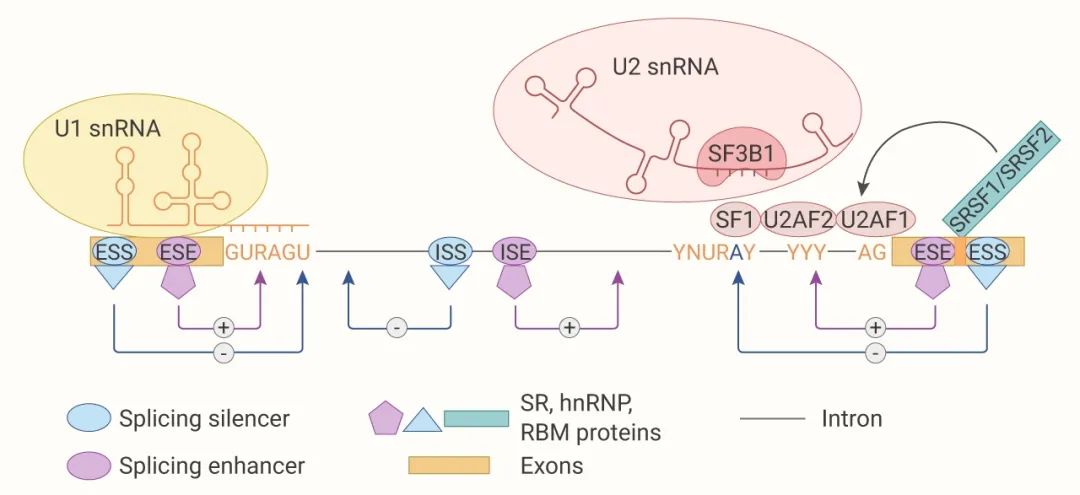
顺式作用元件:外显子剪接增强子 (ESE),内含子剪接增强子 (ISE),外显子剪接沉默子 (ESS) 或内含子剪接沉默子 (ISS)。反式作用元件:SR 蛋白家族和 hnRNPs,同时也有 ESRP1/2、MBNL1/2/3 等其他因子。
RNA 剪接是个高度受控的过程,除了剪接位点和剪接体还有很多剪接调控元件 (SRE) 参与了剪接调控。顺式作用元件招募剪接因子,以促进或抑制附近剪接位点的识别。反式作用元件可以结合至 pre-mRNA 上,调控剪接体的组装和剪接位点的识别。
有研究发现,肿瘤细胞中大量的 RNA 剪接可能产生肿瘤新抗原。Intron retention is a source of neoepitopes in cancer 一文表明:由不完全 RNA 剪接产生的内含子保留翻译的多肽在肿瘤细胞系中可由人主要组织相容性复合物 I (MHC I) 呈现。然而,剪接衍生的多肽是否会引起内源性免疫应答仍是未知的。
Abdel-Wahab 等研究者在 Cell 的前沿研究“Pharmacologic modulation of RNA splicing enhances anti-tumor immunity” 解答了这一问题,他们证实了在 RNA 剪接调节药物的干扰下,肿瘤细胞产生了新抗原,并且这些抗原通过 MHC I 作为新抗原表位呈现,从而激发抗肿瘤免疫。
药物剪接以依赖宿主 T 细胞和肿瘤 MHC i 肽的方式抑制体内肿瘤生长
RBM39 是一种 RNA 结合蛋白,参与转录协同调控和选择性 RNA 剪接。研究人员使用 Indisulam (剪接因子 RBM39 的降解剂) 处理多种肿瘤细胞系后,发现 RBM39 降解对肿瘤细胞的体外生长影响不大 (图 2C)。但是,把这些 Indisulam 处理过肿瘤细胞移植到小鼠体内,肿瘤细胞的体内生长被显著性抑制 (图 2E)。
这种体外和体内生长的明显差异并非是肿瘤细胞的自主效应。研究人员推测他们观察到的现象与 RNA 剪接刺激了抗肿瘤免疫反应有关。
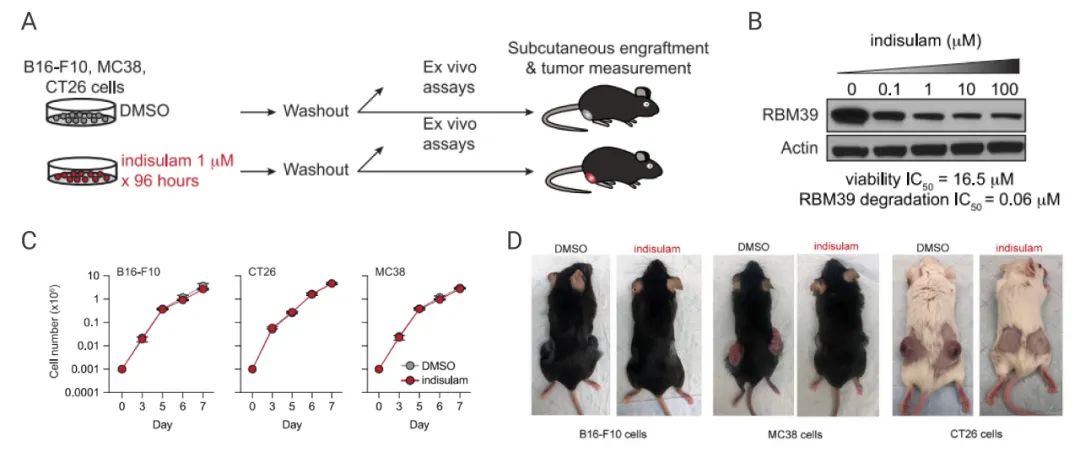
A. 药物处理与洗脱图示;B. B16-F10 细胞中 RBM39 的表达检测;C. 肿瘤细胞的数量变化;D. 来自于 C 中处理的细胞植入小鼠体内。
接下来研究者们验证剪接调节是否通过药物诱导的新抗原产生增强肿瘤免疫识别。
研究者们比较了载有药物治疗肿瘤裂解物的专业抗原提呈细胞 (APCs) 刺激 T 细胞的能力。MC38 细胞用 DMSO、indisulam 或 MS-023 处理,用于生成含有潜在免疫原性肽(MHC I 肽) 的裂解物。野生 C57BL/6 或 B2m 敲除小鼠来源的 BMDCs 被裂解物脉冲,与 CD45.1+ 脾 T 细胞孵育。与对照组相比,Indisulam 或 MS-023 处理的 BMDCs 更能促进 CD8+ T 细胞增殖 (图 3C 和 3D)。在 B2m KO BMDCs 中没有观察到这种效果,这表明 MHC I 肽呈递是必需的。
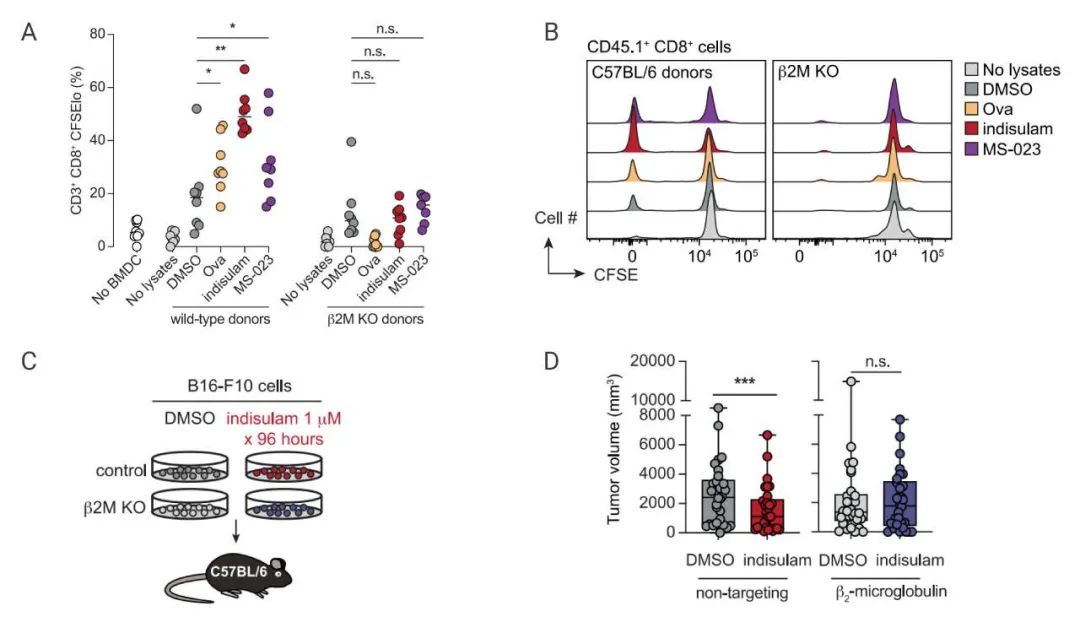
A. DMSO、鸡卵清蛋白(OVA)、Indisulam、或 MS-023 处理细胞,生成含潜在免疫原性肽的裂解物。B. 裂解物与 BMDCs 和 T 细胞共培养。C-D. 处理过的 B16-F10 细胞,植入小鼠体内,分析肿瘤体积的差异。
RNA 剪接调节增强抗肿瘤免疫
接下来,研究人员使用 B16-F10,MC38 和 LLC 肿瘤模型评估 Indisulam 和 PD1 抗体分别单独给药或联合使用对肿瘤生长的作用。
Indisulam 和 PD1 抗体联合使用可显著降低 B16-F10 和 MC38 肿瘤的生长,并且其效果超过了两种药物单独使用的效果 (图 4C)。这说明 Indisulam 和 PD1 抗体联合使用可以协同抑制肿瘤的生长。MS-023 和 PD1 抗体联合使用也具有协同抑制 B16-F10 和 MC38 肿瘤的生长的作用 (图 4D)。这说明,干扰 RNA 剪接和阻断免疫检查点共同促进了对肿瘤生长的抑制作用。
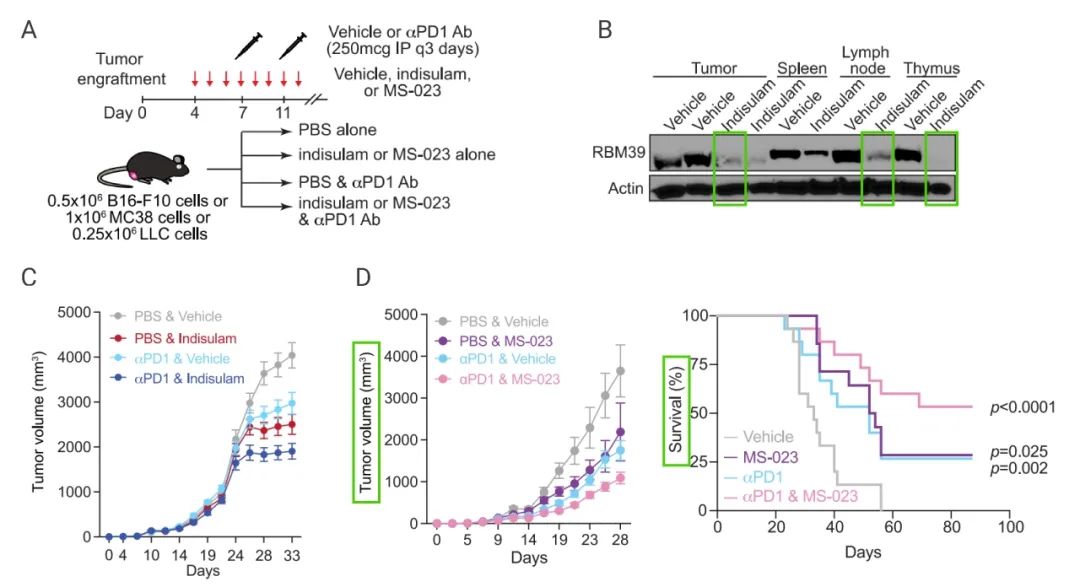
A. 多种肿瘤模型的治疗图示;B. Indisulam 治疗 10 天后各组织中 RBM39 的表达;C-D. Indisulam 和 PD1 抗体分别或联合使用治疗后肿瘤体积变化。
研究人员用转录组测序技术 (RNA-seq) 分析结果表明,Indisulam 处理 B16-F10 细胞后,RNA 内含子保留的覆盖度在细胞质部分十分明显 (图 5A),并且这些 mRNA 可以被翻译成潜在的新抗原 (图 5B)。这些结果表明,剪接调节能够诱导大量新抗原的表达,导致潜在的免疫抗原的产生。
研究人员还检测了所有 8-14 个氨基酸长度的多肽序列以此来分析 RNA 剪接调节药物处理后人和鼠肿瘤细胞系内新抗原的变化。
在人和小鼠细胞系中1763 种新抗原在两个物种中都有 (图 5C)。研究人员还纯化分离了与 H-2Kb 和 H-2Db (即 MHC-I 类分子) 结合的多肽 (图 5D),并创建了 RNA 亚型和蛋白质组数据库 (图 5E)。质谱检测结果分别发现了 366 种 (H-2Db) 和 518 种 (H-2Kb) 新抗原(即 MHC-I 类分子)仅存在于Indisulam 处理的样本中 (图 5F)。
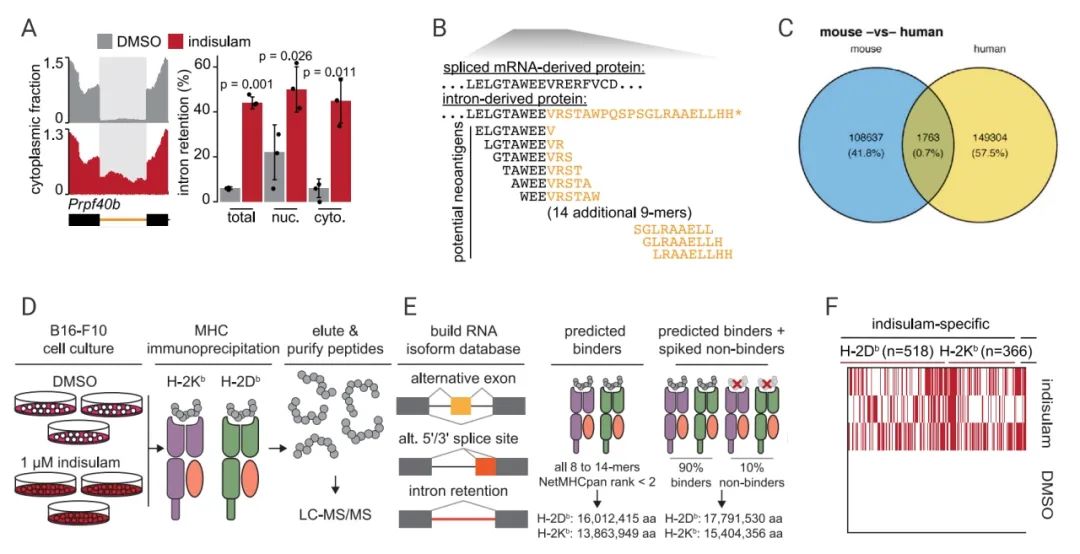
A. 细胞质部分 Prpf40b 内含子保留的 RNA 序列覆盖率;B. 由 Prp40b 内含子保留产生的多肽。C. Indisulam 处理的人和小鼠细胞系共有新抗原;D. RNA 亚型和蛋白质组数据库创建;F . H-2K b 和 H-2D b 结合物蛋白质组的热力图。
研究人员使用候选肽对小鼠进行免疫,检测引流淋巴结中 CD8+ T 细胞分泌 IFN-γ 以及杀死肿瘤细胞的能力 (图 6A,6D)。发现根据 RNA-seq 和质谱分析选取的 70 个候选肽中,有 30 个在小鼠中引发 CD8+ T 细胞免疫反应 (图 6B)。根据 RNA-seq 和 MHC I 结合预测选取的 39 个候选肽中的 11 个 引发 CD8+ T 细胞免疫反应 (图 6C)。
这些数据表明 RNA 剪接调节触发了特定的新抗原的产生,其水平足以驱动识别这些抗原的 CD8+ T 细胞的增殖。
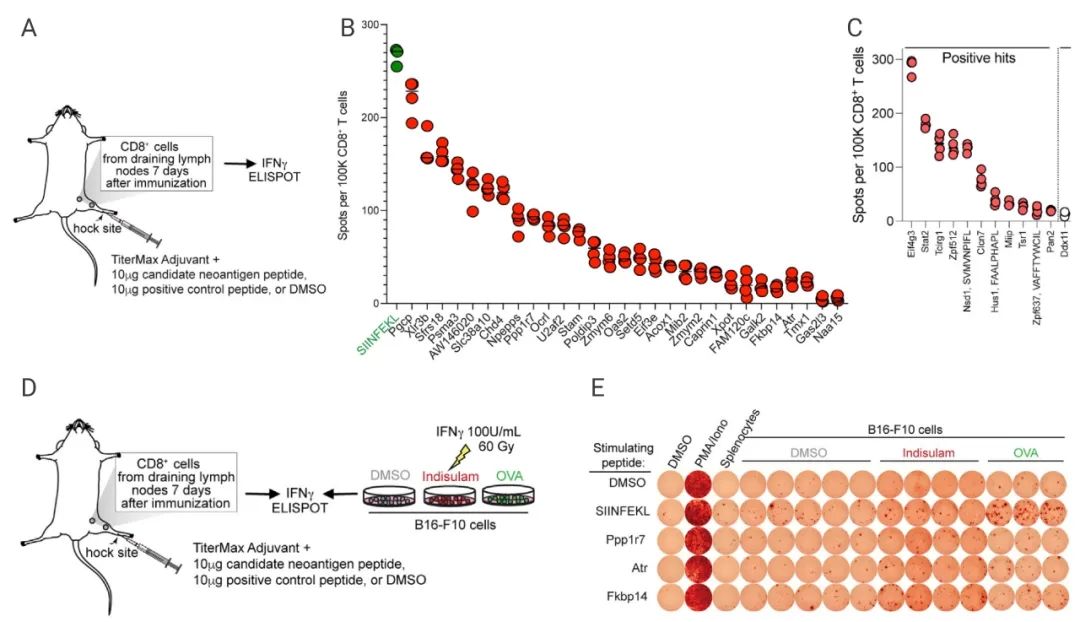
A-C. 小鼠 CD8 + T 细胞与负载候选肽的 B16-F10 细胞共培养,B16-F10 的细胞毒性研究 ; D-E. CD8 + T 细胞与 B16-F10 细胞共培养,检测 CD8 + T 细胞的 IFN-γ 酶联免疫斑点个数 。
总结:
当前研究最多的肿瘤抗原主要源于基因突变,而本研究确定了 RNA 剪接调节药物可以诱导肿瘤细胞产生潜在的抗原,并证明了剪接衍生的新抗原会引起内源性抗肿瘤免疫应答,具有增强免疫检查点抑制疗效的效力。这表明 RNA 剪接调节可作为肿瘤抗原的潜在来源,具有应用于肿瘤免疫检查点治疗的潜力。
| 相关产品 |
| Indisulam Indisulam (E 7070) 是一种具有抗癌活性的 RNA 剪接调节的药物,通过募集 DCAF15 来诱导 RBM39 降解。 |
| MS023 MS023 是一种人 I 型蛋白精氨酸甲基转移酶 (PRMTs) 抑制剂。 |
| Pladienolide B Pladienolide B 是一种癌细胞生长抑制剂,靶向剪接体的 SF3B1 亚基,通过抑制 pre-mRNA 剪接发挥抗肿瘤活性。 |
| Herboxidiene Herboxidiene (GEX1A) 是链霉菌 (Streptomyces sp. A7847) 的一种强植物毒性聚酮,通过结合剪接体相关蛋白 (SAP) 155 来抑制 pre-mRNA 的剪接过程。 |
| EPZ015666 EPZ015666 (GSK3235025) 是一具有口服活性的 PRMT5 抑制剂。 |
MCE 的所有产品仅用作科学研究或药证申报,我们不为任何个人用途提供产品和服务。
参考文献
1. Topalian SL, Taube JM, Anders RA, Pardoll DM. Mechanism-driven biomarkers to guide immune checkpoint blockade in cancer therapy. Nat Rev Cancer. 2016 May;16(5):275-87.
2. Supek F, Miñana B, Valcárcel J, Gabaldón T, Lehner B. Synonymous mutations frequently act as driver mutations in human cancers. Cell. 2014 Mar 13;156(6):1324-1335.
3. Jung H, Lee D, Lee J, Park D, Kim YJ, Park WY, Hong D, Park PJ, Lee E. Intron retention is a widespread mechanism of tumor-suppressor inactivation. Nat Genet. 2015 Nov;47(11):1242-8.
4. Jayasinghe RG, Cao S, Gao Q, Wendl MC, Vo NS, Reynolds SM, .et al. Systematic Analysis of Splice-Site-Creating Mutations in Cancer. Cell Rep. 2018 Apr 3;23(1):270-281.e3.
5. Kahles A, Lehmann KV, Toussaint NC, Hüser M, Stark SG, .et al. Comprehensive Analysis of Alternative Splicing Across Tumors from 8,705 Patients. Cancer Cell. 2018 Aug 13;34(2):211-224.e6.
6. Smart AC, Margolis CA, Pimentel H, He MX, Miao D, Adeegbe D, Fugmann T, Wong KK, Van Allen EM. Intron retention is a source of neoepitopes in cancer. Nat Biotechnol. 2018 Dec;36(11):1056-1058.
7. Bonnal SC, López-Oreja I, Valcárcel J. Roles and mechanisms of alternative splicing in cancer-implications for care. Nat Rev Clin Oncol. 2020 Aug;17(8):457-474.
8. Lu SX, De Neef E, Thomas JD, Sabio E,. Pharmacologic modulation of RNA splicing enhances anti-tumor immunity. Cell. 2021 Jul 22;184(15):4032-4047.e31.






![[附源码]java毕业设计酒店管理系统](https://img-blog.csdnimg.cn/07310cd38519468e8c4f7148edb20ed9.png)