论文标题:Mol-Instructions: A Large-Scale Biomolecular Instruction Dataset for Large Language Models
论文链接:
https://arxiv.org/pdf/2306.08018.pdf
Github链接:
https://github.com/zjunlp/Mol-Instructions
模型下载(wisemodel):
https://www.wisemodel.cn/datasets/zjunlp/Mol-Instructions
https://wisemodel.cn/models/zjunlp/llama2-molinst-molecule-7b
https://wisemodel.cn/models/zjunlp/llama2-molinst-biotext-7b
https://wisemodel.cn/models/zjunlp/llama-molinst-protein-7b
数据及模型下载(huggingface):
https://huggingface.co/datasets/zjunlp/Mol-Instructions
https://huggingface.co/zjunlp/llama2-molinst-molecule-7b
https://huggingface.co/zjunlp/llama2-molinst-biotext-7b
https://huggingface.co/zjunlp/llama-molinst-protein-7b
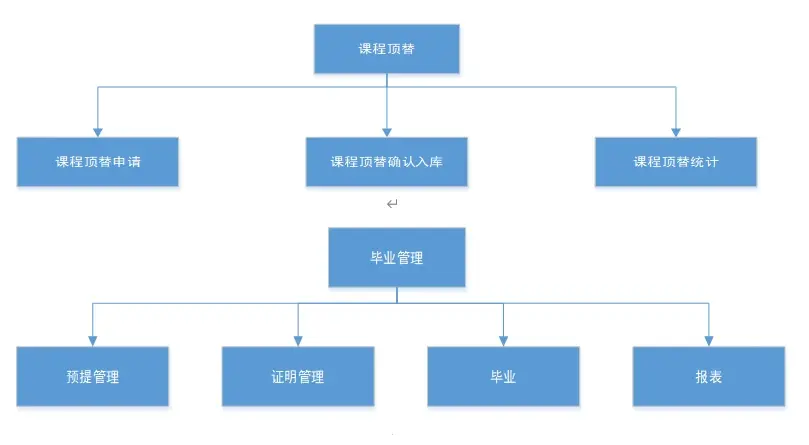
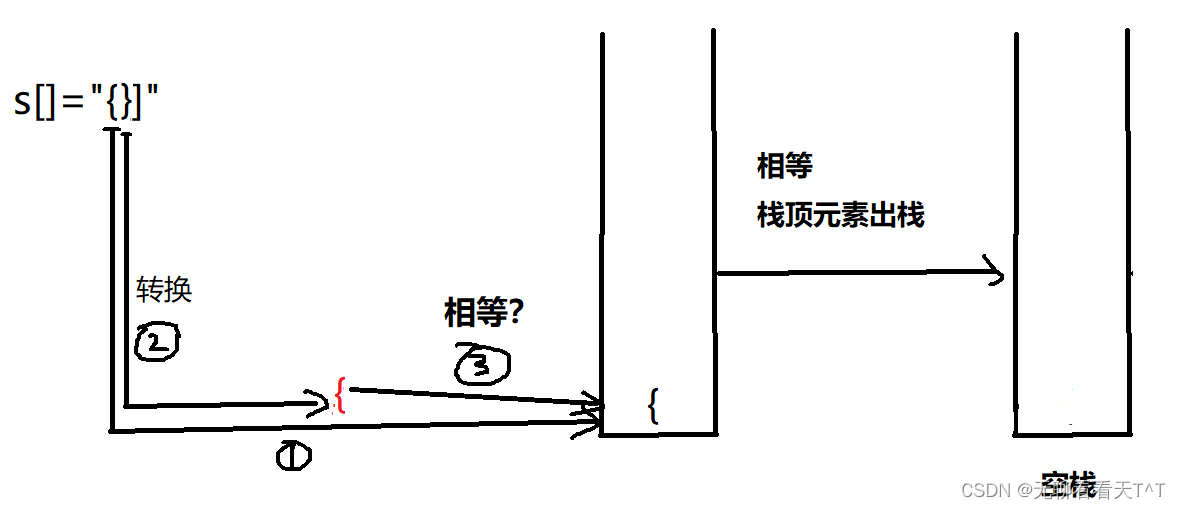
大语言模型(Large Language Model, LLM)在自然语言处理(NLP)领域的各种下游任务中表现出了卓越的性能,具备强大的文本理解和生成能力。随着这些大型模型逐渐突破传统文本处理的边界,它们在生物学、计算化学和药物开发等领域展示了巨大的潜力。然而,生物分子领域面临一系列挑战,如专用数据集的缺乏、数据注释的复杂性、多样的知识需求和缺乏标准化的表示方法等。为此,本文提出了Mol-Instructions,一个专门为生物分子研究中的各项任务定制的指令数据集(图1)。
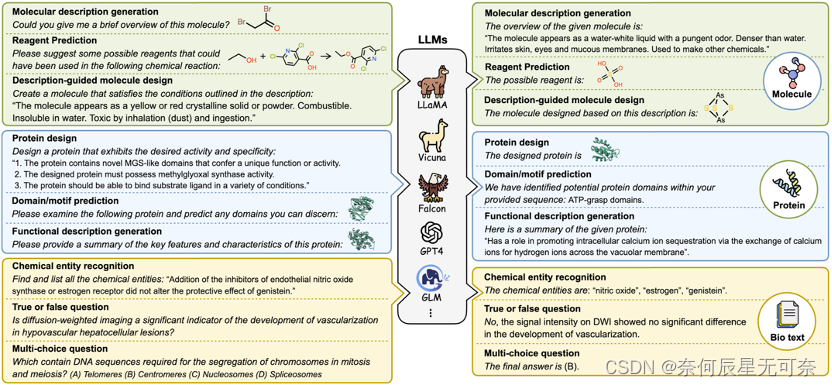
图1: Mol-Instructions为大模型赋能,解锁生物分子领域的各类挑战
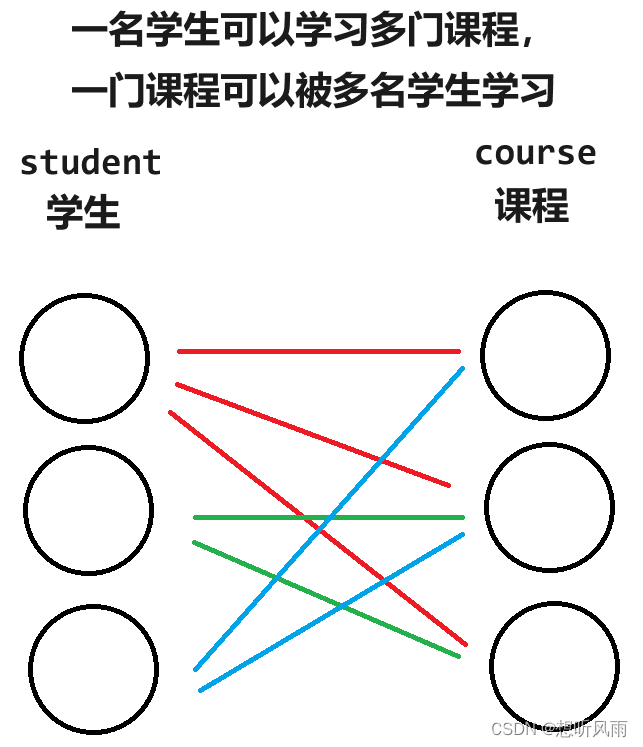
一、构建

图2: Mol-Instructions的构建过程
如图2所示,Mol-Instructions的构建遵循以下过程:
- 利用LLM的能力,生成多样化的任务描述,模拟人类需求的多样性。
- 通过不同的预处理方式将现有数据库中的数据转换为指令形式。
- 将结构化功能注释通过模版转化为文本形式。
- 对小分子和蛋白质序列进行质量控制,确保避免化学无效和冗余的序列。
二、概览
Mol-Instructions数据集包含2043K条指令数据,覆盖小分子、蛋白质和生物分子文本三大领域的17个关键任务(图3),包含了不同复杂度和结构的生物分子及丰富的文本描述(图4)。
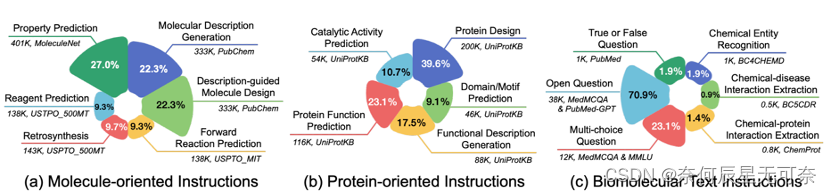
图3: Mol-Instructions涵盖的任务领域

图4:Mol-Instructions的数据多样性
三、实验分析
为衡量Mol-Instructions对LLMs理解和预测生物分子的帮助,本文对LLaMA-7B模型进行了指令微调,并从多个角度进行了定量实验分析。实验结果如图5、6、7所示,经Mol-Instructions指令微调的LLM在各项任务中的表现均优于其他对照模型,证明了Mol-Instructions在增强LLMs的生物分子理解和生成能力方面的重要性。
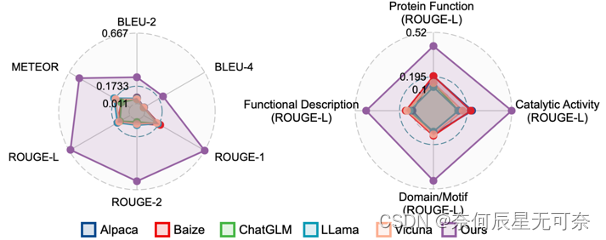
图5:小分子与蛋白质理解任务结果
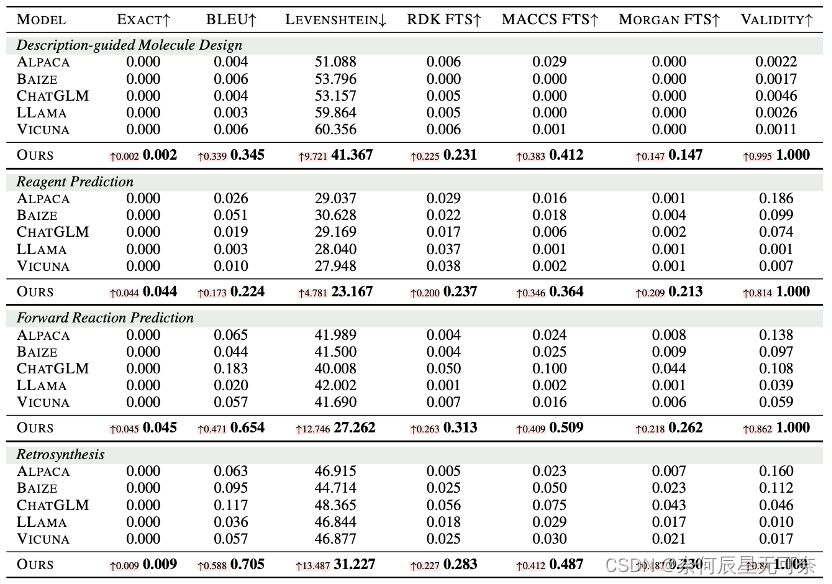
图6:分子生成任务结果
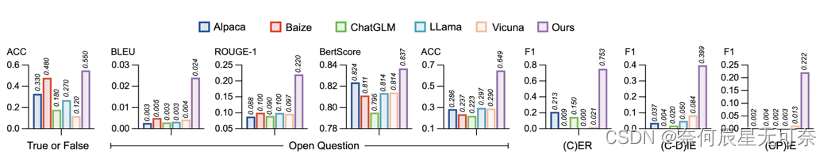
图7:自然语言处理任务结果
四、总结
Mol-Instructions可用于评估通用模型在从人类语言到生命语言的跨模态理解能力,显著提升大型模型对生物分子的理解能力,并可作为后续研究更深入探索生物分子设计和处理复杂生物问题的重要数据来源。由于文本与生物分子在表示空间上的差异,以及LoRA训练策略的限制,当前大型模型在掌握生物分子语言方面尚未达到掌握人类语言的熟练度。因此,探索扩展词汇表或将生物分子语言作为另一种模态纳入,可能是提高大型模型在生物分子任务中理解和性能的关键。