目录
- 6.4 多重序列比对数据库
- 6.5 基因组区域的多重序列比对
- 6.6 展望
- 6.7 常见问题
- 总结
6.4 多重序列比对数据库
Pfam:基于谱隐马尔可夫模型构建的蛋白质家族数据库
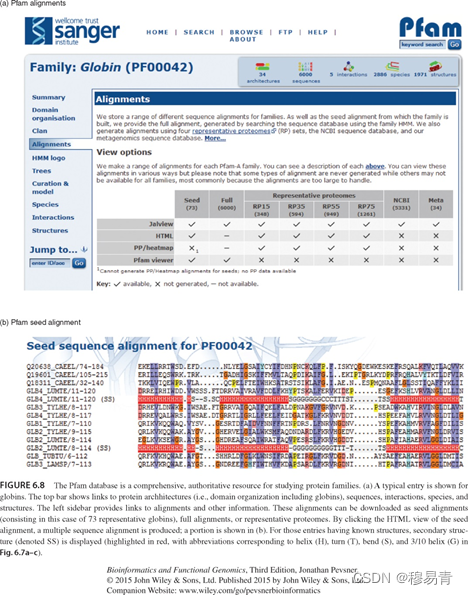

SMART:简易分子构型研究工具,与细胞信号传导、细胞外结构域以及染色质功能相关的蛋白质家族数据库
保守结构域数据库(CDD):基于序列或文本对Pfam和SMART进行查询,鉴别序列中的保守结构域
多序列比对数据库整合平台:InterPro和iProClass

6.5 基因组区域的多重序列比对
基因组区域与传统多重序列比对的差别
序列数目少,但序列更长
远缘物种通常包含明显的岛状保守区段,产生了关于多重序列比对中“锚”的思想
真核生物存在很多重复区域
出现大片明显的染色体缺失或倒位
基因组的比对没有基准集
使用UCSC分析基因组DNA比对
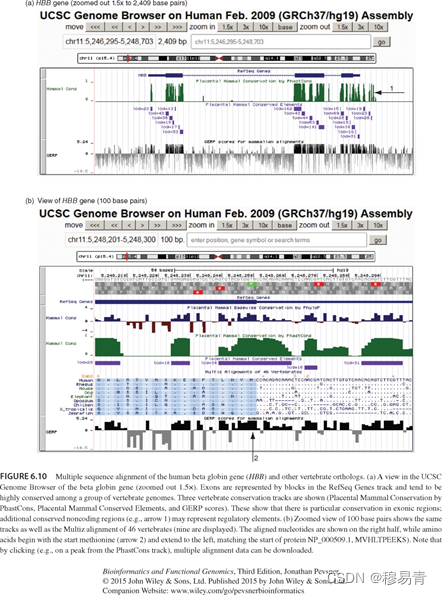
使用Galaxy分析基因组DNA比对
比对提取器:检索,裁剪感兴趣的区块进行比对
格式转换器:MAF转FASTA
MAF装订器:连接相邻区块
使用Ensembl分析基因组DNA比对
比对工具:BLASTZ与translated BLAT
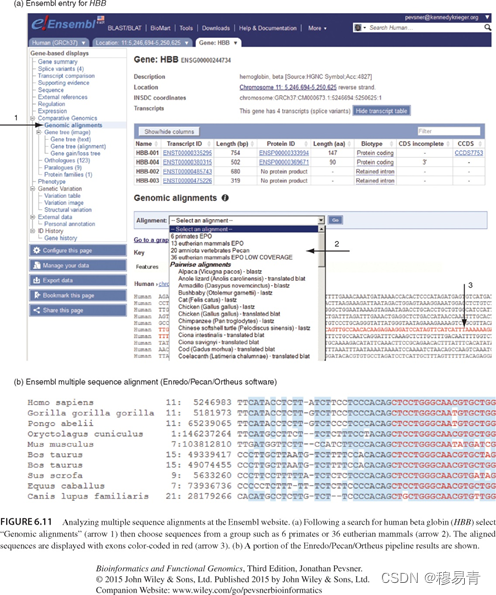
EPO流程

Alignathon大赛评估各种全基因组比对方法
6.6 展望
多重序列比对是针对可能划分在一起的蛋白质家族的操作
能够满足多种研究目的,包括鉴别保守的有重要功能的残疾
渐进式比对,基于迭代,一致性或结构信息的多重序列比对方式
大多数程序对于近缘物种比对表现较好,而远缘物种则性能差距较大
6.7 常见问题
由于缺乏标准,无法评估哪个程序对于远缘物种与其他近缘物种的比对结果更准确。因此,建立一个合适的多重序列比对对于分子系统发育的研究尤为重要。
总结
欢迎私信加群交流,vx:bbplayer2023。人数不多,但大家学习上进,互帮互助,值得一探究竟。









![【算法每日一练]-快速幂,倍增,滑动窗口(保姆级教程 篇1) #麦森数 #青蛙跳](https://img-blog.csdnimg.cn/550e34936324463ea1c1b964065b7854.png)

