文章目录
- 简介
- Install
- 必须参数
- 谱系数据集
- 输出结果
- 自动谱系选择
- 结果解读
- 完整
- 片段化
- 缺失
- 自动选择:多domain和污染匹配
- 注意
- BUSCO报告
- 常用脚本
- 真核
- Ref
简介
Busco评估基因组质量的核心原理在于通过计算基因组的通用单拷贝标记基因的比例来估计基因组的完整性。其中两个重要概念,高通用标记基因(High university)以及低重复比例(Low duplicability)。高通用标记基因定义为在**大于90%的物种中存在的直系同源基因(高通用性),低重复比例则意味着超过90%**的物种中是单拷贝基因(唯一性),核心思想就是保证尽可能地使用所有物种谱系中都存在的唯一基因。

Install
- conda一步安装
mamba create -n busco -c conda-forge -c bioconda busco=5.5.0
其他安装方式参见https://busco.ezlab.org/busco_userguide.html#manual-installation
必须参数
busco -i [SEQUENCE_FILE] -l [LINEAGE] -o [OUTPUT_NAME] -m [MODE] [OTHER OPTIONS]
-i或–in 定义要分析的输入文件,根据 BUSCO 模式,输入文件可以是核苷酸 fasta 文件或蛋白质 fasta 文件。从 5.1.0 版开始,输入参数也可以是包含 fasta 文件的目录,以便在批处理模式下运行。
-o或–out定义了包含所有结果、日志和中间数据的文件夹。
-m或–mode 设置评估模式:基因组、蛋白质、转录,有效选项
- geno or genome, for genome assemblies (DNA)
- tran or transcriptome, for transcriptome assemblies (DNA)
- prot or proteins, for annotated gene sets (protein)
-l或–lineage_dataset(谱系数据集)可以是数据集名称,如 bacteria_odb10,也可以是路径,如 ./bacteria_odb10 或 /home/user/bacteria_odb10。在前一种情况下,即推荐使用的情况下,BUSCO 会自动下载相应的数据集并将其版本化。在后一种情况下,将使用在给定路径中找到的数据集。如果运行自动系谱选择,可忽略系谱
基因组模式的默认基因预测工具是 Metaeuk(自 v5.0.0)(Metaeuk用于大规模真核生物元基因组学的敏感、高通量基因发现和注释技术)。之前的默认值是 Augustus。可选择使用 Augustus 或 Miniprot 代替 Metaeuk。要使用 Augustus,请使用 --augustus 标志。要使用 Miniprot,请使用 --miniprot 标志。Miniprot 5.5.0 测试版已经发布。
谱系数据集
BUSCO 利用谱系特异性信息来识别分析序列中的 BUSCO 基因。该信息可由用户指定,也可在细菌和古细菌中自动选择。
打印完整列表:
busco --list-datasets
(busco) [yut@node02 GTDBTK_r214_data]$ busco --list-datasets
2023-11-08 17:56:39 INFO: Downloading information on latest versions of BUSCO data...
2023-11-08 17:56:44 INFO: Downloading file 'https://busco-data.ezlab.org/v5/data/information/lineages_list.2021-12-14.txt.tar.gz'
2023-11-08 17:56:45 INFO: Decompressing file '/datanode02/yut/Database/GTDBTK_r214_data/busco_downloads/information/lineages_list.2021-12-14.txt.tar.gz'
################################################
Datasets available to be used with BUSCO v4 and v5:
bacteria_odb10
- acidobacteria_odb10
- actinobacteria_phylum_odb10
- actinobacteria_class_odb10
- corynebacteriales_odb10
- micrococcales_odb10
- propionibacteriales_odb10
- streptomycetales_odb10
- streptosporangiales_odb10
- coriobacteriia_odb10
- coriobacteriales_odb10
- aquificae_odb10
- bacteroidetes-chlorobi_group_odb10
- bacteroidetes_odb10
- bacteroidia_odb10
- bacteroidales_odb10
- cytophagia_odb10
- cytophagales_odb10
- flavobacteriia_odb10
- flavobacteriales_odb10
- sphingobacteriia_odb10
- chlorobi_odb10
- chlamydiae_odb10
- chloroflexi_odb10
- cyanobacteria_odb10
- chroococcales_odb10
- nostocales_odb10
- oscillatoriales_odb10
- synechococcales_odb10
- firmicutes_odb10
- bacilli_odb10
- bacillales_odb10
- lactobacillales_odb10
- clostridia_odb10
- clostridiales_odb10
- thermoanaerobacterales_odb10
- selenomonadales_odb10
- tissierellia_odb10
- tissierellales_odb10
- fusobacteria_odb10
- fusobacteriales_odb10
- planctomycetes_odb10
- proteobacteria_odb10
- alphaproteobacteria_odb10
- rhizobiales_odb10
- rhizobium-agrobacterium_group_odb10
- rhodobacterales_odb10
- rhodospirillales_odb10
- rickettsiales_odb10
- sphingomonadales_odb10
- betaproteobacteria_odb10
- burkholderiales_odb10
- neisseriales_odb10
- nitrosomonadales_odb10
- delta-epsilon-subdivisions_odb10
- deltaproteobacteria_odb10
- desulfobacterales_odb10
- desulfovibrionales_odb10
- desulfuromonadales_odb10
- epsilonproteobacteria_odb10
- campylobacterales_odb10
- gammaproteobacteria_odb10
- alteromonadales_odb10
- cellvibrionales_odb10
- chromatiales_odb10
- enterobacterales_odb10
- legionellales_odb10
- oceanospirillales_odb10
- pasteurellales_odb10
- pseudomonadales_odb10
- thiotrichales_odb10
- vibrionales_odb10
- xanthomonadales_odb10
- spirochaetes_odb10
- spirochaetia_odb10
- spirochaetales_odb10
- synergistetes_odb10
- tenericutes_odb10
- mollicutes_odb10
- entomoplasmatales_odb10
- mycoplasmatales_odb10
- thermotogae_odb10
- verrucomicrobia_odb10
archaea_odb10
- thaumarchaeota_odb10
- thermoprotei_odb10
- thermoproteales_odb10
- sulfolobales_odb10
- desulfurococcales_odb10
- euryarchaeota_odb10
- thermoplasmata_odb10
- methanococcales_odb10
- methanobacteria_odb10
- methanomicrobia_odb10
- methanomicrobiales_odb10
- halobacteria_odb10
- halobacteriales_odb10
- natrialbales_odb10
- haloferacales_odb10
eukaryota_odb10
- alveolata_odb10
- apicomplexa_odb10
- aconoidasida_odb10
- plasmodium_odb10
- coccidia_odb10
- euglenozoa_odb10
- fungi_odb10
- ascomycota_odb10
- dothideomycetes_odb10
- capnodiales_odb10
- pleosporales_odb10
- eurotiomycetes_odb10
- chaetothyriales_odb10
- eurotiales_odb10
- onygenales_odb10
- leotiomycetes_odb10
- helotiales_odb10
- saccharomycetes_odb10
- sordariomycetes_odb10
- glomerellales_odb10
- hypocreales_odb10
- basidiomycota_odb10
- agaricomycetes_odb10
- agaricales_odb10
- boletales_odb10
- polyporales_odb10
- tremellomycetes_odb10
- microsporidia_odb10
- mucoromycota_odb10
- mucorales_odb10
- metazoa_odb10
- arthropoda_odb10
- arachnida_odb10
- insecta_odb10
- endopterygota_odb10
- diptera_odb10
- hymenoptera_odb10
- lepidoptera_odb10
- hemiptera_odb10
- mollusca_odb10
- nematoda_odb10
- vertebrata_odb10
- actinopterygii_odb10
- cyprinodontiformes_odb10
- tetrapoda_odb10
- mammalia_odb10
- eutheria_odb10
- euarchontoglires_odb10
- glires_odb10
- primates_odb10
- laurasiatheria_odb10
- carnivora_odb10
- cetartiodactyla_odb10
- sauropsida_odb10
- aves_odb10
- passeriformes_odb10
- stramenopiles_odb10
- viridiplantae_odb10
- chlorophyta_odb10
- embryophyta_odb10
- liliopsida_odb10
- poales_odb10
- eudicots_odb10
- brassicales_odb10
- fabales_odb10
- solanales_odb10
viruses (no root dataset)
- alphaherpesvirinae_odb10
- baculoviridae_odb10
- rudiviridae_odb10
- betaherpesvirinae_odb10
- herpesviridae_odb10
- poxviridae_odb10
- tevenvirinae_odb10
- aviadenovirus_odb10
- enquatrovirus_odb10
- teseptimavirus_odb10
- bclasvirinae_odb10
- fromanvirus_odb10
- skunavirus_odb10
- betabaculovirus_odb10
- pahexavirus_odb10
- alphabaculovirus_odb10
- tunavirinae_odb10
- simplexvirus_odb10
- gammaherpesvirinae_odb10
- varicellovirus_odb10
- cheoctovirus_odb10
- guernseyvirinae_odb10
- tequatrovirus_odb10
- chordopoxvirinae_odb10
- peduovirus_odb10
- iridoviridae_odb10
- spounavirinae_odb10
这些数据集以文件夹形式排列,其中应包含

输出结果
执行 BUSCO 评估管道将创建一个以您为运行指定的名称命名的目录(使用 -o OUTPUT_NAME 强制选项设置)。
该目录将包含多个文件和目录:


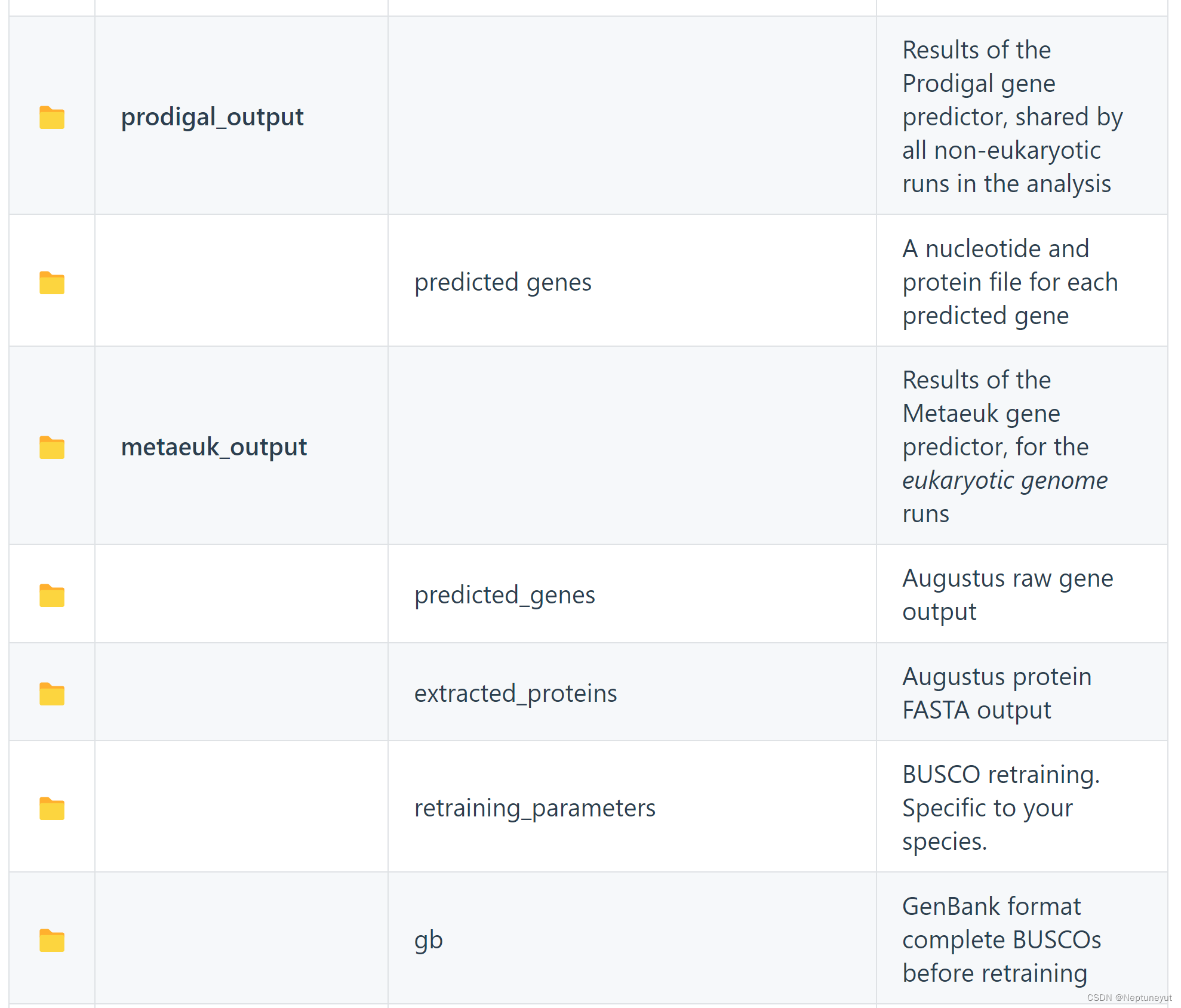

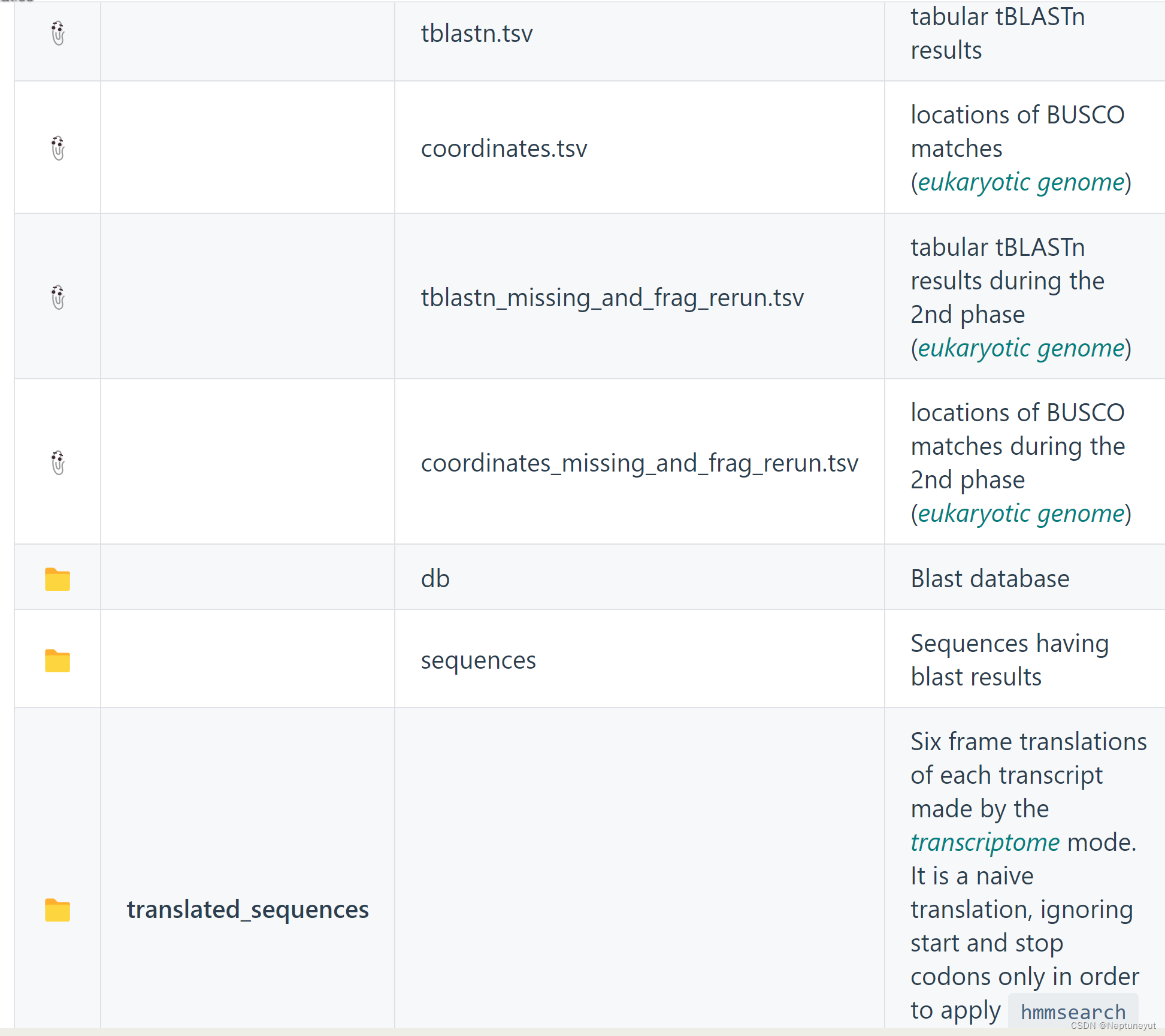
以批处理模式运行时(目录作为输入参数),输出目录的顶层包含以输入文件命名的子目录,其他结构与上述相同。在这种情况下,整个批次的日志文件只有一个 busco.log,保存在顶层的 logs/ 文件夹中。此外,还有一个 batch_summary.txt 文件,其中包含批次中所有运行的计分结果。
自动谱系选择
Requires SEPP
busco -m MODE -i INPUT -o OUTPUT --auto-lineage
or ignoring eukaryotes to save runtime, if compatible with your experimental goal.
busco -m MODE -i INPUT -o OUTPUT --auto-lineage-prok
or ignoring non-eukaryotes to save runtime, if compatible with your experimental goal.
busco -m MODE -i INPUT -o OUTPUT --auto-lineage-euk
结果解读
BUSCO 试图根据基因组组装、转录组或注释基因集的预期基因含量,对完整性进行定量评估。结果被简化为完整和单拷贝、完整和重复、片段或缺失的 BUSCOs 类别(Complete and single-copy, Complete and duplicated, Fragmented, or Missing BUSCOs.)。
BUSCO 完整性结果只有在生物体的生物学背景下才有意义。您必须了解缺失或重复的基因是生物起源还是技术起源。例如,高水平的重复可能是由最近的整体重复事件(生物学原因)或单倍型的嵌合组合(技术原因)造成的。转录组和蛋白质集如果没有经过同工酶筛选,就会导致高比例的重复。因此,您应该在进行 BUSCO 分析前对其进行过滤。最后,在转录组实验中专注于特定组织或特定生命阶段和条件不太可能产生 BUSCO 完整的转录组。在这种情况下,您的目标是使所有样本保持一致。
完整
如果发现是完整的,无论是单拷贝还是重复的,BUSCO 匹配结果的得分都在预期范围内,与 BUSCO 配置文件的比对长度也在预期范围内。如果输入数据集中实际上不存在直系同源物,或者直系同源物仅部分存在(高度片段化),而存在高识别度的全长同源物,则该同源物有可能被误认为是完整的 BUSCO。对评分阈值进行了优化,以尽量减少这种可能性,但这种情况仍有可能发生。
片段化
如果发现是片段,则表示 BUSCO 匹配的得分在得分范围内,但不在与 BUSCO 配置文件的长度比对范围内。对于转录组或注释基因组,这表示转录本或基因模型不完整。对于基因组组装,这可能表明基因仅部分存在,或者序列搜索和基因预测步骤未能生成全长基因模型,尽管全长基因可能确实存在于组装中。在运行真核生物数据集时,对产生这种片段结果的匹配会给予 “第二次机会”,即进行第二轮序列搜索和基因预测,并使用在发现完整的 BUSCOs 上训练的参数,但这仍可能无法恢复完整的基因。因此,基因组组装评估中的一些片段化 BUSCO 可能是完整的,但由于差异太大或基因结构非常复杂,因此很难对其进行完整的定位和预测。
缺失
如果发现缺失,则要么根本没有重要匹配,要么 BUSCO 匹配得分低于 BUSCO 配置文件的得分范围。对于转录组或注释基因组,这表明这些直系同源物确实缺失,或者转录本或基因模型非常不完整/支离破碎,甚至不符合被视为片段化的标准。对于基因组组装,这可能表明这些直系同源物确实缺失,或者序列搜索步骤未能识别出任何重要的匹配项,或者基因预测步骤甚至未能产生可能被识别为片段 BUSCO 匹配项的部分基因模型。与片段匹配一样,在运行真核生物数据集时,对第一轮序列搜索后缺失的 BUSCO 也会给予 “第二次机会”,即进行第二轮序列搜索,并使用在完整 BUSCO 上训练的参数进行基因预测,但这仍可能无法恢复基因。因此,在基因组组装评估中缺失的一些 BUSCO 可能部分存在,甚至可能(但不太可能)完整,但它们的差异太大或基因结构非常复杂,因此很难正确定位和预测,甚至很难部分预测。
自动选择:多domain和污染匹配
自动谱系选择过程在古菌、细菌和真核生物等领域的通用谱系数据集上运行 BUSCO。一旦选择了最佳领域,BUSCO 就会根据系统发育位置自动尝试寻找最合适的 BUSCO 数据集。当使用适当的数据集时,即数据集属于要测试物种的系时,BUSCO 评估才有效。由于域间存在重叠标记/假匹配,因此在另一个域中的 busco 匹配并不一定意味着您的基因组/蛋白质组中包含了该域的序列。不过,多个域中的高 busco 分数可能有助于您识别可能的污染。
注意
- 一般来说,为评估而选择的品系应是现有的最具体的品系,例如,评估鱼类数据时,应选择翼手目品系,而不是中生代品系。
- 如果要评估大量的物种/品系/版本等,那么为了尽量缩短运行时间(以牺牲分辨率为代价),可以选择较少 BUSCO 的不太特定的品系集,例如,如果要评估 20 个鸟类基因组,每个基因组都有几个不同的组装版本,那么可以选择脊椎动物或后生动物metazoa ,而不是鸟类品系,至少在最初几轮评估中是这样。
- 评估通常会产生多个文件夹,其中包含大量文件。这些文件是为您准备的,以便您可以更详细地检查个别案例和/或将数据用于下游分析。
- 请花一些时间检查日志文件,这些文件是为您的利益准备的,以便突出显示 BUSCO 运行期间可能出现的潜在问题。
- 将您的数据评估结果与其他密切相关物种的相应公开数据评估结果进行比较。这样,BUSCO 的结果就可以用来证明您的数据集与现有的类似物种公开数据集一样好,甚至更好。
- 如果对注释基因集进行了人工整理,请报告整理前后的 BUSCO 结果,以量化改进情况。
BUSCO报告
- 用简单的 BUSCO 符号报告结果:C:89.0%[S:85.8%,D:3.2%],F:6.9%,M:4.1%,n:3023
- 使用 generate_plot.py(见下文)脚本为出版物的辅助在线信息生成简单的图表摘要(可轻松定制)。
- 报告您使用的所有第三方组件的版本。我们强烈推荐使用 BUSCO 容器,其版本足以安全地重现运行。
- 报告您用于评估的 BUSCO 数据集。请注明数据集的创建日期,而不仅仅是名称,例如 archaea_odb10 (2019-01-04)。
- 报告您使用的 BUSCO 选项。
- 报告您评估的基因组组装、注释基因组或转录组的版本
常用脚本
通过 scripts/generate_plot.py 脚本,用户可以在易于理解的条形图中快速查看 BUSCO 的汇总结果。脚本/generate_plot.py 使用 R (https://www.r-project.org/) 和 ggplot2 (http://ggplot2.org/) 对 BUSCO 运行结果进行汇总,以便进行并排比较。该脚本会生成一张 PNG 图像(如果 R 和 ggplot2 都可用),以及一个 R 源代码文件,该文件可用于在 R 和 ggplot2 都可用的不同机器上运行,也可对其进行编辑,以完全自定义所生成的条形图(颜色、标签、字体、坐标轴等)。
usage: python3 generate_plot.py -wd [WORKING_DIRECTORY] [OTHER OPTIONS]
BUSCO plot generation tool.
Place all BUSCO short summary files (short_summary.[generic|specific].dataset.label.txt) in a single folder. It will be your working directory, in which the generated plot files will be written
See also the user guide for additional information
required arguments:
-wd PATH, --working_directory PATH
Define the location of your working directory
optional arguments:
-rt RUN_TYPE, --run_type RUN_TYPE
type of summary to use, `generic` or `specific`
--no_r To avoid to run R. It will just create the R script file in the working directory
-q, --quiet Disable the info logs, displays only errors
-h, --help Show this help message and exit
要运行 scripts/generate_plot.py,首先要创建一个文件夹,例如 mkdir BUSCO_summaries,然后将要绘制的每个运行的 BUSCO 简摘要文件复制到该文件夹中。
cp XX1/short_summary.*.lineage_odb10.XX1.txt BUSCO_summaries/.
cp XX2/short_summary.*.lineage_odb10.XX2.txt BUSCO_summaries/.
cp XX3/short_summary.*.lineage_odb10.XX3.txt BUSCO_summaries/.
然后,只需运行脚本,并将您创建的包含要绘制的摘要的文件夹名称(或完整路径,如果不在同一工作目录下)作为参数。
python3 scripts/generate_plot.py –wd BUSCO_summaries
python3 scripts/generate_plot.py –wd /full/path/to/my/folder/BUSCO_summaries
生成的 PNG 图像和相应的 R 源代码文件将放在包含 BUSCO 摘要的同一文件夹中。默认情况下,运行名称被用作每个绘制结果的标签,而这是从简短摘要文件名中自动提取的:因此对于 short_summary.generic.lineage_odb10.XX1.txt,标签应为 XX1。您也可以编辑 R 源代码文件,更改任何绘制参数,然后在 R 环境中手动运行代码,绘制个性化条形图。
示例 scripts/generate_plot.py 条形图:
mkdir my_summaries
cp SPEC1/short_summary.generic.lineage1_odb10.SPEC1.txt my_summaries/.
cp SPEC2/short_summary.generic.lineage2_odb10.SPEC2.txt my_summaries/.
cp SPEC3/short_summary.specific.lineage2_odb10.SPEC3.txt my_summaries/.
cp SPEC4/short_summary.generic.lineage3_odb10.SPEC4.txt my_summaries/.
cp SPEC5/short_summary.generic.lineage4_odb10.SPEC5.txt my_summaries/.
python3 scripts/generate_plot.py –wd my_summaries

真核
(busco) [yut@node02 TestData]$ time busco -i SX22-37_unicycler/assembly.fasta -c 8 -m geno -l eukaryota_odb10 -f --out busco_out
# -i Input sequence file in FASTA format. Can be an assembled genome or transcriptome (DNA), or protein sequences from an annotated gene set. Also possible to use a path to a directory containing multiple input files.
-m MODE, --mode MODE Specify which BUSCO analysis mode to run.
There are three valid modes:
- geno or genome, for genome assemblies (DNA)
- tran or transcriptome, for transcriptome assemblies (DNA)
- prot or proteins, for annotated gene sets (protein)
-l LINEAGE, --lineage_dataset LINEAGE:bacteria_odb10,archaea_odb10,eukaryota_odb10,viruses
Specify the name of the BUSCO lineage to be used.
busco --list-datasets
-c N, --cpu N Specify the number (N=integer) of threads/cores to use.
-f, --force Force rewriting of existing files. Must be used when output files with the provided name already exist.



![P1131 [ZJOI2007] 时态同步](https://img-blog.csdnimg.cn/img_convert/0115519d25d9a72ea66466f0e3a245b8.png)