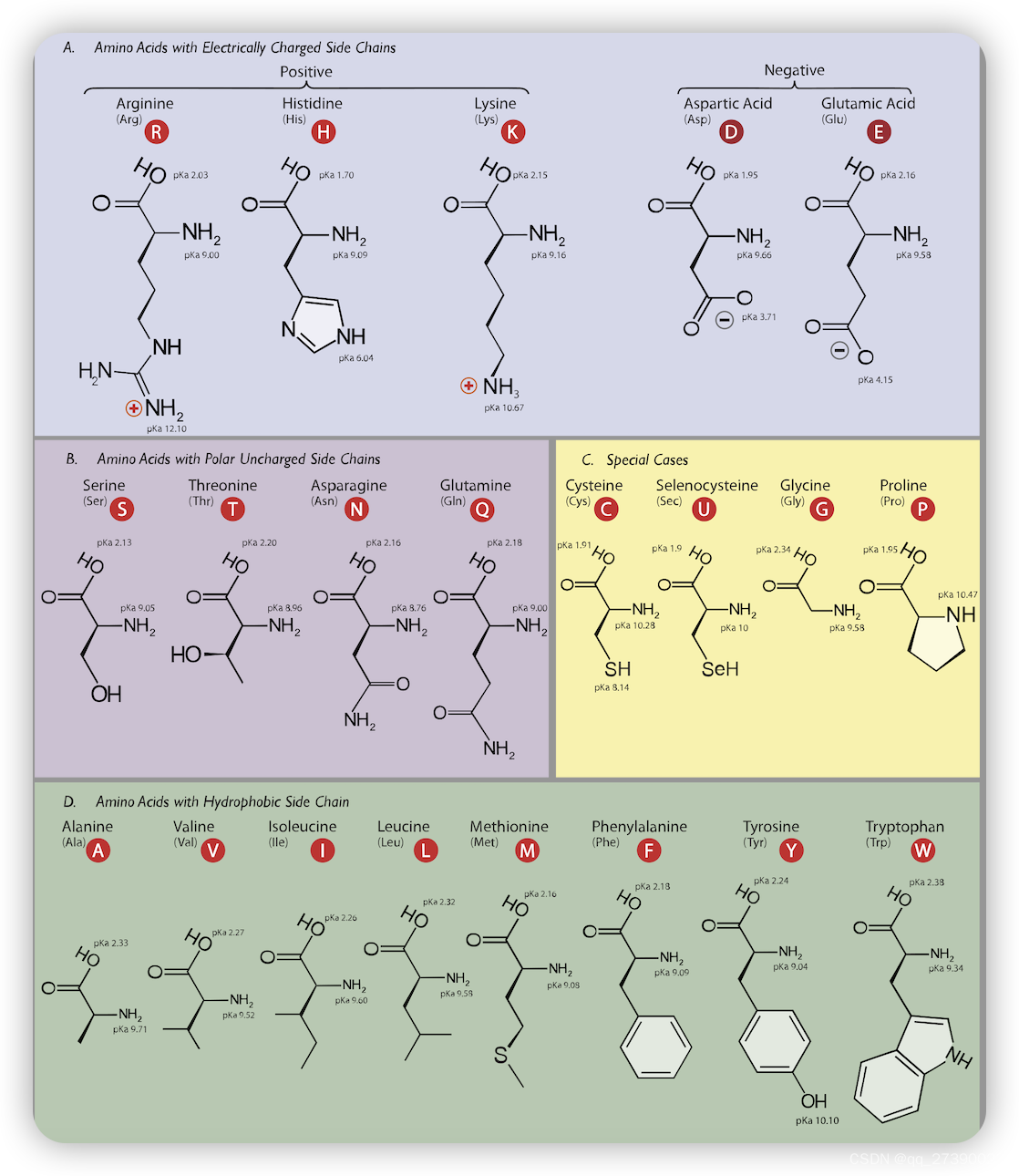
蛋白质中的"chi角"(chi angle)通常是指侧链自由旋转的二面角,用于描述氨基酸侧链中的旋转构象。侧链是氨基酸分子的一部分,它们附着在氨基酸主链上,并可以以不同的角度自由旋转。chi角用于描述侧链旋转的几何构象。
不同氨基酸的侧链具有不同数量的chi角,通常分为chi1、chi2、chi3、chi4等。每个chi角表示了侧链上的一个二面角,通常是在侧链的主要链或原子之间的角度。
Chi角的值通常以角度(度)来表示,可以从0度到360度。Chi角的不同构象可以影响蛋白质的立体构象和功能,因此对于研究蛋白质的结构和功能具有重要意义。
import numpy as np
### 1. 定义常量
## 蛋白质中氨基酸种类,单字母表示
restypes = [
'A', 'R', 'N', 'D', 'C', 'Q', 'E', 'G', 'H', 'I', 'L', 'K', 'M', 'F', 'P',
'S', 'T', 'W', 'Y', 'V'
]
## 氨基酸单字母和三字母表示的转化
restype_1to3 = {
'A': 'ALA',
'R': 'ARG',
'N': 'ASN',
'D': 'ASP',
'C': 'CYS',
'Q': 'GLN',
'E': 'GLU',
'G': 'GLY',
'H': 'HIS',
'I': 'ILE',
'L': 'LEU',
'K': 'LYS',
'M': 'MET',
'F': 'PHE',
'P': 'PRO',
'S': 'SER',
'T': 'THR',
'W': 'TRP',
'Y': 'TYR',
'V': 'VAL',
}
## 不同氨基酸侧链的二面角的原子,Chi1 角,Chi2 角,Chi3 角,Chi4 角
chi_angles_atoms = {
'ALA': [],
# Chi5 in arginine is always 0 +- 5 degrees, so ignore it.
'ARG': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'CD'],
['CB', 'CG', 'CD', 'NE'], ['CG', 'CD', 'NE', 'CZ']],
'ASN': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'OD1']],
'ASP': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'OD1']],
'CYS': [['N', 'CA', 'CB', 'SG']],
'GLN': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'CD'],
['CB', 'CG', 'CD', 'OE1']],
'GLU': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'CD'],
['CB', 'CG', 'CD', 'OE1']],
'GLY': [],
'HIS': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'ND1']],
'ILE': [['N', 'CA', 'CB', 'CG1'], ['CA', 'CB', 'CG1', 'CD1']],
'LEU': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'CD1']],
'LYS': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'CD'],
['CB', 'CG', 'CD', 'CE'], ['CG', 'CD', 'CE', 'NZ']],
'MET': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'SD'],
['CB', 'CG', 'SD', 'CE']],
'PHE': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'CD1']],
'PRO': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'CD']],
'SER': [['N', 'CA', 'CB', 'OG']],
'THR': [['N', 'CA', 'CB', 'OG1']],
'TRP': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'CD1']],
'TYR': [['N', 'CA', 'CB', 'CG'], ['CA', 'CB', 'CG', 'CD1']],
'VAL': [['N', 'CA', 'CB', 'CG1']],
}
# This mapping is used when we need to store atom data in a format that requires
# fixed atom data size for every residue (e.g. a numpy array).
atom_types = [
'N', 'CA', 'C', 'CB', 'O', 'CG', 'CG1', 'CG2', 'OG', 'OG1', 'SG', 'CD',
'CD1', 'CD2', 'ND1', 'ND2', 'OD1', 'OD2', 'SD', 'CE', 'CE1', 'CE2', 'CE3',
'NE', 'NE1', 'NE2', 'OE1', 'OE2', 'CH2', 'NH1', 'NH2', 'OH', 'CZ', 'CZ2',
'CZ3', 'NZ', 'OXT'
]
atom_type_num = len(atom_types) # := 37.
### 2. 定义函数
def chi_angle_atom(atom_index: int) -> np.ndarray:
"""Define chi-angle rigid groups via one-hot representations."""
chi_angles_index = {}
one_hots = []
for k, v in chi_angles_atoms.items():
## 查看 chi_angles_atoms,atom_types的数据结构
## 20种氨基酸每一个chi角的第n位(参数,0-3)原子的编号
indices = [atom_types.index(s[atom_index]) for s in v]
## 有的氨基酸侧链短,没有四个chi角,用-1 填充indices
indices.extend([-1]*(4-len(indices)))
## 键是氨基酸名称,值是indices;例 ARG:[1, 3, 5, 11]
chi_angles_index[k] = indices
for r in restypes:
res3 = restype_1to3[r]
one_hot = np.eye(atom_type_num)[chi_angles_index[res3]]
one_hots.append(one_hot)
# list,含有21个array,每个array给出chi角index原子的one-hot编码
one_hots.append(np.zeros([4, atom_type_num])) # Add zeros for residue `X`.
#print("one_hots:")
#print(len(one_hots))
#print(one_hots)
# list -> np.ndarray, 指定轴上堆叠多个数组
one_hot = np.stack(one_hots, axis=0)
# print(one_hot.shape) # (21, 4, 37)
one_hot = np.transpose(one_hot, [0, 2, 1])
# print(one_hot.shape) # (21, 37, 4)
return one_hot
### 3.调用函数
chi_atom_1_one_hot = chi_angle_atom(1)
chi_atom_3_one_hot = chi_angle_atom(3)
print(chi_atom_1_one_hot)
print(chi_atom_3_one_hot)