今天给同学们分享一篇生信文章“Single-cell transcriptomics reveals immune infiltrate in sepsis”,这篇文章发表在Front Pharmacol期刊上,影响因子为5.6。

结果解读
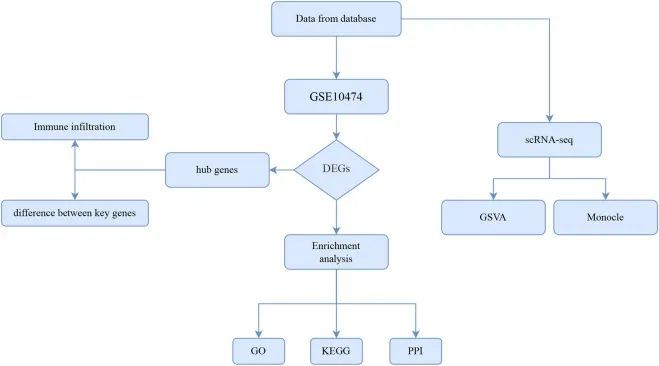
作者研究的流程图
作者首先制定了这项研究的总体技术路线,如图1所示。
数据标准化和免疫相关基因筛选
根据GEO数据平台对DEG的数据进行总结和整理,并将分析的脓毒症样本和正常样本的数据进行比较。作者从一个探针中移除了对应于多个分子的探针,当遇到对应于同一分子的探针时,只保留了信号值最大的探针。在对GSE28750进行数据预处理后,共过滤了21655个分子,其中1198个分子满足|log2(FC)|>1和p.adj<0.05。在上述阈值下(|log2(FC)|>1和p.adj<0.05),与对照组相比,脓毒症组599个基因上调,599个下调。作者使用R软件从基因表达矩阵中提取相互差异表达的基因,如热图(图2A)和火山图(图2B)所示。作者手动搜索了包含免疫相关基因的原始文献和综述

免疫相关基因和单细胞富集分析
作者对免疫相关基因进行了GO分析,结果显示交叉基因主要与T细胞受体结合、免疫受体活性、SH2结构域结合、信号受体复合物衔接子活性、趋化因子活性等生物学现象有关(图4A)。结果表明,交集基因主要与T细胞受体结合、免疫受体活性、SH2结构域结合、信号受体复合物衔接子活性、趋化因子活性等生物学现象有关。KEGG分析结果显示,交叉基因富集于原发性免疫缺陷、癌症的造血细胞谱系、PD-L1表达和PD-1检查点通路、癌症的T细胞受体信号通路、PD-L1表现和PD-1检测点通路、T细胞受体信号通路和细胞因子-细胞因子受体相互作用通路(图4B)。

基于免疫相关基因PPI网络的Hub基因筛选
作者使用STRING工具对免疫相关基因进行蛋白质-蛋白质相互作用(PPI)分析,并可视化每个免疫相关基因之间的相互作用数量。所涉及的基因越大,节点的程度就越大,线越粗,介数就越大(图5A)。随后,作者利用基于Cytoscape软件的cytoHubba插件的最大群体中心性(MCC)、最大邻域分量(MNC)和最大邻域分量密度(DMNC)算法筛选了7个关键基因(图5B)。选择通过上述三种算法筛选的关键基因的交叉点,筛选结果将七个关键基因描述为CD28、CD3D、CD2、CD4、IL7R、LCK和CD3E(图5B)。

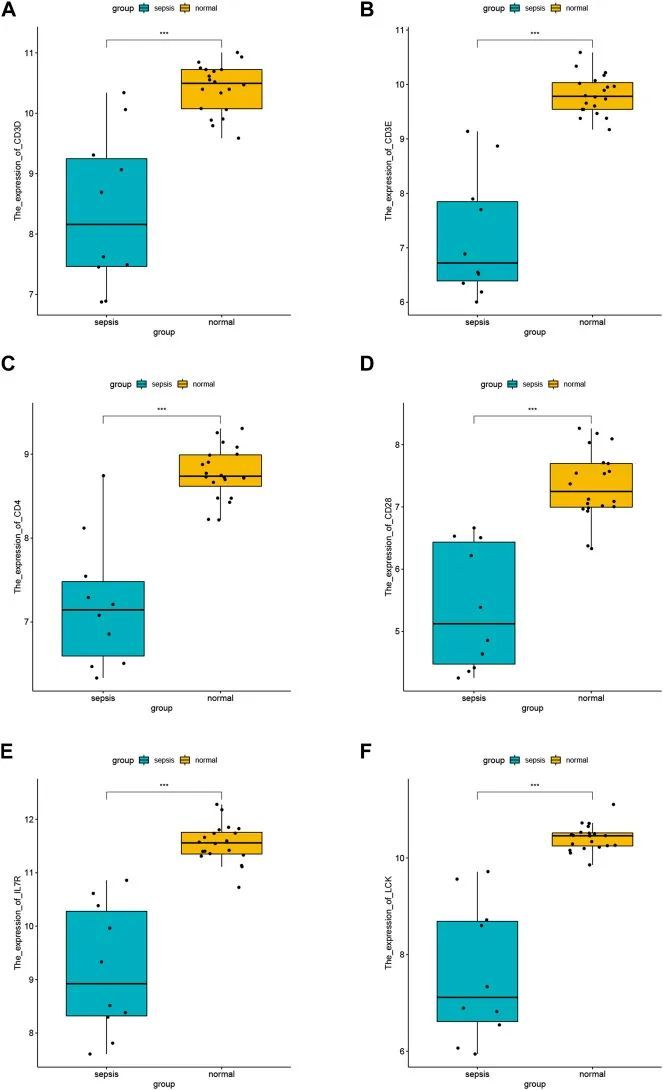
hub基因的差异分析
作者在数据集GSE28750中可视化了除CD2外的其他六个关键基因的表达值,并显示了差异。结果显示,脓毒症组和正常组之间CD3D、CD3E、CD4、CD28、IL7R和LCK的表达水平存在统计学差异。在脓毒症样本中观察到hub基因的低表达,这在所有六个hub基因中都是一致的。
使用GSE28750数据集的脓毒症免疫浸润分析
基于CIBERSORT算法,对GSE28750数据中的脓毒症样本和对照样本进行免疫细胞浸润分析,分析每个样本中免疫细胞的比例,得到脓毒症样本和控制样本中22种免疫细胞的占比(图7A)。此外,作者对脓毒症样本和对照样本之间的22种类型的免疫细胞进行了组间比较(图7B)。作者观察到脓毒症样本和对照样本之间B细胞幼稚、浆细胞、T细胞CD8、T细胞CD4幼稚、T细胞CD4记忆静息、T细胞t4记忆激活、T细胞γ-δ、NK细胞静息、单核细胞、巨噬细胞M0、嗜酸性粒细胞、中性粒细胞的显著差异(图7B)。此外,根据基因表达和相应免疫细胞的丰度,以及相互作用,分析了关键基因与免疫细胞之间的相关性

细胞的轨迹分析
根据3.4分析结果显示,差异基因在T细胞中的得分较高。接下来,作者能够在T细胞的轨迹分析中捕捉到T细胞分化为记忆性T细胞、NK T细胞、CD4+T细胞、CD8+T细胞,Th T细胞和Treg T细胞(图8A)。

体内动物实验
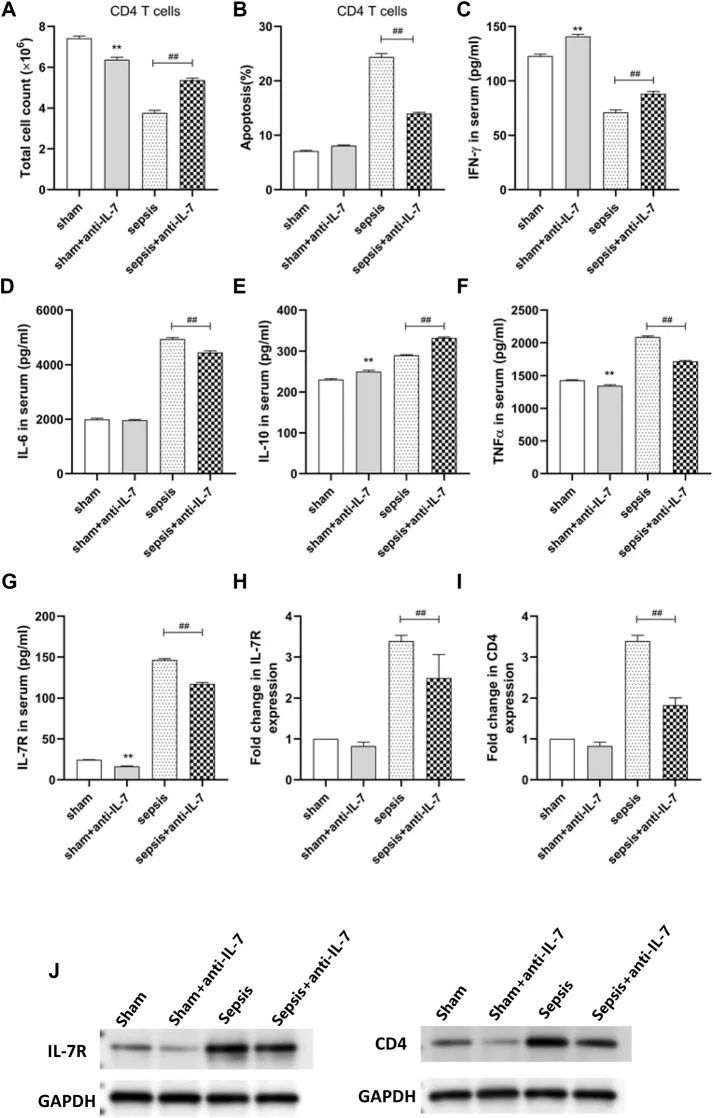
作者发现,CD4 T细胞的细胞计数在假手术组中最高,而在脓毒症组中最低(图9A)。IL-7抗体治疗可以增加CD4 T细胞的计数(图9A),这可能是细胞凋亡增加的原因(图9B)。脓毒症组外周血中IFN-γ浓度最低,IL-7抗体治疗可增加IFN-γ的浓度(图9C)。脓毒症组外周血中IL-6浓度最高,IL-7抗体治疗可降低IL-6浓度(图9D)。IL-7抗体治疗可以增加IL-10的浓度。脓毒症组外周血中TNF-γ浓度最高,IL-7抗体治疗可降低TNF-α浓度。脓毒症组外周血中IL-7R的浓度最高,IL-7抗体治疗可降低IL-7R浓度。IL-7 a
总结
脓毒症一直是一个重要的临床问题。早期发现和及时治疗脓毒症具有重要意义。目前,有许多与脓毒症相关的生存预测模型。例如,基于调节性T细胞中单个免疫检查点的PD-1过表达可以预测脓毒症患者的预后,以及其验证脓毒症患者28天死亡率的能力。然而,脓毒症的发病机制是复杂的,尤其是免疫机制。