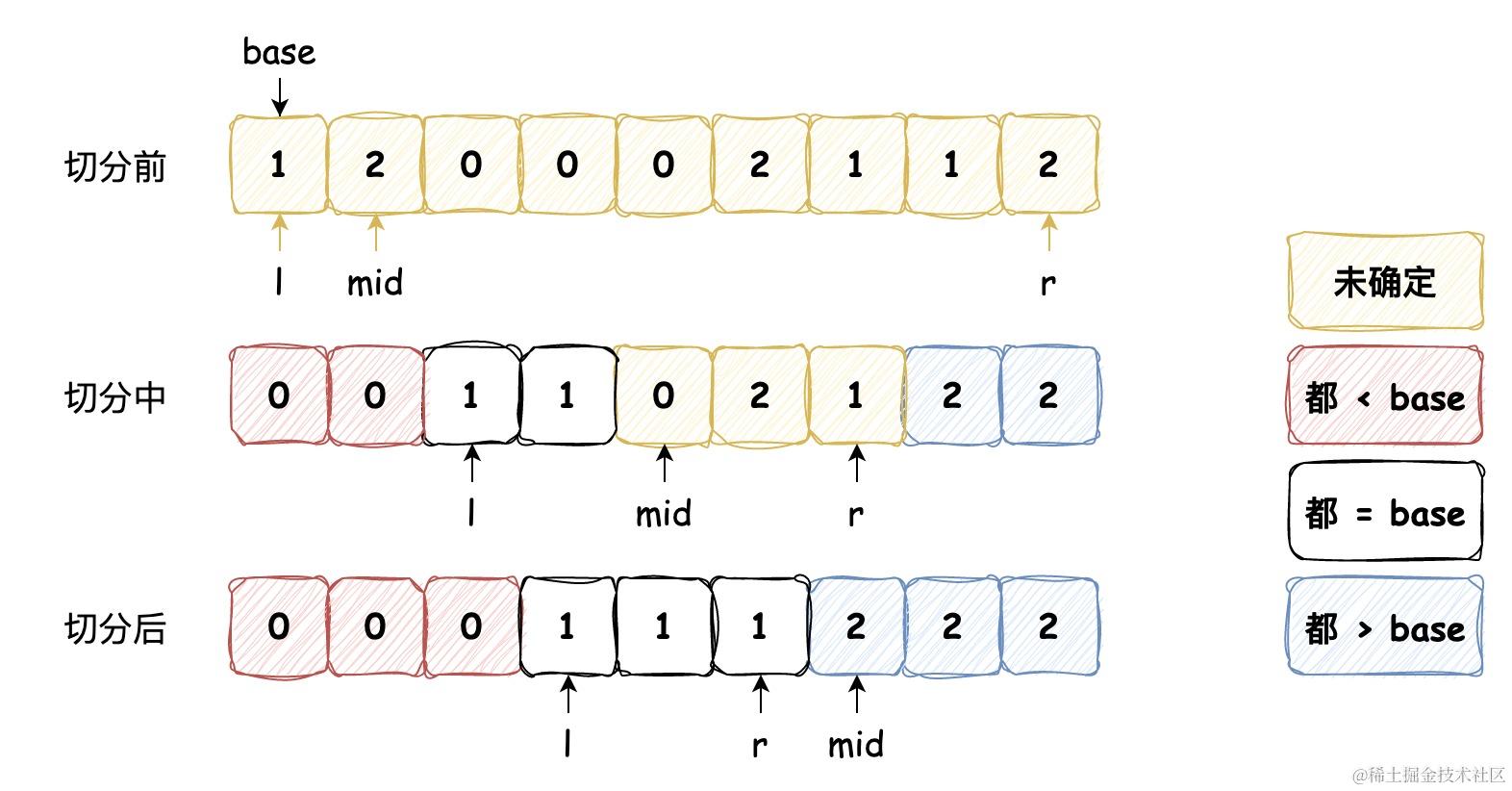
困难,还是一如既往的不会做,但是得写写自己的想法
- 先从根节点开始作深度搜索,对于每一个以 node 为根的子树,我们返回该子树排序后的基因集合,类似与归并排序
- 显然在每次合并集合进行排序的时候我们就可以知道 node 子树内缺失的最小基因值
- 我是觉得时间复杂度就在 O(nlogn),顶多n之前多几个常数系数,但还是超时了
正解:
- set集合的查找是 O(1) 的,当通过 set1.update(set2) 来合并两个 set 时(其中 set1 比 set2 大),同时对树作 dfs 是 O(nlogn)
- 所以不用排序合并,直接从 node 节点的子树中的最大缺失的最小基因值开始找 node 树的缺失的最小基因值,这个过程是 O(n)
- 所以总共是 O(nlogn)
class Solution:
def smallestMissingValueSubtree(self, parents: List[int], nums: List[int]) -> List[int]:
n = len(parents)
child = defaultdict(list)
for i in range(n):
if parents[i] == -1:
continue
child[parents[i]].append(i)
ans = [1] * n
def find(node):
childs = [[nums[node]]]
for c in child[node]:
childs.append(find(c))
l, ans[node] = sort0(childs)
return l
def sort0(ll):
a, b = [], []
if len(ll) > 2:
a, _ = sort0(ll[:len(ll)//2])
b, _ = sort0(ll[len(ll)//2:])
elif len(ll) == 2:
a = ll[0]
b = ll[1]
else:
return ll[0], 1 if ll[0][0] != 1 else 2
t = []
m = 1
while len(a) > 0 and len(b) > 0:
if a[0] > b[0]:
if b[0] == m:
m += 1
t.append(b[0])
b.pop(0)
else:
if a[0] == m:
m += 1
t.append(a[0])
a.pop(0)
while len(a) > 0:
if a[0] == m:
m += 1
t.append(a[0])
a.pop(0)
while len(b) > 0:
if b[0] == m:
m += 1
t.append(b[0])
b.pop(0)
return t, m
find(0)
return ans