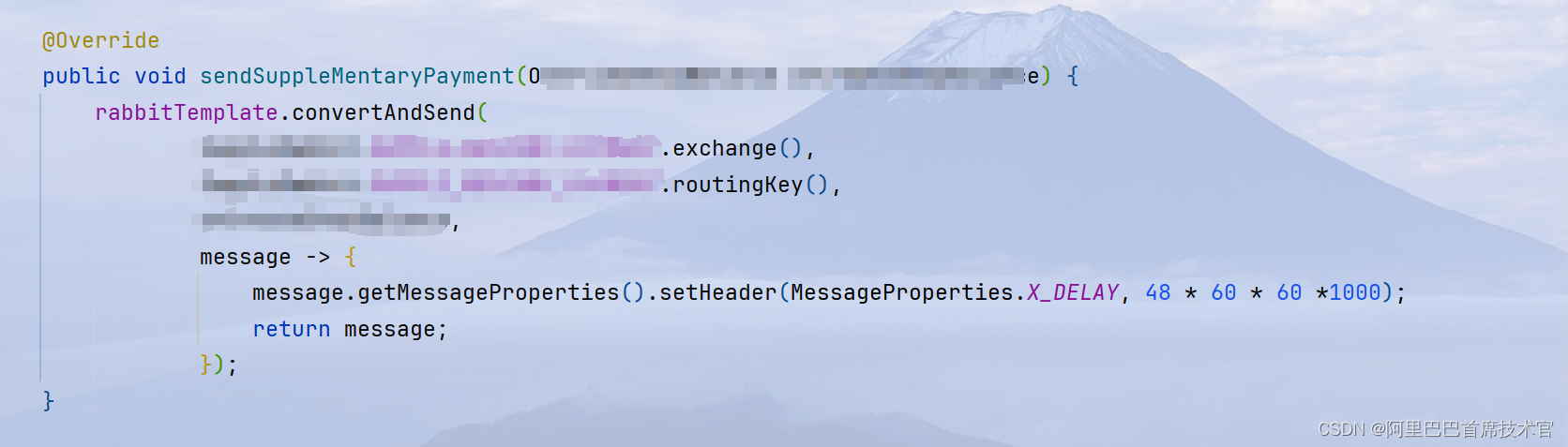
NNUNet使用自定义医疗图像分割数据集进行分割训练
主要讲解怎么把自己的数据放到nnUnet进行训练,不涉及nnUnet的原理和推导讲解。
1、转换的思路。
从NNUNet的开源代码中可以看到,NNUnetV2已经支持了很多的数据格式。但是因为其底层的逻辑主要是解决医学十项全能的任务,所以对于医疗图像的分割,建议按照医学十项分割的数据格式准备你的数据,从而有利于执行后续的转换。下图列举了医学十项全能的数据存储结构:
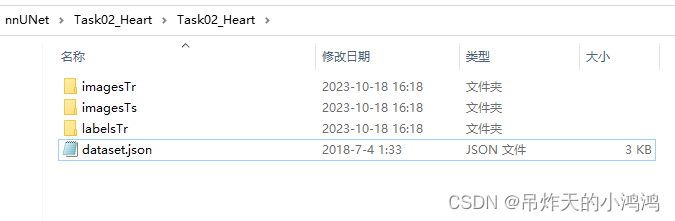 imagesTr代表训练集的图像数据,imagesTs代表的是测试集的图像。labelsTr代表的是测试集图像。
imagesTr代表训练集的图像数据,imagesTs代表的是测试集的图像。labelsTr代表的是测试集图像。
dataset.json—存储数据的基础信息,一般如下:

如果按照上图这样存储的数据,很多时候直接调用NNUNet代码中Dataset_conversion中的相关代码就可以执行转换了。可以看到这里面也提供了一些非十项全能任务的转换代码。
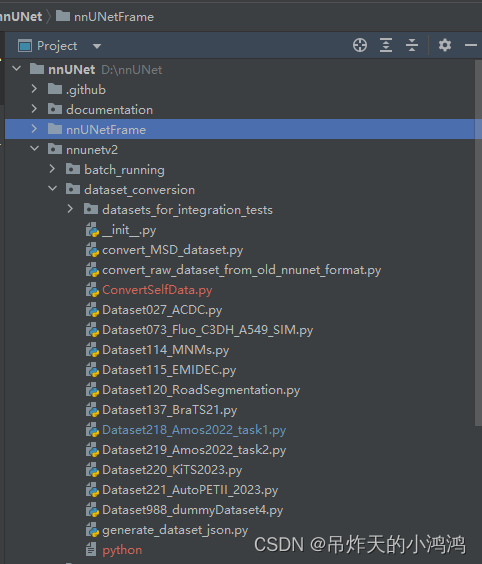
重头戏l来了。要转换,就要知道为什么要转换。接下来直接贴NNUNet官网的数据结构,下图为
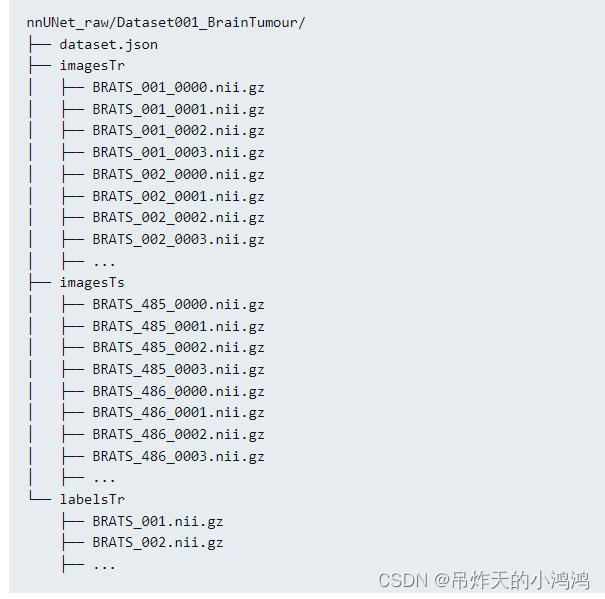
再看看我们刚才自己的数据存储结构不一样:

可以看到其实主要的差别就是在存储图像的时候,NNUNetV2在图像的后面都加上了通道数0000、0001这样的标识。当然这里dataset.json保存的信息也有一定的变化:如下

大致和前面十项全能的类似的,但是将 “modality"改成了"channel_names” 这个主要是为了通用;第二个增加了file_endding这个关键字,这个是确定NNUNet调用什么库对图像进行读取的。
从上面的讲解中,大家应该知道了NNUNet所采取的文件结构以及dataset.json的内容。所以接下来我们就开始真正的进行数据转换。当然也可以直接把你的数据就按照NNUNet所要求的进行准备,那就不用转换了。
二、转换过程。
此处讲解的是如下结构的数据转换:
可以看到我们这里和NNUNetV2要求的数据存储结构,主要的区别就是在于文件的末尾没有添加通道标识,以及缺少dataset.json一般自己采集的数据都没有。
所以我们接下的任务就有两个:
1、更改文件名,并将其数据转移到nnUnet_raw对应的文件夹中
2、生成dataset.json文件。
以上两个任务呢就可以通过写脚本来实现了,刚刚在前面也提到了,NNUNet的官网已经提供了一些示例。我们就可以站在巨人的肩膀上,根据自己实际的数据结构进行适当的改动就可以了。
过程:
1、在图中的位置新建一个ConVertSelfData.py(文件名可以自由决定,建议按照官方代码的格式来命名)
2、在py文件中,写脚本完成上面说的两个任务,此处给我我自己写的代码作为参考:
import os.path
from batchgenerators.utilities.file_and_folder_operations import *
import shutil
from glob import glob
from nnunetv2.dataset_conversion.generate_dataset_json import generate_dataset_json
from nnunetv2.paths import nnUNet_raw
def convert_selfData(data_path:str, nnunet_dataset_id:int =101):
"""
转换自己的数据为nnUnet需要的数据格式,主要操作 将原本的数据文件末尾后加入通道数对应的四位数字:
例如原始的文件名为case1.nii.gz 如果只有单通道图像--->改变后为:case1_0000.nii.gz
然后将文件复制到nnUnet_Raw文件夹中。
最后按照一定的要求调用generate_dataset_json生成dataset.json 文件
"""
task_name = "Pancer_segmentation"
foldername= "Dataset%03.d_%s" % (nnunet_dataset_id,task_name) #此变量用于生成在nnUnet_raw保存的文件夹名称,格式按照官方要求为Dataset任务号_任务名称
# setting up nnU-Net folders
out_base = join(nnUNet_raw, foldername)
print(out_base)
imagestr = join(out_base, "imagesTr")
imagests = join(out_base, "imagesTs")
labelstr = join(out_base, "labelsTr")
maybe_mkdir_p(imagestr)
maybe_mkdir_p(imagests)
maybe_mkdir_p(labelstr)
### 大致的数据结构方式请参考医学十项全能的数据格式对数据进行存储和整理。
### 接下来的部分需要自己写代码遍历自己的数据,然后重命令文件,并按照imagesTr,imagesTs,labelsTr重新保存在nnUnet_raw的文件夹中
### imagesTr---训练集的输入图像,imagesTs-测试集的输入图像,labelsTr-训练集的标签图像。如果你的数据带有验证集,请把验证集的数据一并放到训练集中
### nnUnet采取的是使用交叉验证的方法对数据进行训练和筛选。且segmentation的图像的标签要为0,1,2,3
train_img_list =sorted(glob(os.path.join(data_path,"imagesTr/*.nii.gz")))
test_img_list = sorted(glob(os.path.join(data_path,"imagesTs/*.nii.gz")))
train_mask_list = sorted(glob(os.path.join(data_path,"labelsTr/*.nii.gz")))
# print(len(train_mask_list),len(test_img_list))
#
for i in range(len(train_img_list)):
tr = train_img_list[i].split('\\')[-1][:-7]
shutil.copy(train_img_list[i], join(imagestr, f'{tr}_0000.nii.gz'))
shutil.copy(train_mask_list[i], join(labelstr, f'{tr}.nii.gz'))
for i in range(len(test_img_list)):
tra = test_img_list[i].split('\\')[-1][:-7]
shutil.copy(test_img_list[i], join(imagests, f'{tra}_0000.nii.gz'))
label_dict = {"background": 0,"pancreas": 1}### 这个根据自己的数据定义标签 二分类的话形如:{"background": 0,"spleen": 1}
### 解释一下参数
"""
out_base:nnUnet_raw+数据任务文件夹地址
{0:"CT"} 代表的是图像的通道数,如果是单一图像来源 就只有一个通道,比如CT。对于MR有多个序列的,一个数字对应有一个序列:
如{0:"T1",1:"T2"} 如果是多个通道,那么上面迁移数据的时候 就要记得在对应通道后面的文件名上加上对应的编号,如case01_0000.nii.gz,
case01_0001.nii.gz
labels:标签对应的映射表
datasetname:数据集或者自己要分割任务的名称
overwrite_image_reader_writer:确定nnUnet数据读取的方式。如果是医疗图像 且为nii文件,不用修改。其他格式文件请参考官网dataformat部分
description:对数据集的描述,相当于自己写备忘录。
"""
generate_dataset_json(out_base, {0: "CT"}, labels=label_dict,
num_training_cases=len(train_img_list), file_ending='.nii.gz',
dataset_name=task_name, reference='Private data no reference ',
release='Private data no publice link',
overwrite_image_reader_writer='NibabelIOWithReorient',
description="This is a private dataset,don't provide data description.")
3、在文件末尾添加,然后给出你自己数据集的地址,以及一个任务ID号就可以愉快的完成数据转换了。转换成功后 便可以在nnUNet_raw文件夹中看到数据。
if __name__ == '__main__':
# convert_amos_task1(r"D:\amos22 (1)\amos22", 218)
convert_selfData(r"E:\Pytorch\DataProcess\胰腺炎\NNUNetRawNII",300)
关于NNUNet的配置和训练就可以按照我的上一篇博客开始愉快的训练等结果啦!