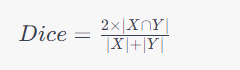目录
- 摘要
- 1 引言
- 2 方法
- 2.1 预处理和数据增强
- 2.2 网络的体系结构
- 2.3 训练过程
- 2.4 推理与后处理
- 3 实验与结果
- 4 结论与讨论
摘要
U-Net在各种医学图像分割挑战中取得了巨大成功。一些新的、带有花里胡哨功能的架构可能在某些数据集中在使用最佳超参数时取得成功,但它们的泛化性能无法保证。在这项工作中,我们专注于基本的U-Net架构,并提出了一种多尺度监督的3D U-Net,用于KiTS19挑战中的分割任务。为了提高性能,我们的工作可以总结为三个方面:首先,在解码器路径中使用多尺度监督,可以鼓励网络从深层预测正确的结果;其次,为了缓解肾脏和肿瘤样本不平衡的不良影响,我们采用了指数对数损失;第三,设计了一种基于连通组件的后处理方法,以去除明显错误的体素。在已发布的 KiTS19 训练数据集(共210名患者)中,我们将42名患者分为测试数据集,并最终获得了分别为0.969和0.805的肾脏和肿瘤的DICE分数。在挑战赛中,我们最终在106支团队中取得第7名,综合DICE分数为0.8961,即肾脏为0.9741,肿瘤为0.8181。
关键词:医学图像分割;3D U-Net;多尺度监督
1 引言
自动语义分割为探索肿瘤形态与其相应的手术结果之间的关系以及开发先进的手术规划技术提供了一个有前途的工具[1,2,3],但由于形态异质性,要实现良好的性能仍然具有挑战性。
KiTS19挑战[4]旨在加速可靠的肾脏和肾脏肿瘤语义分割方法的开发。它拥有300名独特的肾癌患者的CT扫描图像,其中有210张用于模型的训练和验证,另外的90名患者的扫描图像将保留作为测试数据集。提交模型的最终性能是基于肾脏和肿瘤分割的平均DICE系数来衡量的。
深度卷积神经网络(CNNs)已被公认为各种图像分类和分割任务的最先进方法。带有编码器-解码器架构的U-Net [5] 是医学图像分割的一个稳定成功的网络。由于体积数据在生物医学数据分析中比2D图像更丰富,因此为了充分利用像CT和MRI这样的3D图像的空间信息,提出了3D卷积并认为它更加有效。Fabian[6] 对基于3D U-Net [7] 进行了轻微修改(nnunet),并在许多医学图像分割挑战中获得了冠军或前几名,这证明了经过优化的U-Net具有比许多其他新架构更好性能的潜力。
受到[6]的启发,我们还放弃了一些常见的架构技巧,如残差块[8]、稠密块[9]、注意机制[10]、特征金字塔网络[11]和特征重校准[12]。在我们看来,医学图像远不如自然图像多样化,因此它们不需要太深的卷积层或太多的连接。只有5层的基本U-Net足以表示或学习用于分类的特征。
在遵循这些建议的基础上,我们将工作重点放在更好地训练3D U-Net并更有效地利用有限的训练数据集上。由于最终的全分辨率预测是从更深的低分辨率层上采样的,因此保证在深层次预测的准确性非常重要。因此,我们设计了多尺度监督的3D U-Net,以鼓励网络不仅在最后一层进行正确的预测,而且在每个分辨率级别都进行正确的预测,从而显著提高了最后一层的性能。为了减轻由于不平衡的类别数据带来的负面影响,我们使用了增强的焦点损失[13]和指数对数损失[14]。最后,我们的后处理方法会去除与肾脏未附着的分散的肾脏或肿瘤。
2 方法
在这一部分,我们将详细介绍我们的方法,不仅包括网络架构,还包括预处理、数据增强、训练过程、推断和后处理,因为这些因素对于实现3D U-Net应有的性能非常重要。
2.1 预处理和数据增强
为了去除可能来自某些金属物体的异常强度值,我们将CT图像的强度值截取到它们的0.5和99.5百分位数之间。然后按照惯例,我们使用全局前景均值和标准差进行数据归一化,这是由于典型的权重初始化方法。需要强调的是,3D数据的各向异性会破坏3D卷积的优势,因为它无法学习具有相同感受野的不同体素空间数据的统一表示。因此,如果数据不在同一体素空间中,我们会对其进行重新采样。
异常值去除:某些CT图像中可能包含异常强度值,这些异常值可能来自于金属物体的存在或其他因素。这些异常值可能会对后续的图像处理和分析造成干扰,因此需要将它们去除,以确保数据的质量和准确性。
数据范围限制:将强度值限制在0.5和99.5百分位数之间意味着去除了图像中最极端的1%强度值。这可以帮助确保大多数像素的强度值在一个相对较小的范围内,使数据更加一致和易于处理。
数据标准化:截取后,通常会对图像进行标准化,以使其具有零均值和单位方差。这有助于训练深度学习模型时,确保数据的尺度一致,从而更容易进行模型训练和收敛。
总之,通过将CT图像的强度值截取到0.5和99.5百分位数之间,可以去除异常值,限制数据范围,并为后续的数据处理和分析提供更稳定和一致的数据。这有助于提高图像处理和分析的准确性和可靠性。
处理3D数据(三维数据,如医学图像中的CT或MRI扫描)时的一个重要问题,即数据的各向异性问题。
"3D数据的各向异性"指的是数据在三维空间中不同方向上的分辨率和性质可能不同。这意味着,对于同一个3D图像,横向、纵向和纵深方向的信息可能有所不同。这种不同可能是由于扫描仪的特性、扫描过程中的图像畸变、采样间距不均匀等因素引起的。
"3D卷积的优势"指的是使用卷积神经网络(CNN)中的三维卷积操作来处理3D数据的潜在好处。3D卷积可以捕获3D图像中的空间信息,有助于更好地理解和分割体积性数据,比如医学图像中的器官或肿瘤。
然而,如果数据存在各向异性,即不同方向上的特征和分辨率差异较大,那么传统的3D卷积可能不够有效。因为它无法充分学习到不同方向上的特征,不能提供统一的表示。这可能会导致模型在某些方向上的性能不佳,因为模型无法有效地处理各向异性数据。
为了解决这个问题,一种方法是将数据进行等间距的重新采样,以使不同方向上的分辨率一致,从而允许3D卷积更好地学习统一的表示。这可以提高模型对各向异性数据的处理能力。所以,原文提到的"3D数据的各向异性会破坏3D卷积的优势" 意味着如果不处理各向异性,模型可能无法充分利用3D卷积的优势。
手动标注医学图像通常很繁琐,因此带标签的数据集通常有限。我们采用了强大的数据增强方法,以避免模型过拟合,包括随机旋转、随机缩放、随机弹性变形、伽马校正增强以及镜像等。
2.2 网络的体系结构
U-Net [5] 是一种经典的编码-解码分割网络,在近年来引起了很多关注。编码器路径类似于典型的分类网络,逐层提取更高层次的语义特征。然后,解码器路径恢复每个体素的本地化信息并利用特征信息对其进行分类。为了利用编码器中嵌入的位置信息,我们在同一阶段的层之间构建了直接连接。
我们的网络是基于3D U-Net [7] 设计的。该框架如图1所示。这个多尺度监督网络从解码器路径的不同层面进行预测,不同于传统的3D U-Net只从最后一层进行预测。这些分割结果将与相应分辨率的标签进行比较,然后用于计算最终的损失函数。这种监督鼓励网络从低分辨率特征图中进行正确的预测,这些特征图将被上采样为完整分辨率的特征图。
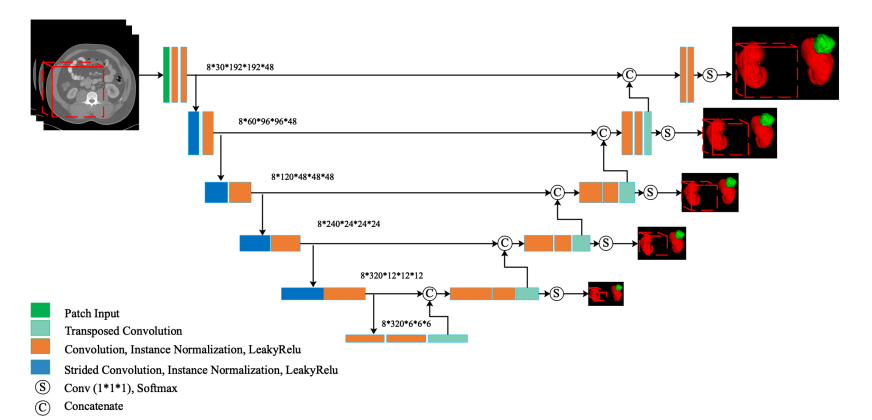
图1. 我们的多尺度监督3D U-Net的结构(最好在彩色中查看)。实际架构是3D的,但出于简化,我们在这里使用2D。根据我们的经验,我们采用步进卷积而不是池化操作,并用转置卷积代替三线性插值进行上采样。为了减小模型的体积,我们将基本特征数设置为30
- 步进卷积(strided convolution)是卷积神经网络(CNN)中的一种卷积操作。它与常规卷积操作相似,但在应用卷积核(滤波器)时,会跳过输入数据的一定数量的像素,以减小输出特征图的尺寸(常规卷积也就是stride=1的步进卷积)
- 转置卷积(Transpose Convolution),也称为反卷积(Deconvolution),是一种卷积神经网络(CNN)中的操作,用于将特征图的尺寸从较小的空间分辨率上采样到较大的空间分辨率。与标准卷积操作相反,转置卷积允许扩大特征图的尺寸,从而实现上采样操作
2.3 训练过程
由于GPU内存的限制,我们选择将 patch 大小设置为192×192×48,并在2个GPU上(Tesla,32GB)使用数据并行方式将 batch size 设置为8。patch 是随机抽样的,我们将一个epoch定义为250次迭代。我们使用Adam作为优化器。学习率初始化为3×10^-4,如果训练损失在30个epoch内没有进一步改善,学习率将减小0.2倍。
在CT图像中进行肾脏和肿瘤分割时,背景样本的数量远远超过肾脏和肿瘤体素的数量。此外,由于肿瘤形态的多样性,肿瘤更难分类。为了减轻这种不平衡,我们使用了指数对数损失[14]。这种损失强调了困难样本的影响,并通过使损失变得非线性,使它们具有更大的权重。同时,我们手动为背景、肾脏和肿瘤分配了不同的权重。
我们结合了Soft Dice和交叉熵来训练我们的模型。我们的最终损失格式可以总结如下:
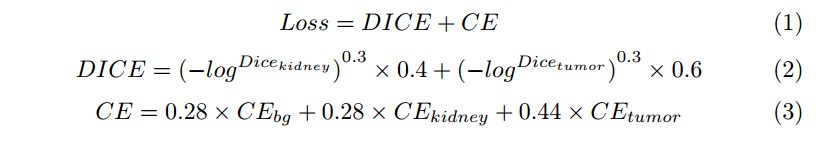
Soft Dice Coefficient 是 Dice Coefficient的变体,用于度量两个集合之间的相似性。在图像分割中,这两个集合通常分别表示真实分割结果和模型预测的分割结果。
Dice Coefficient(或F1 Score)通常定义为:
其中,X 是真实分割结果的像素集合,Y 是模型预测的分割结果的像素集合,|X ∩ Y| 表示两者的交集的像素数量,|X| 和 |Y|分别表示它们的像素数量。
在 Soft Dice 中,与传统的 Dice Coefficient不同,它将真实分割结果和模型预测的分割结果的像素值视为连续值(soft values),而不是二进制(0或1)。这允许 Soft Dice在像素级别度量两个分割结果的相似性,而不仅仅是匹配或不匹配。
通常,Soft Dice 范围在0到1之间,其中1表示完美的重叠,0表示没有重叠。模型的目标是最大化 Soft Dice 分数,以获得更好的分割结果。这使 Soft Dice成为一种重要的评估指标,尤其是在医学图像分割中,其中精确的分割结果对于诊断和治疗规划至关重要。
2.4 推理与后处理
在进行病例预测时,我们采用滑动窗口方法,使预测之间存在重叠。为提高准确性,我们将原始数据和镜像数据的预测结果进行组合。
一些常见的人类知识可以帮助进一步提高分割模型的性能。例如,一个病人最多只有两个肾脏,而肿瘤应该紧贴在肾脏上。因此,我们设计了一个基于连通组件的简单后处理方法来移除明显错误的预测。其效果如图2所示。
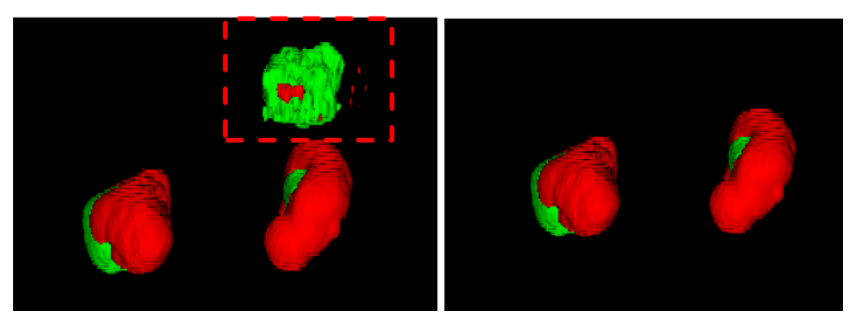
图2. 我们后处理方法的效果。左图是经过后处理之前的图像,虚线框中的区域包含一些明显错误的体素。右图是经过后处理之后的图像,额外的体素已经被移除。
3 实验与结果
共有210例患者的CT扫描数据用于训练模型。我们将其中的42例划分为测试数据集,使用其他图像来训练我们的模型。训练过程耗时约5天,运行在2块GPU上(Tesla 32GB)。训练期间的损失曲线如图3所示。可以观察到我们提出的损失持续稳定下降,这个过程大约持续了700个时期,直到学习率达到我们忍耐的极限。
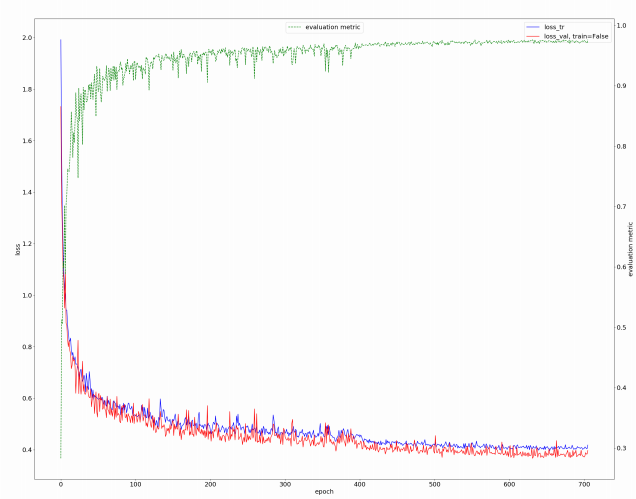
图3. 训练过程中损失的变化情况。红线和蓝线分别表示验证损失和训练损失。绿线是一个滑动验证损失度量,用于选择最佳的检查点。
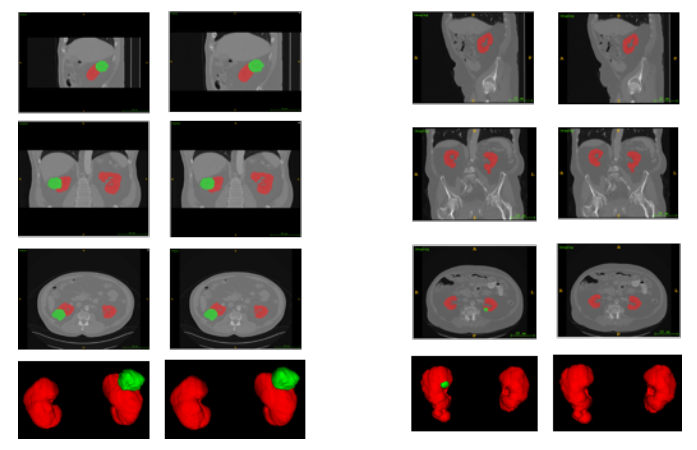
图4. 我们的分割输出的两个示例。从上到下的行分别为2D中的矢状面、冠状面、横断面和3D视图(最佳在彩色视图中查看)。从左到右的列分别是一个常见案例的真实情况和预测情况,以及最糟糕情况。
我们的分割结果样本如图4所示。我们观察了来自不同视图的2D切片和3D视图以分析性能。显然,在CT图像中,肾脏的分割效果非常好。预测结果与真实情况非常接近。然而,一些肿瘤太小以致于难以获得良好的Dice系数,并且也难以找到。一些具有小肿瘤大小的病例会显著降低平均Dice系数。
我们的测试数据集的箱线图如图5所示。测试数据集的平均Dice系数分别为0.969和0.805,用于肾脏和肿瘤的分割。此外,肾脏的方差非常小,表明我们的算法在肾脏分割方面非常稳定。
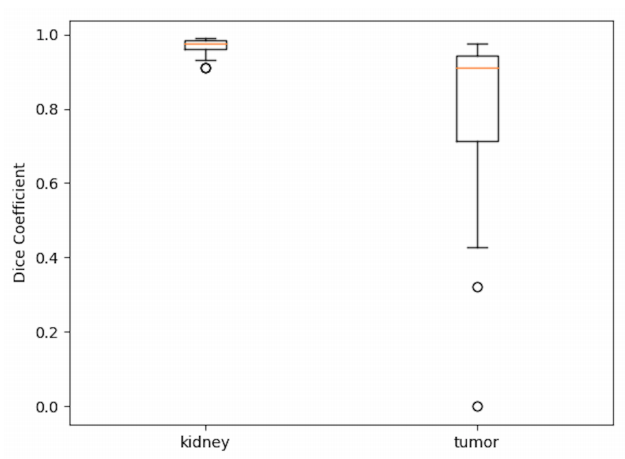
图5. 划分测试数据集中42名患者的分割结果的箱线图。
4 结论与讨论
在本论文中,我们展示了我们的模型及其在KiTS19数据集中的性能。我们的方法基于经典的3D U-Net,并通过多尺度监督、指数对数损失和基于连通组件的后处理进行了增强。在已发布的数据上测试,我们的方法分别实现了肾脏和肿瘤的平均Dice系数为0.969和0.805。
在挑战中,我们获得了综合Dice系数为0.8961,其中肾脏的Dice系数为0.9741,肿瘤为0.8181,排名在所有106支队伍中的第7位。尽管肾脏分割的Dice系数略低于最高0.9743,但肿瘤分割的Dice系数超过了顶尖团队的表现。由于我们遵循了Fabian [6]的精神,我们在这里没有使用太多架构技巧,而是侧重于训练过程。肿瘤由于其严重的形态异质性,总是难以进行良好的分割。在未来,我们计划尝试肿瘤分割的两阶段方法,即首先提出感兴趣区域,然后使用可变形卷积而不是传统卷积来适应肿瘤特征。