目录
一、问题描述
二、解决方法
1.模拟退火
1.1 算法思路
1.2 求解代码
1.3 计算结果
2.粒子群算法
2.1 算法思路
2.2 求解代码
2.3 计算结果
3.遗传算法
3.1 算法思路
3.2 求解代码
3.3 计算结果
一、问题描述
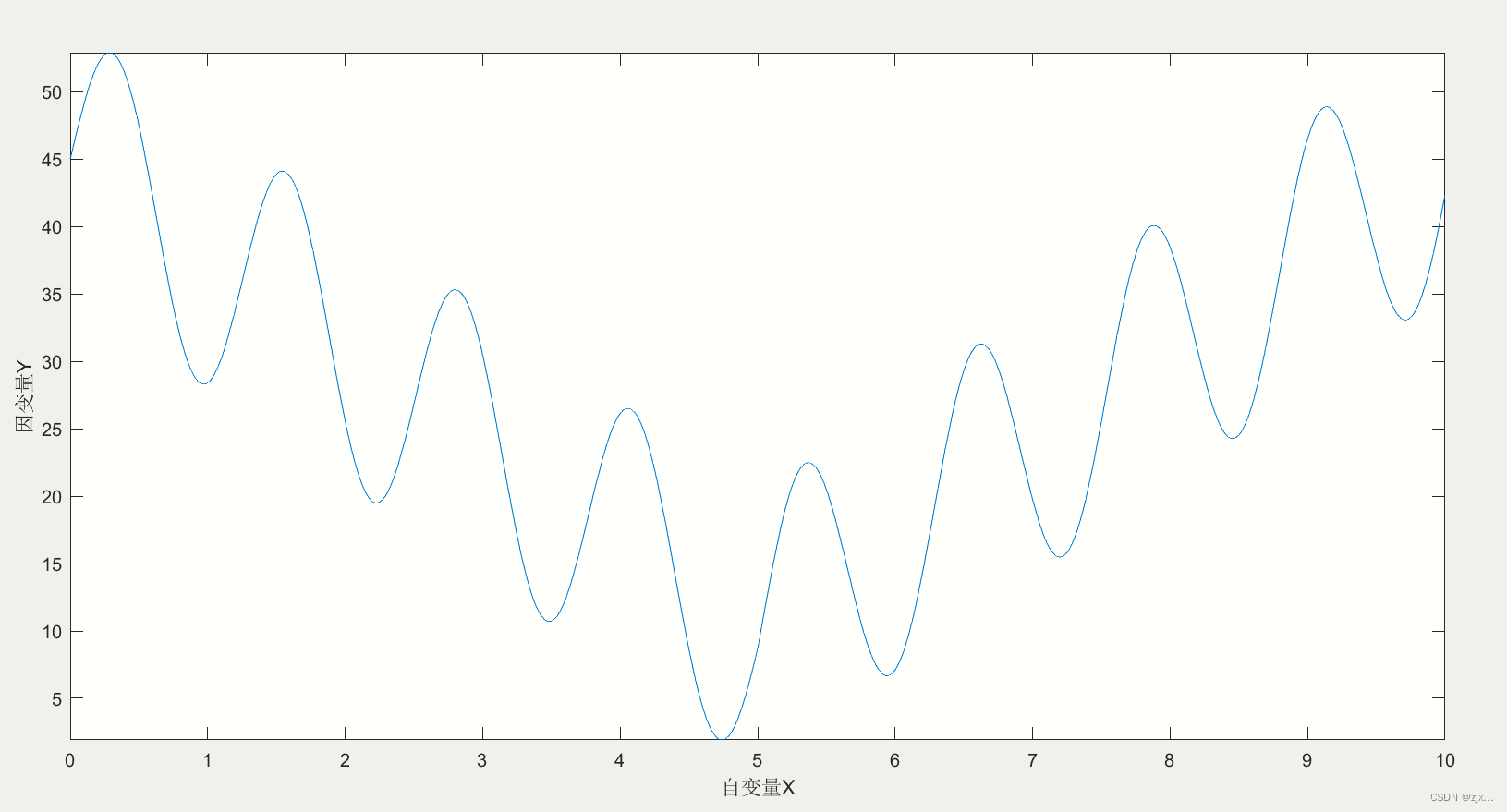
本篇文章所做的是分别用模拟退火、粒子群算法、遗传算法求解一元函数
这一函数在0-10区间的最大值问题,其函数图像为:
二、解决方法
1.模拟退火
1.1 算法思路
- 首先,定义目标函数 f,该函数以一个变量 x 为输入,并返回一个数值。然后使用 fplot 函数绘制了这个函数在取值范围(即 0 - 10)之间的图像。
- 接着,定义了初始温度 T,结束温度 T_final,以及降温系数 alpha。
- 设定了自变量的取值范围 xlimit,在取值范围内随机选择一个值来定义初始解 init_x。
- 定义了最大迭代次数 max_iterations。
- 初始化了最优解 best_x 和最优解的函数值 best_f,它们的初始值设为初始解及其函数值。
- 开始模拟退火算法的主要循环,迭代次数从1到最大迭代次数。
- 在每次迭代中,在初始解上添加一个随机噪声得到一个新的解 new_x,并将其将新解限制在取值范围内。
- 计算新解与当前解的函数值的差值 delta_f。
- 如果新解的函数值比当前最优解的函数值更好(即 delta_f 大于0),或者满足Metropolis准则(即以一定的概率接受比当前最优解更差的解),那么接受新解作为当前解。
- 如果新解比当前最优解更好,更新最优解。
- 每次迭代后降低温度,如果温度已经低于结束温度,结束算法,否则继续迭代。
1.2 求解代码
function SA()
% 定义目标函数,并展示图像
f = @(x)10.*sin(5.*x)+7.*abs(x-5)+10;
figure(1);
fplot(f,[0,10]);
hold on
xlabel('自变量X');
ylabel('因变量Y');
% 定义初始温度和结束温度与降温系数
T = 1000;
T_final = 1e-6;
alpha = 0.99;
% 定义取值范围
xlimit = [0,10];
% 定义初始温度下的初始解
init_x = xlimit(1, 1) + (xlimit(1, 2) - xlimit(1, 1)) * rand();
% 定义最大迭代次数
max_iterations = 10000;
% 初始化最优解和最优解的函数值
best_x = init_x;
best_f = f(init_x);
% 开始模拟退火算法
for iter = 1:max_iterations
% 生成新解
new_x = init_x + randn;
% 控制解的范围在给定区间内
new_x = max(min(new_x, xlimit(2)), xlimit(1));
% 计算目标函数值的差值
delta_f = f(new_x) - f(init_x);
% 如果新解更好,或者满足Metropolis准则,则接受新解
if delta_f > 0 || exp(-delta_f/T) > rand()
init_x = new_x;
if f(new_x) > best_f
best_x = new_x;
best_f = f(new_x);
end
end
% 展示当前最优解的情况
plot(best_x,best_f,'b*','MarkerSize',5);
title(strcat('当前迭代次数:',num2str(iter)));
pause(0.03);
% 降温
T = T* alpha^(iter/100);
% 如果温度足够低,结束算法
if T < T_final
break;
end
end
% 展示最终的结果
plot(best_x,best_f,'r*','MarkerSize',10);
grid on;
hold off;
fprintf(['The best x is --->\t',num2str(best_x),'\nThe fitness value is --->\t',num2str(best_f),'\n'])
end
1.3 计算结果
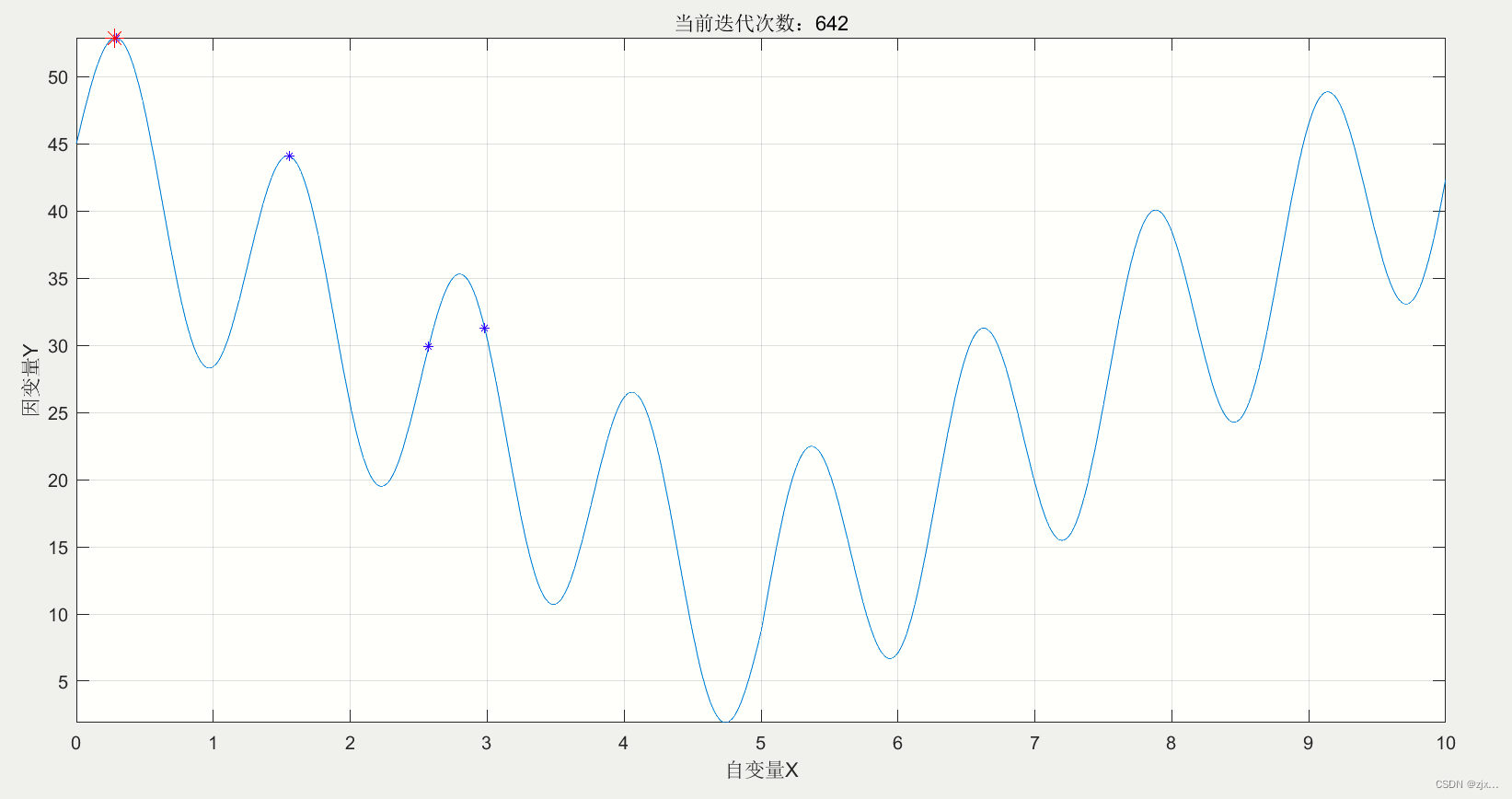
The best x is ---> 0.28093
The fitness value is ---> 52.8958
2.粒子群算法
2.1 算法思路
- 首先定义了种群个数、迭代次数、惯性权重、可信度因子以及允许的精度误差。
- 然后定义位置与速度的限制范围。
- 接着,初始化了种群位置(x)、种群进化速度(v)、个体历史最佳位置(individual_best_x)、个体历史最佳适应度(individual_best_fitness)、种群历史最佳位置(global_best_x)、种群历史最佳适应度(global_best_fintness)等参数。
- 进行粒子群算法的优化迭代。
- 记录当前的种群历史最佳适应度(old_global_best_x)。
- 然后计算每个粒子的适应度存储到 fitness 中。
- 更新个体最佳适应度与位置、种群最佳适应度与位置。
- 更新速度,公式为:
其中
v:粒子的速度向量pBest:粒子自身历史最优位置present:当前粒子的位置向量gBest:全局最优位置c1和c2:学习因子rand():[0, 1)之间的随机数 - 限定速度范围,更新位置,限定位置范围。
- 判断迭代的历史最佳适应度和位置不会有太大的精度变化,若变化小于精度值则停止迭代。
2.2 求解代码
function PSO()
N = 50; % 初始种群个数
iterations = 200; % 迭代的次数
w = 0.8; % 惯性权重
c1 = 0.5; % 可信度因子1
c2 = 0.5; % 可信度因子2
precision = 0.001; % 允许的精度误差
% 展示函数图像
figure(1);
fplot(@(x)10.*sin(5.*x)+7.*abs(x-5)+10,[0,10]);
hold on
xlabel('自变量X');
ylabel('因变量Y');
xlimit = [0,10]; % 设置位置参数限制
vlimit = [-1,1]; % 设置速度限制
% 初始化种群位置
x = xlimit(1, 1) + (xlimit(1, 2) - xlimit(1, 1)) * rand(1,N);
% 初始化种群速度
v = rand(1,N);
% 每个个体的历史最佳位置初始化
individual_best_x = x;
% 每个个体的历史最佳适应度初始化
individual_best_fitness = zeros(1,N);
for i = 1:N
individual_best_fitness(i) = objective_function(x(i));
end
% 种群的最佳历史位置初始化
global_best_x = 0 ;
% 种群最佳历史适应度初始化
global_best_fintness = -inf;
iter = 1;
while iter <= iterations
% 将历史最佳位置进行copy
old_global_best_x = global_best_x;
% 计算每个粒子适应度,并记录在 fitness 中
fitness = zeros(1,N);
for i = 1:N
fitness(i) = objective_function(x(i));
end
% 遍历每个粒子,如果该粒子的当前适应度大于其历史最佳适应度,则将该粒子的当前适应度作为历史最佳适应度,并将历史最佳位置赋值为当前位置。
for i = 1:N
if individual_best_fitness(i) < fitness(i)
individual_best_fitness(i) = fitness(i);
individual_best_x(i) = x(i);
end
end
% 如果所有粒子中的最大适应度已经大于了全局历史最佳适应度,则将最大适应度最为全局历史最佳适应度,并记录位置
if global_best_fintness < max(individual_best_fitness)
[global_best_fintness, idx] = max(individual_best_fitness); % idx 为最佳适应度的位置标号
global_best_x = individual_best_x(idx);
end
% 更新速度
v = v .* w + c1 .* rand(1,N) .* (individual_best_x - x) + c2 .* rand(1,N) .* (repmat(global_best_x, 1, N) - x);
% 将速度调整在限定范围内
for i =1:size(v,2)
v(i) = max(min(v(i), vlimit(2)), vlimit(1));
end
% 更新位置
x = x + v;
% 将位置调整在限定范围内
for i =1:size(x,2)
x(i) = max(min(x(i), xlimit(2)), xlimit(1));
end
% 如果迭代的历史最佳适应度和位置不会有太大的精度变化就停止
if iter > 100
if abs(global_best_x - old_global_best_x) < precision
break
end
end
% 展示每一次迭代中的最优解
plot(global_best_x,global_best_fintness,'b*','MarkerSize',5);
title(strcat('当前迭代次数:',num2str(iter)));
pause(0.03);
iter = iter + 1;
end
% 展示最终的结果
plot(global_best_x,global_best_fintness,'r*','MarkerSize',10);
grid on;
hold off;
fprintf(['The best x is --->\t',num2str(global_best_x),'\nThe fitness value is --->',num2str(global_best_fintness),'\n'])
end
% 定义目标函数
function fitness=objective_function(decimal_value)
fitness=10*sin(5*decimal_value)+7*abs(decimal_value-5)+10;
end2.3 计算结果
The best x is ---> 0.28607
The fitness value is --->52.899
3.遗传算法
3.1 算法思路
- 初始化种群、染色体、交叉率、变异率、迭代次数等一系列参数
- 遗传算法循环迭代
- 将二进制编码转化为十进制数值,即将二进制字符串转化为实际的自变量值。
- 计算每个个体的适应度,即通过目标函数对应的数学计算,求得每个个体适应度值。
- 选择操作,根据适应度值进行轮盘赌选择,选择出新的种群。
- 交叉操作,采用单点交叉的方式对新种群进行交叉,生成新的子代个体。
- 变异操作,以一定的概率对新种群进行变异,引入新的个体差异性。
- 更新种群,计算新种群的适应度,记录最佳适应个体和适应度值,并绘制当前最佳个体的散点图。
- 重复以上步骤,直到达到最大迭代次数。
3.2 求解代码
function GA()
figure(1);
fplot(@(x)10.*sin(5.*x)+7.*abs(x-5)+10,[0,10]);
hold on
xlabel('自变量X');
ylabel('因变量Y');
population_size=20; % 种群大小
chromosome_length=20; % 染色体长度
crossover_possibility=0.7; % 交叉率
mutation_possibility=0.1; % 变异率
max_generations=200; % 最大迭代次数
bestfitness=-inf; % 最佳适应度
bestindividual=0; % 最佳适应个体
result=zeros(2,max_generations); % 每次迭代的最佳适应个体与适应度
xlimit=[0,10];
% 初始化种群
population=round(rand(population_size,chromosome_length)); % 生成 population_size 个长度为 chromosome_length 字符串
% 循环迭代
for iter=1:max_generations
% 1.二进制转化为十进制
decimal_value = transcoding(population,xlimit,chromosome_length);
% 2.计算函数目标值
fitvalue=objective_function(decimal_value);
% 3.选择操作(轮盘赌法)
new_population=select(population,fitvalue);
% 4.交叉操作(单点交叉)
new_population = crossover(new_population,crossover_possibility);
% 5.变异操作
new_population = mutation(new_population,mutation_possibility);
% 更新种群,重新计算种群的适应度,记录Best
population=new_population;
decimal_value = transcoding(population,xlimit,chromosome_length);
fitvalue=objective_function(decimal_value);
[row,~]=size(population);
for i=1:row
if bestfitness<fitvalue(i)
bestfitness=fitvalue(i);
bestindividual=decimal_value(i);
end
end
result(1,iter)=bestindividual;
result(2,iter)=bestfitness;
plot(result(1,iter),result(2,iter),'b*','MarkerSize',5);
title(strcat('当前迭代次数:',num2str(iter)));
pause(0.03);
end
plot(result(1,end),result(2,end),'r*','MarkerSize',10);
grid on;
hold off;
fprintf(['The best x is --->\t',num2str(bestindividual),'\nThe fitness value is --->',num2str(bestfitness),'\n'])
end
% 定义目标函数
function fitness=objective_function(decimal_value)
fitness=10*sin(5*decimal_value)+7*abs(decimal_value-5)+10;
end
% 选择
function new_population=select(population,fitvalue)
[row,col]=size(population);
new_population=zeros(row,col);
fitvalue_possibility=fitvalue/sum(fitvalue); % 适应度与所有适应度的之和的比
fitvalue_possibility=cumsum(fitvalue_possibility); % 离散的概率转为0-1区间的累计和
p_rand = sort(rand(row,1)); % 一列由小到大排列的随机数
% 用p_rand去与做完了累计和的p_fitvalue作比较
% 适应度越高就有越大的机率在p_fitvalue的累计和中占据更大的区间
% 将会有较多的p_rand值落在区间
% 用这种方法保留了适应度较大的基因
i=1;
j=1;
while i <= row
if p_rand(i)<fitvalue_possibility(j)
new_population(i,:)=population(j,:);
i=i+1;
else
j=j+1;
end
end
end
% 交叉
function new_population=crossover(population,crossover_possibility)
[row,col]=size(population);
new_population=zeros(row,col);
for i=1:2:row-1 % 让i与i+1进行交叉,所以i遍历染色体中的编号为奇数就可以
if rand<crossover_possibility
point=round(rand*col); % 在py位二进制中随机选一个交叉点位
new_population(i,:)=[population(i,1:point),population(i+1,point+1:end)];
new_population(i+1,:)=[population(i+1,1:point),population(i,point+1:end)];
else
% 有一定几率不发生交叉
new_population(i,:)=population(i,:);
new_population(i+1,:)=population(i+1,:);
end
end
end
% 变异
function new_population=mutation(population,mutation_possibility)
[row,col]=size(population);
new_population=zeros(row,col);
for i=1:row
if rand<mutation_possibility % 发生变异
mutation_point=round(rand*col); % 发生变异时的位点
if mutation_point<=0
mutation_point=1; % 检测越界
end
new_population(i,:)=population(i,:); % 变异后的种群存储在newpop3中
if new_population(i,mutation_point)==0 % 将变异点位上的二进制数取反
new_population(i,mutation_point)=1;
else
new_population(i,mutation_point)=0;
end
else % 没有发生变异
new_population(i,:)=population(i,:);
end
end
end
% 编码转换
function decimal_value = transcoding(population,xlimit,chromosome_length)
[row,col]=size(population); % 种群的行列数
convert=zeros(row,col);
for j=1 : col
convert(:,j)=population(:,j)*2^(col-j); % 将二进制对应为 1 的部分赋值为相应的数值
end
temp=sum(convert,2); % 将这些数值加起来转换为10进制
decimal_value=xlimit(1)+temp*((xlimit(2)-xlimit(1))/2^chromosome_length); % 将这些数据转化为定义的上下区间内的值
end
3.3 计算结果
The best x is ---> 0.2987
The fitness value is --->52.8792