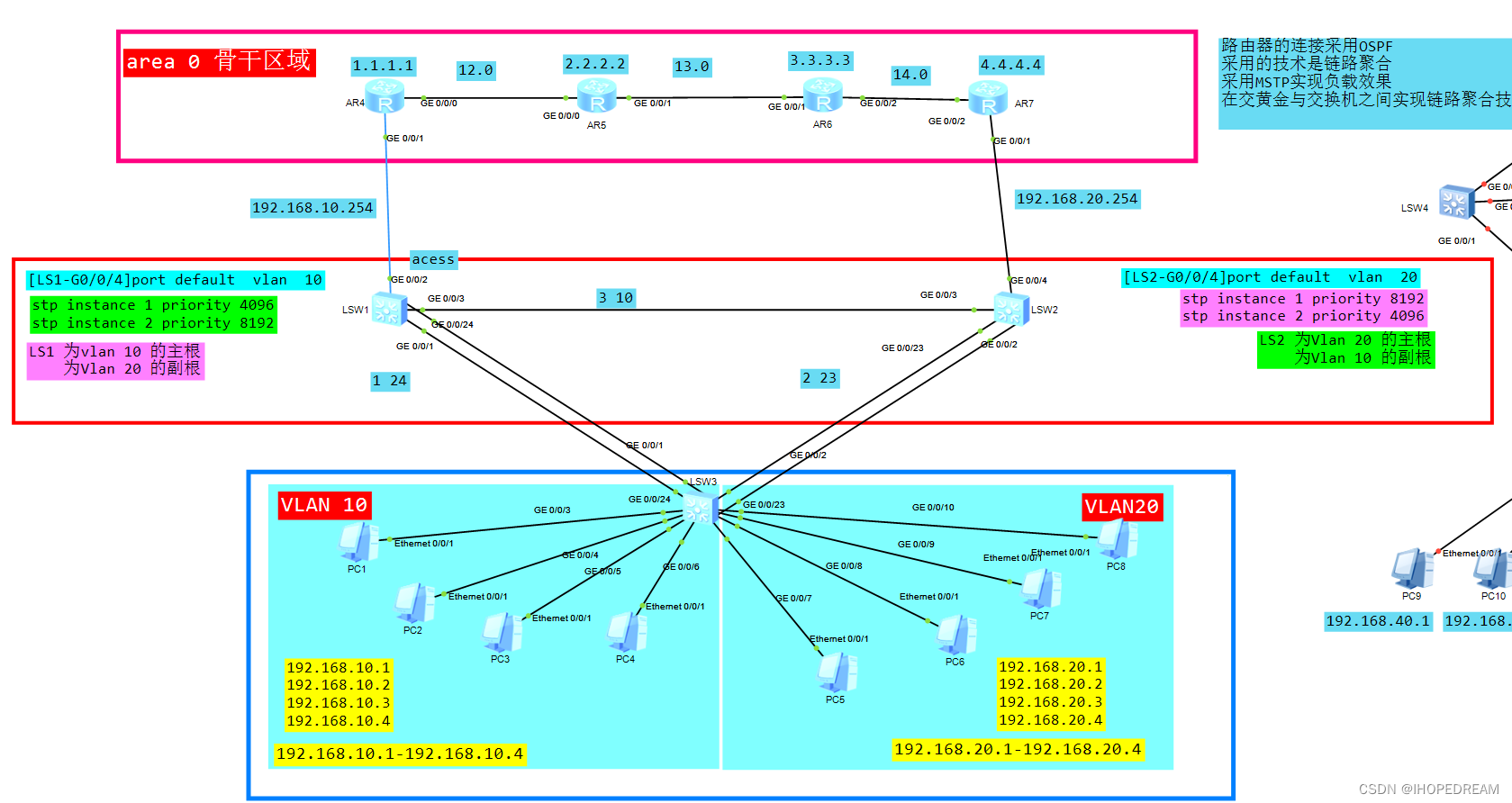
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
哺乳动物的损伤皮肤屏障完整性恢复通过创面愈合基本机制实现,这是一个包括凝血、炎症、再上皮化(re-epithelialization)、肉芽组织形成和疤痕重塑的多步骤过程。再上皮化是决定创面成功愈合的重要因素,再上皮化受损是创伤、糖尿病、血管疾病、感染或辐射导致的所有慢性创面无法愈合的原因。因此研究创面再上皮化调控机制具有重要的临床意义,并可能为加速创面愈合的治疗策略提供新的见解。
N6-甲基腺苷(m6A)是真核生物中最普遍的内部mRNA修饰,哺乳动物m6A修饰受“writers”(如METTL3、METTL14、WTAP)和“erasers”(如FTO、ALKBH5))的动态和可逆调控。同时m6A修饰可以被特异性RNA结合蛋白识别(称为“readers”,如YTHDF1-3、HNRNP、IGF2BP)。m6A修饰被“readers”蛋白识别后,可以影响RNA代谢的各个方面,包括RNA稳定性、翻译、剪接、易位和高级结构。近年来大量研究表明m6A修饰广泛参与毛囊形态发生、紫外线介导的皮肤损伤、系统性硬化、寻常型银屑病、增生性瘢痕和肿瘤发生的调控。同时,m6A修饰在创面愈合过程中也发挥着重要作用,然而m6A修饰是否在调控角质形成细胞的迁移能力中发挥作用仍不清楚。此外对m6A修饰在创面再上皮化中的作用缺乏全面了解。
2023年7月14日,上海交通大学医学院附属第九人民医院整复外科Xin Huang、Yixuan Zhao、湖南中医药大学附属第二医院创面修复科刘代明为共同第一作者,上海交通大学医学院附属第九人民医院整复外科昝涛、李青峰、Yixuan Zhao为共同通讯作者, 以“ALKBH5-mediated m6A demethylation fuels cutaneous wound re-epithelialization by enhancing PELI2 mRNA stability”为题在《Inflammation and Regeneration》杂志发表研究论文,该研究通过甲基化RNA免疫沉淀测序(MeRIP-seq)和对应的RNA-seq联合分析揭示了ALKBH5是创面再上皮化的内源性促进因子,提示了开发针对难治性创面的重编程m6A靶向疗法的前景。

标题:ALKBH5-mediated m6A demethylation fuels cutaneous wound re-epithelialization by enhancing PELI2 mRNA stability(ALKBH5介导的m6A去甲基化通过增强 PELI2 mRNA 稳定性促进皮肤创面再上皮化)
时间:2023-07-14
期刊:Inflammation and Regeneration
影响因子:IF 8.1
技术平台:m6A-seq(MeRIP-seq)、RNA-seq、m6A-RIP-qPCR 、qRT-PCR、Dot-blot、WB/IF等
研究摘要:
创面再上皮化受损会导致皮肤屏障重建功能障碍。m6A RNA修饰参与RNA命运决定,m6A甲基化变异会触发许多疾病发病机制。然而,m6A在创面再上皮化中的作用仍然未知。本研究构建ALKBH5-/-小鼠以研究ALKBH5敲除后创面再上皮化率。采用甲基化RNA免疫沉淀测序(MeRIP-seq)和RNA-seq结合的高通量测序分析方法鉴定出ALKBH5的下游靶标。通过体外和体内挽救实验(In vitro and in vivo rescue experiments)验证下游靶标对ALKBH5缺失细胞或动物的功能表型作用。此外,通过RIP-qPCR、RNA下拉和RNA稳定性分析揭示了互作的reader蛋白和调控机制。
研究结果表明,ALKBH5在创缘表皮中特异性上调。ALKBH5敲除抑制了ALKBH5-/-小鼠的角质形成细胞(keratinocytes)迁移,并导致创面再上皮化延迟。综合高通量分析揭示E3泛素蛋白连接酶PELI2是ALKBH5的下游靶标。体外和体内挽救实验结果表明,外源性PELI2补充可以在体内和体外部分挽救ALKBH5缺失细胞中的角质形成细胞迁移并加速再上皮化。从机制上来说,ALKBH5通过去除PELI2 mRNA中的m6A修饰来促进PELI2表达,并以YTHDF2依赖的方式增强其稳定性。

图形摘要
研究结果
(1)在创缘的角质形成细胞(keratinocytes)中观察到RNA m6A修饰减少和ALKBH5表达增加

图1:健康创面边缘角质形成细胞中RNA m6A修饰降低,ALKBH5表达增加。
- 在小鼠背部产生直径5 mm的全层皮肤创面,切除肉脂膜下方筋膜(红虚线)以防止创面收缩。
- 伤后2 d (PWD2)皮肤创面大体外观及组织学。勾画创面床、创缘及邻近正常皮肤。比例尺:250 μm。
- m6A-DB分析从创面边缘皮肤和邻近正常皮肤提取mRNA的整体m6A水平。亚甲基蓝(Methylene blue)染色法检测总RNA(作为loading control)。实验重复三次。t检验,双侧*P<0.05。
d.创面边缘和正常皮肤m6A“writer”和“eraser”基因表达的热图。实验设4个生物重复。
e.RNA-seq数据(GSE159939)绘制的m6A“writer”和“eraser”基因在创床和创缘表皮和真皮中的表达热图。
f. IF检测创缘皮肤ALKBH5表达。比例尺:100 μm。
g.统计分析每个HPF中ALKBH5角质形成细胞的百分比。取4只小鼠创面样本,ALKBH5细胞百分比以均数±SD表示。t检验,双侧***P<0.001。
h.代表性临床照片,显示了一组以愈合或难治性不愈合创面为特征的慢性创面患者队列。
- IF检测愈合创面、难愈创面及正常皮肤创面边缘ALKBH5的表达。比例尺:100 μm。
j.统计分析每个HPF中ALKBH5表皮细胞的百分比。分析4例愈合创面、6例难愈创面的创缘及相应的正常皮肤对照。ALKBH5细胞的百分比表示为平均值±SD。单因素方差分析,***P < 0.001。Epi表皮。虚线表示表皮边界++++
(2)ALKBH5抑制降低角质形成细胞的迁移

图2:抑制ALKBH5可在体外破坏角质形成细胞迁移。
a-b. 通过qRT-PCR (a)和WB (b)验证,siRNA转染后,HaCaT细胞中的ALKBH5表达降低。实验重复3次。单因素方差分析 ***P < 0.001。
c. 从ALKBH5敲低组和对照组的HaCaT细胞中提取RNA,通过m6A-DB检测siRNA转染后的整体m6A水平。亚甲基蓝染色法检测总RNA(作为对照)。
d. Transwell实验分析ALKBH5敲低组和对照组HaCaT细胞的迁移能力。比例尺:25 μm。
e. Transwell实验的统计分析。所有实验重复3次,共纳入5个随机字段进行分析。迁移细胞面积的百分比用平均值±SD表示。单因素方差分析,***P < 0.001。
f. 采用细胞跟踪成像技术绘制ALKBH5敲低或对照HaCaT细胞划痕实验后0 h、2 h、16 h、32 h的代表性动态细胞速度图。x方向速度为u, y方向速度为v。速度大小表示u和v的合成速度,速度的方向和大小分别用黑色箭头和颜色密度表示。FC相位对比图像。比例尺:250 μm。
g. 不同时间点ALKBH5敲低组和对照组HaCaT细胞的速度大小的统计分析。
h. 统计分析划痕创面愈合率。所有实验重复三次。
- 通过划痕实验评估HaCaT细胞的长期迁移能力。比例尺:100 μm。
j. 创面愈合实验的统计学分析。所有实验重复3次,每个时间点纳入3个随机字段进行分析。单因素方差分析,***P < 0.001
(3)ALKBH5敲除可抑制小鼠创面再上皮化

图3:ALKBH5缺失会抑制小鼠创面的再上皮化。
a .使用CRISPR/Cas9技术敲除ALKBH5基因的策略示意图。
b-c. WB (b)和IF (c)分别证实ALKBH5-/-小鼠皮肤和表皮中ALKBH5表达缺失。
d. m6A-DB分析WT和ALKBH5-/-小鼠创面边缘皮肤的整体m6A水平。亚甲基蓝染色法检测总RNA(作为装载对照)。实验重复三次,m6A修饰的相对含量以平均值±SD表示。t检验,双侧*P<0.05。
e. IF分析揭示ALKBH5 -/-小鼠创面边缘表皮中ALKBH5表达缺失。比例尺:100 μm。虚线表示表皮边界。Epi表皮。
f-g. WT和ALKBH5-/-小鼠在PWD0、PWD2、PWD4、PWD6、PWD8上的皮肤创面代表性图像(f)。比例尺:2 mm。黑色虚线圆描绘了5mm宽的原始创面。创面闭合率(g)通过ImageJ软件进行量化,并表示为不愈合区域的百分比。6只小鼠的12个创面纳入分析。创面闭合的相对百分比以平均值±SD表示。t检验,双侧**P<0.01, ***P<0.001。
h-i. PWD8时WT和ALKBH5-/-小鼠创面再上皮化的组织学分析(h)。箭头线表示原创面间隙宽度为5 mm。钝端线描绘上皮间隙,代表非上皮化的创面区域。比例尺:1mm。在PWD8时对WT和ALKBH5-/-小鼠的再上皮化百分比进行定量分析(i)。6只小鼠的12个创面纳入分析。再上皮化创面面积的相对百分比以均数±SD表示。Mann-Whitney检验,双侧****P< 0.0001。比例尺:1mm。虚线表示表皮边界
(4)全基因组MeRIP-seq和RNA-seq鉴定出PELI2是ALKBH5的下游靶标

图4:在人表皮样本和角质形成细胞系中受ALKBH5调控的m6A修饰基因表征
- m6A修饰下游分析流程图。MeRIP-seq鉴定出人类表皮样本和角质形成细胞系共有的3710个基因,在3'UTR中具有特异性m6A peaks。RNA-seq在 ALKBH5敲低后鉴定出306个下调基因;以|FC|<0.5和P <0.05定义为差异表达基因。FC fold change。最终鉴定出16个同时具有m6A peaks和差异表达候选基因作为ALKBH5的潜在下游靶点。
- 3710个共有基因的GO富集分析图谱。
- 由IPA产生的PELI2和其他15个候选基因的调控网络。未显示C3orf33、PGBD2、ZNF20、ZNF573、ZNF577和ZNF785基因,因为它们与PELI2没有通路连接。
- qRT-PCR检测ALKBH5敲低后16个候选基因的表达。实验重复3次,mRNA的相对表达量以平均值±SD表示。单因素方差分析,*P<0.05;** p<0.01。
- IGV轨迹显示人表皮和HaCaT中PELI2的MeRIP-seq reads覆盖。
- m6A-RIP-qPCR检测证实PELI2转录本的m6A修饰。实验重复三次。将抗m6A抗体组和IgG组的mRNA相对表达量进行比较,以均数±SD表示。单因素方差分析,**P<0.01
(5) ALKBH5调控m6A甲基化和PELI2表达

图5:ALKBH5表达水平与PELI2表达水平呈正相关。
- 根据ALKBH5敲低或对照HaCaT细胞的RNA-seq分析,IGV追踪PELI2表达。实验设置两个重复。
- WB敲低ALKBH5后,检测HaCaT细胞PELI2蛋白表达。
- 通过IF染色分析PWD8中的WT和ALKBH5-/-小鼠创缘皮肤PELI2表达。虚线表示表皮边界。比例尺:100 μm。
- qRT-PCR显示PELI2在WT和ALKBH5-/-小鼠原代角质形成细胞中的mRNA表达。实验重复3次,mRNA相对表达量以平均值±SD表示。t检验,双侧**P<0.01。
- 采用IF染色分析PWD6时WT小鼠创缘表皮PELI2和ALKBH5的表达及共定位情况。比例尺:100 μm。
- IGV轨迹显示ALKBH5敲低后PELI2的MeRIP-seq reads覆盖情况。实验设置2个重复。
- 通过基因特异性m6A-RIP-qPCR检测HACAT细胞中ALKBH5敲低后PELI2转录本的m6A修饰增加。实验重复3次,将每组mRNA的相对表达量与input值进行比较,以平均值±SD表示。单因素方差分析,ns不显著,*P<0.05,****P<0.0001
(6)外源性PELI2过表达可挽救ALKBH5缺失后延迟的创面再上皮化

图6:外源性PELI2过表达挽救了ALKBH5敲低对角质形成细胞迁移的影响。
a-b. qRT-PCR显示在用ALKBH5或NC siRNA处理并用PELI2或NC质粒转染48h后的HaCaT细胞中PELI2(a)和ALKBH5(b)的mRNA表达。实验重复3次,mRNA的相对表达量以平均值±SD表示。单因素方差分析,ns不显著,*P<0.05,****P<0.0001。
c. WB检测ALKBH5和PELI2的蛋白表达。
d. Transwell实验显示外源性PELI2过表达对ALKBH5敲低或对照HaCaT细胞迁移的影响。比例尺:25 μm。
e. 对Transwell实验结果进行统计学分析。所有实验重复3次,共纳入5个随机字段进行分析。迁移细胞的百分比面积表示为平均值±SD。单因素方差分析,***P<0.001。比例尺:25 μm。
f. 采用划痕实验评估外源性PELI2过表达对ALKBH5敲低或对照HaCaT细胞迁移的影响。比例尺:100 μm。
g.创面划痕实验的统计学分析。所有实验重复3次,每个时间点的3个随机字段纳入分析。Mann-Whitney检验(24 h), ALKBH5-si1 + p-PELI2组与ALKBH5-si1 + p-NC组、ALKBH5-si2 + p-PELI2组与ALKBH5-si2 + p-NC组比较,差异均无统计学意义(p>0.05)。单因素方差分析(48 h, 72 h),***P<0.001

图7:外源性PELI2过表达挽救了ALKBH5敲低对再上皮化和创面愈合的影响。
- 创面建立后d0和d1, WT和ALKBH5-/-小鼠创缘皮肤皮内注射Lv-PELI2和Lv-NC (1×107 TU/ml, 1% Lipo3000)示意图。于PWD8时处死小鼠。
- qRT-PCR证实ALKBH5-/-小鼠创缘皮肤PELI2 mRNA表达恢复。5只小鼠的5个创面纳入分析。基因的相对表达水平以平均值±SD表示。单因素方差分析,*P<0.05,**P<0.01。
- IF分析证实Lv-PELI2转染后ALKBH5-/-小鼠创缘表皮PELI2表达恢复。虚线表示表皮边界。Epi表皮。比例尺:25 μm。
d-e. Lv-NC转染的WT小鼠以及Lv-PELI2或Lv-NC转染的ALKBH5-/-小鼠在PWD0、PWD2、PWD4、PWD6、PWD8的皮肤创面代表性图像。比例尺:2mm。黑色虚线圆圈绘制了5 mm宽的原始创面。采用ImageJ软件对创面闭合率(e)进行量化,e表示为创面未愈合面积的百分比。5只小鼠的10个创面纳入分析。创面闭合的相对百分比以平均值±SD表示。单因素方差分析(第2天,第4天,第6天),***P < 0.001。Mann-Whitney检验(第8天),双侧*P< 0.05 (KO + Lv-PELI2 vs. KO + Lv-NC)。
f-g. 用Lv-NC转染的WT小鼠和用Lv-PELI2或Lv-NC转染的ALKBH5-/-小鼠在划痕实验后第8天的创面再上皮化组织学分析(f)。箭头线表示原创面间隙宽度为5mm。钝端线描绘了上皮间隙,表示非上皮化创面宽度。再上皮化百分比定量(g)。5只小鼠的10个创面纳入分析。再上皮化创面的相对百分比以均数±SD表示。Mann-Whitney检验,双侧**P<0.01,****P<0.0001。虚线表示表皮边界。比例尺:200 μm
(7)ALKBH5以YTHDF2依赖性方式稳定PELI2 mRNA表达

图8:YTHDF2与m6A修饰结合并稳定PELI2 mRNA。
- RIP-qPCR分析显示YTHDF1、YTHDF2和YTHDF3在PELI2转录本中富集。实验设置4个重复,mRNA的相对表达量与input值进行比较,用平均值±SD表示。单因素方差分析,****P<0.0001。
- 用于RNA下拉分析的RNA探针示意图。
- 使用带有或不带有m6A修饰的PELI2 RNA探针,从细胞提取物中RNA下拉内源性YTHDF2蛋白。实验设置3个重复。
d-e. 通过qRT-PCR (d)和WB (e)评估ALKBH5和YTHDF2敲低组或对照组HaCaT细胞中的PELI2表达。实验进行3个重复。对PELI2的mRNA相对表达水平进行定量分析,并以平均值±SD表示。单因素方差分析,*P<0.05,**P<0.01。
f. 用放线菌素D (5 μg/mL)处理ALKBH5和YTHDF2敲除或对照HaCaT细胞0h、2 h、4 h、6 h、8 h, qRT-PCR分析PELI2的mRNA表达。实验重复三次。
g. m6A eraser蛋白ALKBH5上调介导m6A甲基化降低如何通过YTHDF2依赖性方式增强PELI2 mRNA的稳定性来促进创面再上皮化示意图
研究结论
本研究通过MeRIP-seq和对应的RNA-seq等分析揭示了创面再上皮化的一种新机制,通过该机制,m6A eraser蛋白ALKBH5在创缘被特异性激活,并通过从PELI2 mRNA中去除m6A位点并保护其免受YTHDF2介导的降解来促进角质形成细胞迁移。本研究从RNA表观转录组调控角度拓展了对创面再上皮化的理解。重要的是,ALKBH5和PELI2可能是开发针对难愈性创面患者的重编程m6A靶向治疗的潜在治疗靶点。
关于易基因RNA m6A甲基化测序(MeRIP-seq)技术
易基因MeRIP-seq技术利用m6A特异性抗体富集发生m6A修饰的RNA片段(包括mRNA、lncRNA等rRNA去除所有RNA),结合高通量测序,可以对RNA上的m6A修饰进行定位与定量,总RNA起始量可降低至10μg,最低仅需1μg总RNA。广泛应用于组织发育、干细胞自我更新和分化、热休克或DNA损伤应答、癌症发生与发展、药物应答等研究领域;可应用于动物、植物、细胞及组织的m6A检测。
大样本量m6A-QTL性状关联分析,传统MeRIP单个样品价格高,通常难以承担。易基因开发建立MeRIP-seq2技术,显著提成IP平行性,实现不同样本间相对定量,降低检测成本。
易基因提供适用于不同科研需求的MeRIP技术:
- m6A甲基化-常量mRNA 甲基化测序(MeRIP-seq)
- m6A甲基化-常量mRNA +lncRNA甲基化测序(lnc-MeRIP-seq)
- m6A甲基化-微量mRNA +lncRNA甲基化测序(Micro-lnc-MeRIP-seq)
- 高通量m6A甲基化-常量mRNA甲基化测序(MeRIP-seq2)
技术优势:
- 起始量低:样本起始量可降低至10-20μg,最低仅需1μg总RNA;
- 转录组范围内:可以同时检测mRNA和lncRNA;
- 样本要求:可用于动物、植物、细胞及组织的m6A检测;
- 重复性高:IP富集重复性高,最大化降低抗体富集偏差;
- 应用范围广:广泛应用于组织发育、干细胞自我更新和分化、热休克或DNA损伤应答、癌症的发生与发展、药物应答等研究领域。
研究方向:
m6A甲基化目前主要运用在分子机制的理论性研究
- 疾病发生发展:肿瘤、代谢疾病(如肥胖/糖尿病)、神经和精神疾病(如阿尔兹海默症/抑郁症)、炎症…
- 发育和分化:早期胚胎发育、个体/组织/器官生长发育、干细胞分化与命运决定、衰老
- 环境暴露与响应:污染、抗逆、生活方式
关于m6A甲基化研究思路
(1)整体把握m6A甲基化图谱特征:m6A peak数量变化、m6A修饰基因数量变化、单个基因m6A peak数量分析、m6A peak在基因元件上的分布、m6A peak的motif分析、m6A peak修饰基因的功能分析
(2)筛选具体差异m6A peak和基因:差异m6A peak鉴定、非时序数据的分析策略、时序数据的分析策略、差异m6A修饰基因的功能分析、差异m6A修饰基因的PPI分析、候选基因的m6A修饰可视化展示
(3)m6A甲基化组学&转录组学关联分析:Meta genes整体关联、DMG-DEG对应关联、m6A修饰目标基因的筛选策略
(4)进一步验证或后期试验

易基因科技提供全面的RNA甲基化研究整体解决方案。

参考文献:
Huang X, Zhao Y, Liu D, Gu S, Liu Y, Khoong Y, Luo S, Zhang Z, Xia W, Wang M, Liang H, Li M, Li Q, Zan T. ALKBH5-mediated m6A demethylation fuels cutaneous wound re-epithelialization by enhancing PELI2 mRNA stability. Inflamm Regen. 2023 Jul 14;43(1):36.
相关阅读:
干货系列:高通量测序后的下游实验验证方法——m6A RNA甲基化篇
干货系列:m6A RNA甲基化研究的数据挖掘思路
干货:m6A RNA甲基化MeRIP-seq测序分析实验全流程解析
项目集锦 | 易基因近期m6A甲基化(MeRIP-seq)研究成果