目录
步骤
拓展:CT值/HU值
步骤
不能直接可视化的原因:
seg.nrrd保存的是标签类别值,如下图标签类别值是[1,6],没有标记的部分值为0

1.将seg.nrrd保存为nrrd格式,并读取
2.将数组*int(255/标签类别数),变为灰度值
若直接将seg.nrrd当nrrd读取(直接读取会警告类型,且对seg.nrrd读取前做的Transforms会失效,但不影响运行)

import nrrd
num_ = 10 #切片数量
add_ = 2#间隔2帧
img_arr, _ = nrrd.read(file)
img_arr*=int(255/6)
D, H, W = image.shape
d0, h0, w0 = int(D/2), int(H/2), int(W/2)#坐标中心点作为原点
# dh
dh_slices = img_arr[:, :, w0-num_ : w0+num_ : add_]#从三轴方向每间隔间隔2帧,找20帧图像,即10帧
for i in range(num_):
cu = dh_slices[:,:,i]
cu_Image = img.fromarray(cu)
cu_Image = cu_Image.transpose(img.FLIP_TOP_BOTTOM)#上下对换
cu_array = np.array(cu_Image)
img_path = "{}/dh_{}.png".format(re_path_, i)
cv2.imwrite(img_path, cu_array)拓展:CT值/HU值
CT值又叫HU(Hounsfiled Unit)值,代表了物体真正的密度。
范围是-1024 -- x ,之所以说是x是因为各种资料众说纷纭,
对于nii格式的图片,nibabel, simpleitk常用的api接口,都会自动的进行原始数据转化HU值过程
(除非专门用nib.load('xx').dataobj.get_unscaled()或者itk.ReadImage('xx').GetPixel(x,y,z)才能取得原始数据)
对于dcm格式的图片,simpleitk, pydicom常用的api接口都不会将原始数据自动转化为Hu!!(itk snap软件读入dcm或nii都不会对数据进行scale操作)
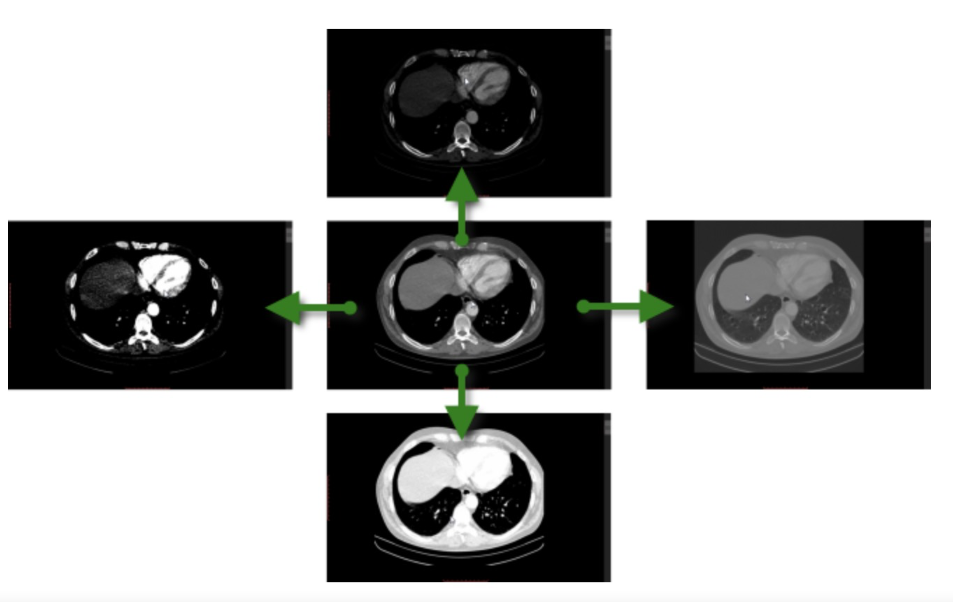
hu值是与设备无关的,不同范围之内的值可以代表不同器官。
hu的范围一般来说很大,这就导致了对比度很差,如果需要针对具体的器官进行处理,效果会不好,于是就有了windowing的方法






![[附源码]计算机毕业设计的汽车租赁系统Springboot程序](https://img-blog.csdnimg.cn/2f5b2572c4bc4856a87905d8e24a6f94.png)