简介
在统计学中很多推论与正态分布有关,并且很多统计量构造为满足正态分布的形式,很多分布在特定条件近似于正态分布。因此,在统计推断中经常需要判断样本的正态性。本文介绍一些常用的方法。
环境和数据准备:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from scipy import stats
from statsmodels.stats import diagnostic
# check out https://github.com/Paradiseeee/Heaven
from Heaven import mpl_tools
mpl_tools.Begin().setting()
# 正态分布样本与二项分布样本(两者方差均值一致)
n_sample = stats.norm.rvs(2, np.sqrt(1.6), 100)
b_sample = stats.binom.rvs(10, 0.2, size=100)
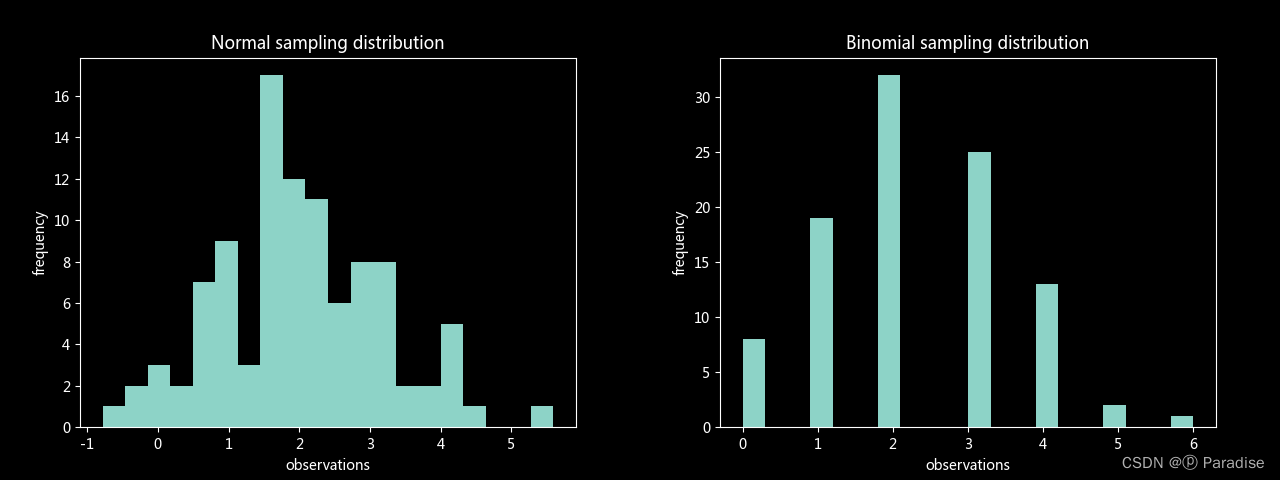
plt.hist(n_sample, bins=20)
plt.title('Normal sampling distribution')
plt.xlabel('observations'); plt.ylabel('frequency'); plt.show()
plt.hist(b_sample, bins=20)
plt.title('Binomial sampling distribution')
plt.xlabel('observations'); plt.ylabel('frequency'); plt.show()
一、通过绘图直观判断
频数(分布密度)图:
通过图像对比判断样本是否服从理论总体的分布,需要总体分布已知(或假设拟合相应分布)
# 模拟总体
pop = stats.norm.rvs(2, np.sqrt(1.6), 10000)
# 绘制常见的分布图形
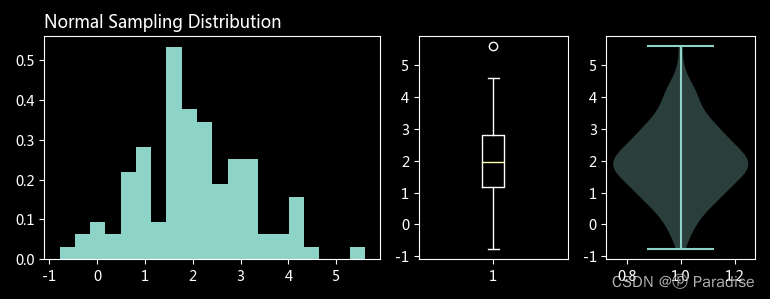
def plot_dist(rv, title):
axes = mpl_tools.Func().get_subplots('AABC')
# 频数密度图
axes['A'].hist(rv, bins=20, density=True)
# 箱形图
axes['B'].boxplot(rv)
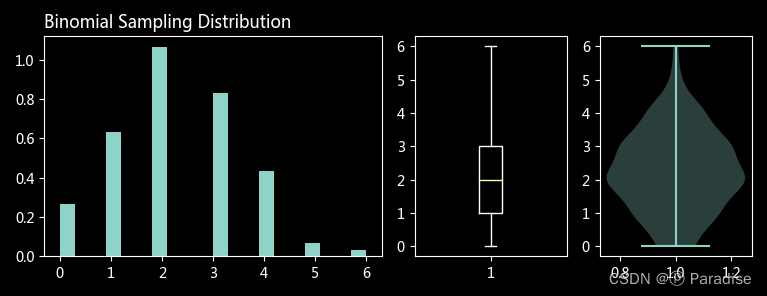
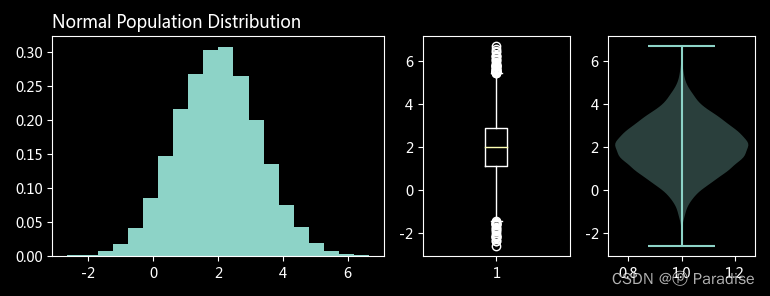
# 小提琴图(竖起来的 KDE 图)
axes['C'].violinplot(rv)
plt.title(title, loc='left')
plt.show()
plot_dist(n_sample, 'Normal Sampling Distribution')
plot_dist(b_sample, 'Binomial Sampling Distribution')
plot_dist(pop, 'Normal Population Distribution')
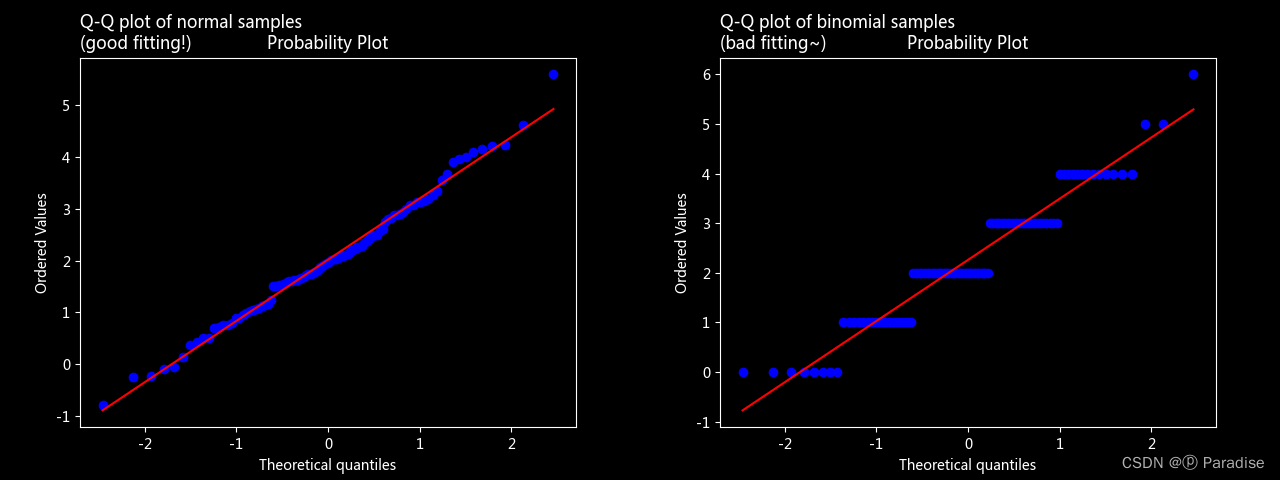
Q-Q 图
通过抽样和总体的分位数进行对比
# stats.probplot 严格来说是“概率图”,坐标轴未缩放的 Q-Q 图
stats.probplot(n_sample, dist='norm', plot=plt)
plt.title('Q-Q plot of normal samples \n(good fitting!)', loc='left')
plt.show()
stats.probplot(b_sample, dist='norm', plot=plt)
plt.title('Q-Q plot of binomial samples \n(bad fitting~)', loc='left')
plt.show()
二、通过参数量化比较
绘图方法优点在于直观,但是得出结论只能是粗略的,只是“看起来像”,缺乏可信度。通过统计参数对比可以更准确的量化样本的正态性。
Shapiro-Wilk test
results = pd.DataFrame(
{'normal':[None]*4, 'binomial':[None]*4},
index=['Shapiro', 'KS', 'Anderson', 'Lilliefors']
)
# 构造了一个统计量,该统计量越高,表示样本越有可能是正态分布
ntest = stats.shapiro(n_sample)
btest = stats.shapiro(b_sample)
results.loc['Shapiro', 'normal'] = (ntest.statistic, ntest.pvalue)
results.loc['Shapiro', 'binomial'] = (btest.statistic, btest.pvalue)
Kolmogorov-Smirnov test
ntest = stats.kstest(n_sample, stats.norm.cdf, args=(2, np.sqrt(1.6)))
btest = stats.kstest(b_sample, stats.norm.cdf, args=(2, np.sqrt(1.6)))
results.loc['KS', 'normal'] = (ntest.statistic, ntest.pvalue)
results.loc['KS', 'binomial'] = (btest.statistic, btest.pvalue)
Anderson-Darling test
ntest = stats.anderson(n_sample, dist='norm')
btest = stats.anderson(b_sample, dist='norm')
# 不提供连续的 p 值,取显著性水平为 1% 时的临界值
results.loc['Anderson', 'normal'] = (ntest.statistic, ntest.critical_values[ntest.significance_level == 1][0])
results.loc['Anderson', 'binomial'] = (btest.statistic, btest.critical_values[ntest.significance_level == 1][0])
Lilliefors’ test
ntest = diagnostic.lilliefors(n_sample, dist='norm')
btest = diagnostic.lilliefors(b_sample, dist='norm')
results.loc['Lilliefors', 'normal'] = ntest
results.loc['Lilliefors', 'binomial'] = btest
results = results.applymap(lambda x: tuple([round(i, 5) for i in x]))
print(results.to_markdown())
| normal | binomial | |
|---|---|---|
| Shapiro | (0.99032, 0.6905) | (0.93856, 0.00016) |
| KS | (0.07716, 0.56435) | (0.23, 4e-05) |
| Anderson | (0.3403, 1.053) | (2.65129, 1.053) |
| Lilliefors | (0.06062, 0.48814) | (0.17224, 0.001) |
所有结果如上表,其中每个元组的第一位是对应的统计量,第二位是 p 值或者临界值,具体解释如下:
-
对于 Shapiro 测试,统计量越大越接近正态分布(用于支撑原假设),p 值越小越拒绝原假设。从统计量来看,两个分布的样本都接近于正态分布,但是二项分布样本的 p 值明显小,在一般的显著性水平下(5%)可以拒绝原假设,即不服从正态分布。
-
对于 KS 测试,正态样本的 p 值大于显著性水平,二项分布样本的 p 值小于显著性水平,拒绝原假设。
-
对于 Anderson 测试,第一项是统计量,第二项是对应显著性水平为 1% 时的临界值(对于正态测试可选的有 15%、10%、5%、2.5%、1%,对于其他分布测试有不同的序列)。可以看到对于正态样本,统计量小于临界值,在 1% 的显著性水平下不能拒绝原假设;对于二项分布样本,拒绝原假设,样本不服从正态分布(2.65 > 1.05)。
-
对于 Lilliefors 测试,根据第二项的 p 值进行判断,可以看到二项分布样本的 p 值为 0.001,可以拒绝原假设,不服从正态分布。
相关阅读 | 使用 fitter 拟合数据分布
阅读原文 | Paradise’s Blog
END