2021年国赛高教杯数学建模
E题 中药材的鉴别解题
原题再现
不同中药材表现的光谱特征差异较大,即使来自不同产地的同一药材,因其无机元素的化学成分、有机物等存在的差异性,在近红外、中红外光谱的照射下也会表现出不同的光谱特征,因此可以利用这些特征来鉴别中药材的种类及产地。
中药材的种类鉴别相对比较容易,不同种类的中药材呈现的光谱的区别比较明显。图 1 为两种不同药材的近红外光谱数据曲线图,容易看出两者的差异比较大。
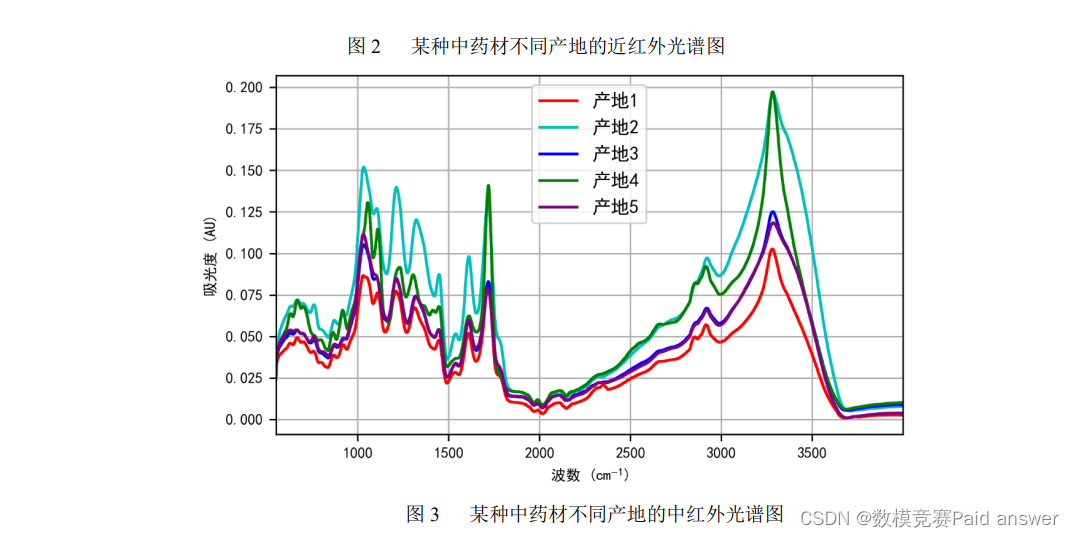
中药材的道地性以产地为主要指标,产地的鉴别对于药材品质鉴别尤为重要。然而,不同产地的同一种药材在同一波段内的光谱比较接近,使得光谱鉴别的误差较大。另外,有些中药材的近红外区别比较明显,而有些药材的中红外区别比较明显(见图 2 和图 3 所给出的来自某药材 5 个不同产地的近红外和中红外光谱数据曲线图)。当样本量不够充足时,我们可以通过近红外和中红外的光谱数据相互验证来对中药材产地进行综合鉴别。
附件 1 至附件 4 是一些中药材的近红外或中红外光谱数据,其中 No 列为药材的编号,Class 列表示中药材的类别, OP 列表示该种药材的产地,其余各列第一行的数据为光谱的波数(单位 cm-1)、第二行以后的数据表示该行编号的药材在对应波段光谱照射下的吸光度(注:该吸光度为仪器矫正后的值,可能存在负值)。试建立数学模型,研究解决以下问题。
问题 1. 根据附件 1 中几种药材的中红外光谱数据,研究不同种类药材的特征和差异性,并鉴别药材的种类。
问题 2. 根据附件 2 中某一种药材的中红外光谱数据,分析不同产地药材的特征和差异性,试鉴别药材的产地,并将下表中所给出编号的药材产地的鉴别结果填入表格中。
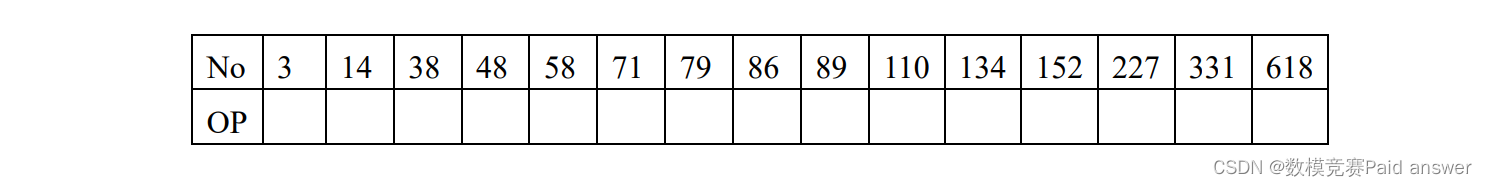
问题 3. 根据附件 3 中某一种药材的近红外和中红外数据,试鉴别该种药材的产地,并将下表中所给出编号的药材产地的鉴别结果填入表中。
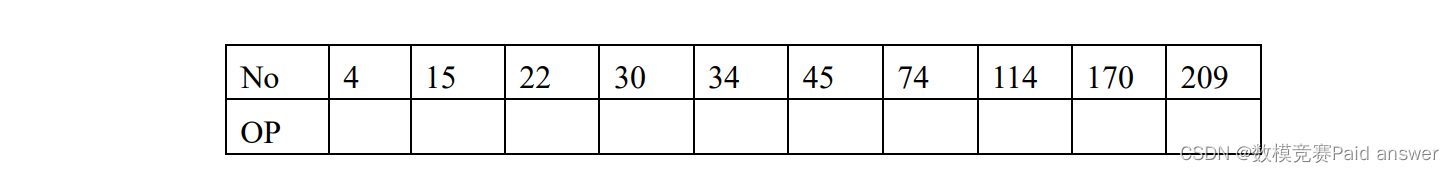
问题 4. 附件 4 给出了几种药材的近红外光谱数据,试鉴别药材的类别与产地,并将下表中所给出编号的药材类别与产地的鉴别结果填入表各中。
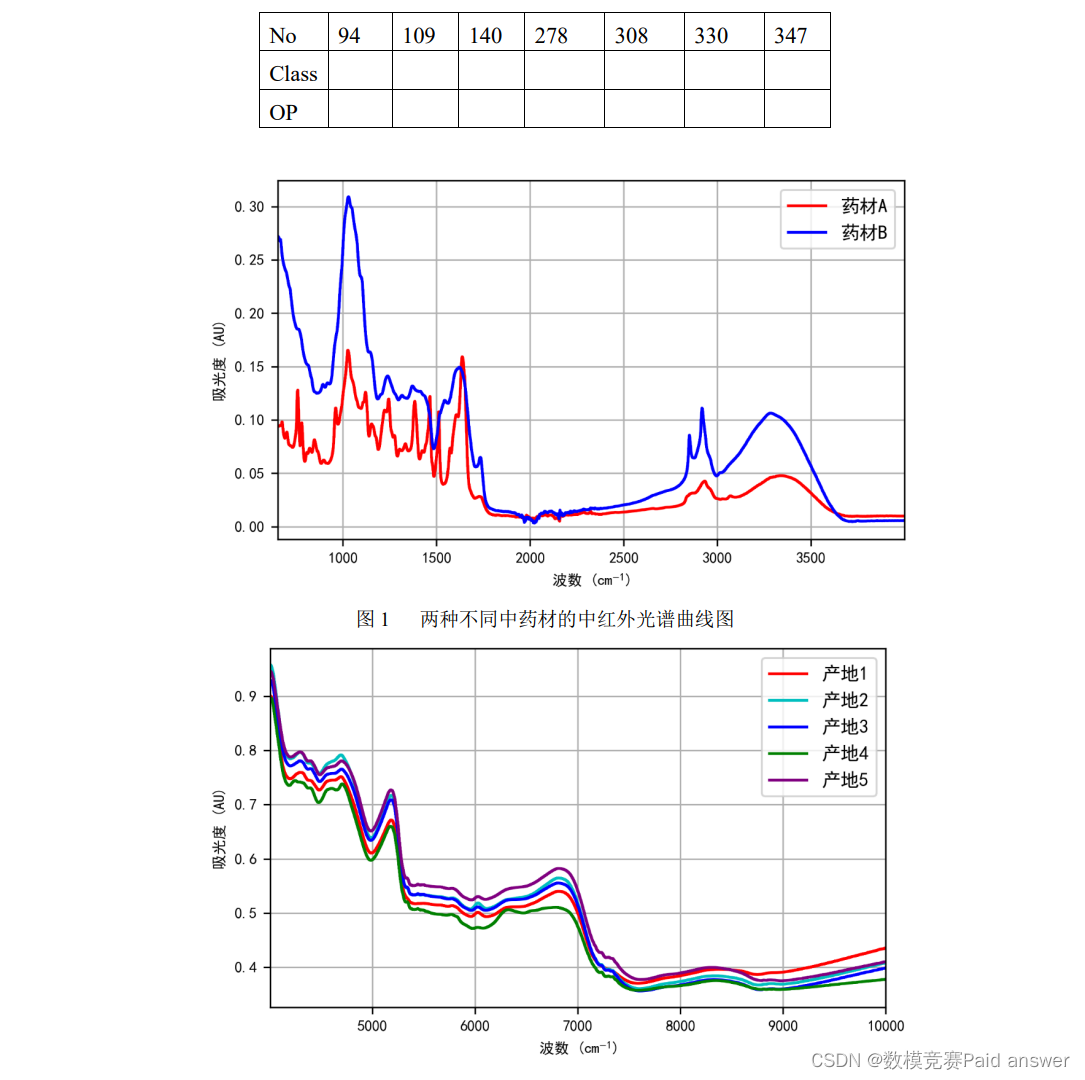
整体求解过程概述(摘要)
科学准确地鉴别中药材类别与产地,对于智慧中药产业具有重要的作用。本文构建了基于智能优化和机器学习结合的中药材鉴别模型,有效提高了中药材的鉴别类别与产地的精度,主要解决以下问题:
针对问题一,首先,对附件1数据进行数据预处理,并将样本进行描述性统计和可视化分析:其次,分别将原始数据、主成分分析降维后数据、提取不同波段长度的特征作为属性数据,通过肘部法则、平均轮廓法和间隔统计量法确定最优聚类数。利用K-means和Ward方法对中药材进行聚类分析,将不同类别的药材数据进行差异性分析。
针对问题二,将11个不同产地药材的中红外光谱数据进行统计分析,绘制出每个产地对应光谱数据的均值曲线和方差曲线,将原始数据进行了降维和分波段计算特征处理,形成了包括原始数据在内的五类不同数据集,分别利用LightGBM、极端梯度增强 (XGBoost ) 、支持向量机 (SVM) 、随机森林 ( RF ) 、梯度提升决策树 ( GBDT ) 和多层感知机(MLP)六种机器学习算法进行分类,采用交叉验证计算四个评价指标对分类结果进行评价。最后,利用不同机器学习方法给出药材产地。鉴别结果详见下表。

针对问题三,基于鲸鱼优化和机器学习的药材产地鉴别模型。首先,提取不同区间波段特征属性,分别将 13 个不同产地药材的近红外光谱数据进行特征分析,分别利用LightGBM 等6种算法对药材产地进行分类识别,并选出有利于产地识别的重要特征;其次,利用中红外光谱数据提取不同区间波段特征属性,利用上述6 种算法对药材产地进行分类识别:利用上述两个方案组成产地识别的特征集,利用不同机器学习方法选定最佳模型:最后,提出基于鲸鱼优化和 LightGBM方法结合的药材产地识别模型,并对药材产地进行识别。鉴别结果详见下表。

针对问题四,将附件4近红外数据进行统计分析并提取特征,参照问题三建模思路,利用不同机器学习算法对中药材的类别和产地进行分类预测;通过构建中药材类别预测模型,为未知的中药材样本类别进行预测和标记,再对中药材产地建模。同理,通过构建中药材产地预测模型,为未知的中药材样本产地进行预测和标记,对中药材类别构建类别预测模型。充分挖掘不同波段对药材类别和产地分类效果差异性,构建基于机器学习的两阶段药材类别与产地识别模型,鉴别结果详见下表。

本文创新之处:采用主成分分析对数据进行降维,利用动态 K-eans 实现中药材聚类并进行差异性分析。提出鲸鱼优化和机器学习结合的中药材类别与产地识别模型,该模型识别效果好、精度高,对葡萄酒分类、精准医疗、故障诊断等领域具有重要的参考价值。
模型假设:
1.不同产地的同一种药材光谱特征不完全相同;
2.对应波段光谱照射下的吸光度为仪器矫正后的值;
3.不同种类中药材的光谱特征之间的存在差异;
4.中药材的红外光谱数据误差在可接受范围内。
问题分析:
本文主要解决红外光谱下中药材的分析与鉴别问题。要求依据所提供近红外和中红外的光谱数据,对不同种类药材的特征和差异性进行分析,并鉴别药材类别与产地。
问题一的分析
根据附件1中已有的几种药材的中红外光谱数据,研究药材的特征和它们之间的差异性,并鉴别药材种类。由于红外光谱的高度特征性,在中红外光谱的照射下要想鉴别药材的种类可以将在对应波段光谱照射下的不同的吸光度来进行分类,将具有相似性的数据认定为同一种类。对附件1进行数据预处理,再对数据进行可视化分析,并将个体样本进行描述性统计分析,再分别利用不同的聚类分析方法对中红外光谱数据进行分类;最后,将不同类别的中药材数据进行特征提取和差异性分析。
问题二的分析
根据附件2中某一种药材的中红外光谱数据,分析不同产地药材的特征和差异性,并鉴别药材的产地。首先,分别将 11个不同产地药材的中红外光谱数据进行综合汇总绘制出每个产地对应光谱数据的均值曲线和方差曲线,同时,为更好衡量不同产地药材的差异性和区分度,我们将原始数据进行了降维和分波段计算特征处理,形成了包括原始数据在内的五类不同数据集,比较分析了支持向量机 (SVM) 、随机森林 ( RP) 、 极端梯度增强 (XGBoost) 、梯度提升决策树 (GBDT)、LihtGBM 和多层感知机 (MLP) 这六种机器学习分类算法。本文中的六种分类算法都是根据训练集建立模型,进行五折交叉验证,从而对测试集进行预测,并与真实结果进行对比,应用的评价指标为Precision、Recall、F1-score 以及Accuracy。最后,用不同机器学习方法给出文中所给编号的药材产地鉴别结果。
问题三的分析
构建基于机器学习的药材产地鉴别模型。首先,利用近红外光谱数据提取不同区间波段特征属性,分别利用 SVM、RF、XGBoost、GBDT、LSTM 和MLP 等不同机器学习算法对药材产地进行分类识别,并筛选出有利于产地识别的重要特征:其次,利用中红外光谱数据提取不同区间波段特征属性,分别利用上述6 种不同机器学习算法对药材产地进行分类识别,并筛选出对产地鉴别有利的重要特征:再次,利用上述两个方案筛选后组成产地识别的特征集,利用不同机器学习方法选定最佳模型:最后,提出基于鲸鱼优化和XGBoost 结合的药材产地识别模型,并计算出药材的鉴别结果。
问题四的分析
借鉴上述问题的建模思路,通过研究不同药材的近红外光谱数据,分析不同药材的特征和差异性,以及不同产地的同一种药材鉴别具有一定难度。本文分三种情形建模:包括已知类别,未知产地的识别模型:未知类别、已知产地的识别模型:类别和产地均未知的识别模型。利用 SVM、RF、XGBoost、GBDT、LSTM 和MLP 等不同机器学习算法对药材类别和产地进行识别,并进行综合评定,最终给出文中所给编号的药材类别与产地的鉴别结果。
模型的建立与求解整体论文缩略图

全部论文请见下方“ 只会建模 QQ名片” 点击QQ名片即可
程序代码:(代码和文档not free)
from sklearn.decomposition import PCA #主成分分析法
#PCA方法降维
#保持90%的信息
pca = PCA(n_components=0.95)
pca_95 = pca.fit_transform(data.iloc[1:,:])
pca_95.shape
pca.explained_variance_ratio_
pca.explained_variance_ratio_.sum()
plt.figure(dpi = 600,figsize = (5,3))
plt.scatter(pca_95[:,0],pca_95[:,1])
# K-Means聚类
# 惯性值
SSE = []
# 轮廓分数
SIL = []
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_score
for i in range(2,8):
kmeans = KMeans(n_clusters = i,n_init = 100)
y_pred = kmeans.fit_predict(pca_95)
SSE.append([i,kmeans.inertia_])
SIL.append([i,silhouette_score(pca_95,kmeans.labels_)])
# 将list转为DataFrame
sse = pd.DataFrame(SSE,columns = ["k","SSE"])
sil = pd.DataFrame(SIL,columns = ["k","SIL"])
# 绘制手肘图
plt.figure(dpi = 600,figsize = (5,3))
plt.plot(sse["k"],sse["SSE"])
plt.scatter(sse["k"],sse["SSE"])
plt.savefig("手肘图.png")
kmeans = KMeans(n_clusters = 3,n_init = 100)
y_pred = kmeans.fit_predict(pca_90)
y_pred