什么是Ka/Ks?
在遗传学中,Ka/Ks表示的是两个蛋白编码基因的非同义替换率(Ka)和同义替换率(Ks)之间的比例。这个比例可以判断是否有选择压力作用于这个蛋白质编码基因。
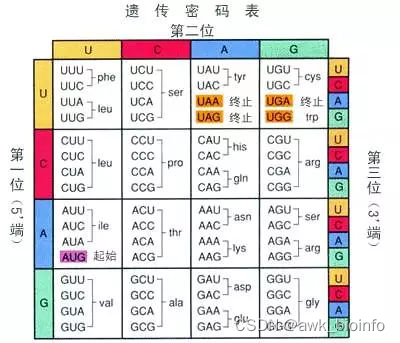
如果你手头有两个不同物种的同一个基因的序列,比如人和小鼠的p53基因,然后把这两个基因的序列进行比对,你会发现这两段序列有差异(进化!)。再仔细观察,你会发现有些碱基的变化导致了编码氨基酸的变化(非同义替换),有些没有导致编码氨基酸的变化(同义替换)。这是由密码子的简并性造成的,因为3个碱基决定1个氨基酸,所以64种碱基组合决定20种氨基酸,会有冗余出现。一般情况下,第三个碱基变化会造成同义替换,而第一二个碱基的变化会非造成同义替换。
Ka和Ks的计算公式:
Ka=发生非同义替换的SNP数/非同义替换位点数
Ks=发生同义替换的SNP数/同义替换位点数
其中同义替换位点数就是不会造成氨基酸变化的位点数的总和,比如编码丝氨酸(ser)的第三位碱基。而非同义替换位点数就是会造成氨基酸变化的位点数的总和,比如编码丝氨酸(ser)的第一二位碱基。对于像第一个方框里面的苯丙氨酸(phe)和亮氨酸(leu)这种情况,第三位碱基的变化只是有可能造成氨基酸的变化,也就是两种不同的氨基酸各占了两个碱基的位点,则在计算非同义替换位点数和同义替换位点数时,各加上1/2,相当于两者平分了这个位点。另外,计算Ka/Ks时,不考虑start codon和stop codon。
好了,现在我们回到上面的人和小鼠的p53基因的例子。我们只需要数一下两个序列发生(非)同义替换的SNP位点数,再除以(非)同义替换位点数,就得到了Ka和Ks值,然后就可以计算Ka/Ks ratio。
上面的计算方法并没有考虑不同碱基之间发生替换的速率的不同,比如,嘌呤之间替换的概率(A=>G)要高于嘌呤替换为嘧啶的概率(A=>C或T),也就是说转换(transition,嘌呤变嘌呤,嘧啶变嘧啶)发生的概率要高于颠换(transversion,嘌呤变嘧啶,嘧啶变嘌呤)发生的概率。很多计算方法都会考虑到这些替换发生概率的不同。
另外,两个物种分化时间的长短也会影响到Ka/Ks的比值。比如有一个位点,原来是A,后来变成了T,再后来又变成了C,虽然发生了两次替换,但最后仅有一次替换被用于计算替换率。再比如有一个位点,原来是A,后来变成T,但同时与它相对应的另一个序列的位点,也发生了A到T的替换,那么我们也是无法用上面的方法来计算替换率。对于这种复杂的情况,我们可以用最大似然法来计算最可能的替换率,这里不再详述。
那么Ka/Ks和进化有什么关系?
细心的小伙伴可能已经看出来了,在上面的密码子表中,同义替换位点比较少。实际上一个基因中大概只有25%的位点是同义替换位点。假设这个基因没有受到自然选择压力,那么根据中性选择理论,非同义替换率和同义替换率应该是相同的。
一般来讲,因为非同义替换会造成氨基酸变化,可能会改变蛋白质的构象和功能,因此会造成适应性的变化,从而带来自然选择的优势或劣势(一般是劣势)。而非同义替换没有改变蛋白质的组成,因此不受自然选择的影响(当然这里我们忽略密码子偏好性的影响),那么Ks就能反映进化过程的背景碱基替换率。Ka/Ks的比值就能说明这个基因是受到了何种选择。
长颈鹿的长脖子基因肯定是受到了正选择…
一般情况下,在某个个体中偶然发生的一个碱基替换(突变),如果没有额外的好处或者坏处的话,慢慢地也就消失了。但是自然选择中会有很多巧合,某些突变就是很幸运地被保留了下来,并且被固定了(突变频率由极小变为100%)。一个这样的突变在一个二倍体种群中被固定的可能性为1/2N,其中N是种群大小。
对于一个没有受到自然选择压力的基因来说,我们可以计算得到这样的结果:Ka/Ks=1。但实际情况下,这个比值都是远小于1的:Ks/Ks<<1,因为一般非同义替换带来的都是有害的性状,只有极少数情况下会造成进化上的优势。
于是,我们有了下面的分类:
Ka>>Ks或者Ka/Ks >> 1,基因受正选择(positive selection)
Ka=Ks或者Ka/Ks=1,基因中性进化(neutral evolution)
Ka<<Ks或者Ka/Ks << 1,基因受纯化选择(purify selection)
KaKs分析方法
1. 可以使用Kaks_calculator2.0计算 KaKs
-
下载地址:https://udomain.dl.sourceforge.net/project/kakscalculator2/KaKs_Calculator2.0.rar
输入文件为axt格式,具体要求如下example.axt:

文件总共三行,第一行使配对基因或同源基因的名字,后面是两个比较物种的基因序列。 -
运行脚本:
/biodata/02.software/KaKs_Calculator2.0/bin/Linux/KaKs_Calculator -i example.axt -o example.axt.kaks -m GMYN
2. 使用ParaAT套件分析KaKs
- 下载地址:https://download.cncb.ac.cn/bigd/tools/ParaAT2.0.tar.gz
准备输入文件:
- test.homologs: 同源基因对儿
- test.cds: 同源基因对儿的cds序列
- test.pep:同源基因对儿的pep序列
- proc:线程数
以上文件均可在ParaAT文件夹中找到,可做参考。
比对参数推荐使用muscle,速度快,效果好。
- 运行脚本:
ParaAT.pl -h test.homologs -n test.cds -a test.pep -p proc -m muscle -f axt -g -k -o result_dir
- -h, 同源基因名称文件
- -n, 指定核酸序列文件
- -a, 指定蛋白序列文件
- -p, 指定多线程文件
- -m, 指定比对工具
- -g, 去除比对有gap的密码子
- -k, 用KaKs_Calculator 计算kaks值
- -o, 输出结果的目录
- -f, 输出比对文件的格式
- *** 也可通过-f参数得到其他软件分析ka/k所需的格式
参考:https://zhuanlan.zhihu.com/p/144271268?utm_id=0