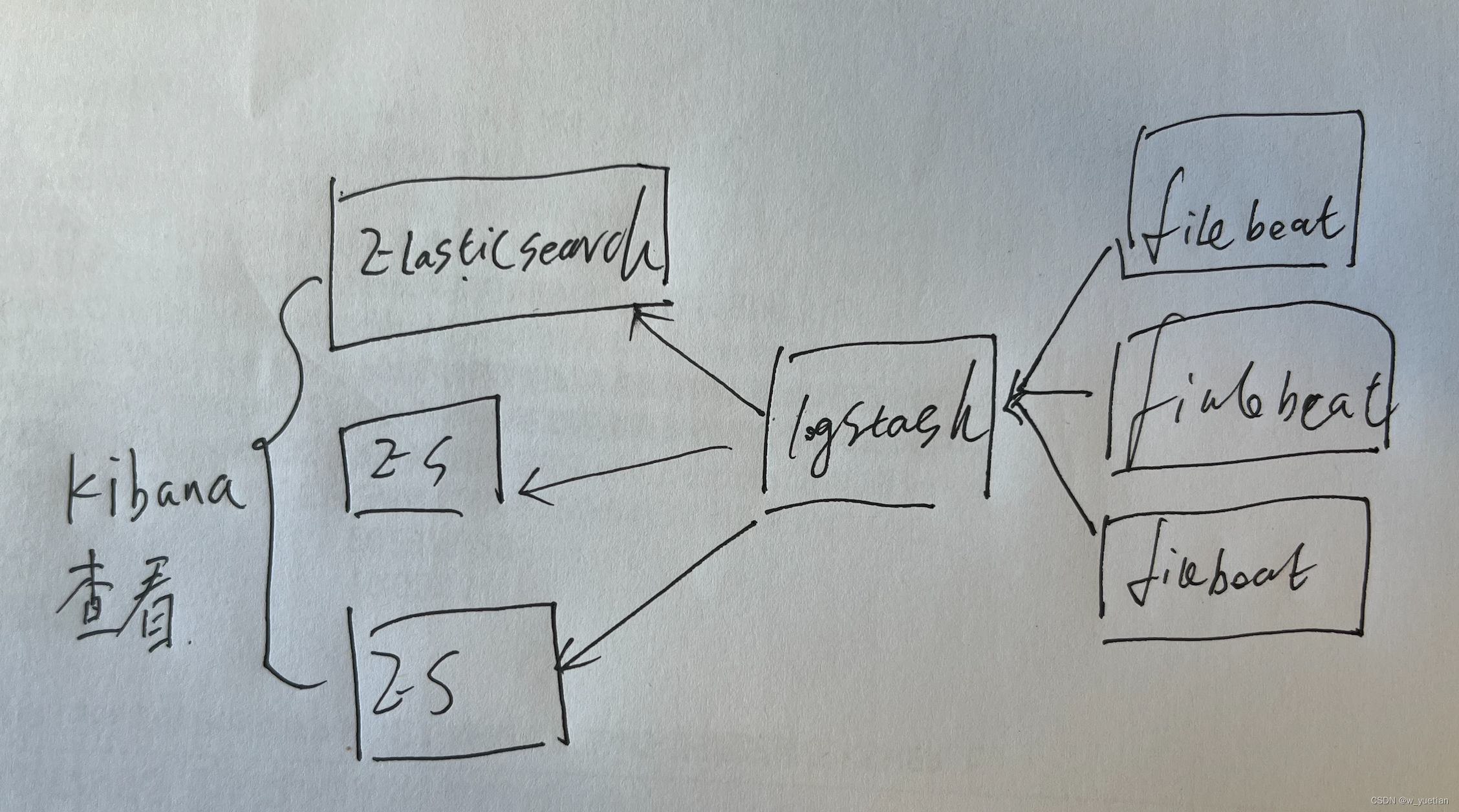
一、引言
乳腺癌作为全球常见的恶性肿瘤,给患者和医学界带来了巨大的挑战。据世界卫生组织的数据显示,乳腺癌是妇女中最常见的癌症之一,并且是全球癌症相关死亡的主要原因之一[1]。因此,研究乳腺癌,并努力提高其早期检测、预防和治疗的效果,对于减少乳腺癌的发病率和提高患者生活质量至关重要。
无监督学习作为一种机器学习方法,可用于从未标记的数据中发现模式和关联性。在乳腺癌研究中,无监督学习提供了一种新的方式,可以发现隐藏在大规模乳腺癌数据中的信息。同时,R语言作为一种功能强大的统计分析工具,为乳腺癌研究提供了丰富的数据处理和可视化功能。
以往的研究已经证明了无监督学习和R语言在乳腺癌研究中的潜力。例如,一项研究使用无监督学习技术对乳腺癌组织样本进行聚类,并成功地将其分为不同的亚型,有助于更好地了解乳腺癌的生物学特征和预后[2]。此外,研究人员还利用R语言的强大数据处理功能,对乳腺癌患者的基因表达数据进行分析,并发现了一些与肿瘤进展相关的关键基因[3]。
因此,本文旨在进一步探讨无监督学习和R语言在乳腺癌研究中的应用。通过引用相关文献的支持,我们将深入探讨无监督学习和R语言在乳腺癌研究中的具体应用案例,并讨论它们是如何提高我们对乳腺癌的理解和诊断的。
二、乳腺癌研究概述
无监督学习是一种机器学习方法,其目的是从未标记的数据中发现隐藏的模式和关系。在乳腺癌研究中,无监督学习可以帮助研究人员探索和理解不同乳腺癌亚型之间的差异,以及乳腺癌患者的分类和预后。
R语言是一种广泛应用于统计学和数据分析的编程语言,并且在生物医学领域得到了广泛的应用。R语言具有丰富的数据处理、统计模型和可视化的功能,适合处理和分析乳腺癌相关的大规模数据集。
在乳腺癌研究中,研究人员可以利用R语言编写脚本来对乳腺癌患者的基因表达数据、突变数据、临床特征等进行统计分析和可视化。这些分析可以帮助研究人员发现与乳腺癌发展和治疗相关的关键因素。
无监督学习和R语言的结合可以为乳腺癌研究带来许多优势。首先,无监督学习可以帮助研究人员在不依赖领域知识的情况下,从大量的乳腺癌数据中发现新的亚型和模式。其次,R语言作为一个开源的工具,拥有丰富的乳腺癌研究包和函数,可以提供高效、可重复和灵活的分析和可视化方法。
然而,引入无监督学习和R语言也存在一些挑战。首先,乳腺癌数据往往具有高维度和复杂性,需要合适的算法和技术来处理和分析。其次,无监督学习的结果通常需要经过进一步的验证和解释,以确保其在生物学上的合理性。
总之,无监督学习和R语言作为新的研究手段可以为乳腺癌研究提供新的视角和方法。通过利用这些工具,我们可以更好地理解乳腺癌的异质性,并为乳腺癌的预后评估和个体化治疗提供更多的信息和支持。
三、无监督学习简介
3.1 解释无监督学习的基本原理和目标
无监督学习是一种机器学习方法,其目标是从未标记的数据中发现潜在的模式和关系,而无需依赖人工标记的输出。与监督学习不同,无监督学习不需要已知的目标变量或标签来指导学习过程,而是依靠算法自动发现数据中的结构和相似性。
无监督学习的基本原理是通过聚类和降维等技术,将相似的数据样本聚集在一起,同时将不相似的数据样本分开。聚类可用于识别数据中的簇或群组,而降维则可用于减少数据的维度,以便更好地可视化和理解数据。
无监督学习的目标包括发现数据的隐藏结构、提取数据的重要特征、检测异常值和离群点等。通过无监督学习,研究人员可以深入了解数据的内在性质和模式,为后续的分析和应用提供基础。
3.2 介绍无监督学习常用的算法和技术
在无监督学习中,常用的算法和技术包括:
-
1. 「聚类算法」:如K-means聚类算法、层次聚类算法(Hierarchical Clustering)和密度聚类算法(Density-Based Clustering)等。这些算法可以将相似的数据样本聚集在一起,形成簇或群组。 -
2. 「降维算法」:如主成分分析(Principal Component Analysis,PCA)和独立成分分析(Independent Component Analysis,ICA)等。这些算法可以将高维数据映射到低维空间,以便更好地可视化和理解数据。 -
3. 「关联规则挖掘」:用于发现数据中的频繁项集和关联规则。关联规则挖掘可以揭示数据中的相关性和依赖关系。
3.3 阐述无监督学习在乳腺癌研究中的潜在应用价值
无监督学习在乳腺癌研究中具有许多潜在应用价值。首先,无监督学习可以用于乳腺癌亚型的发现和分类。通过对乳腺癌患者的基因表达数据进行聚类分析,可以发现不同亚型之间的差异和相似性,有助于进一步理解乳腺癌的异质性和疾病进展。
其次,无监督学习可以帮助识别乳腺癌中的重要基因和特征。通过降维算法如PCA,可以从大规模的基因表达数据中提取最具代表性和相关性的基因,从而揭示与乳腺癌发展和治疗响应密切相关的生物学特征。
此外,无监督学习还可以用于乳腺癌的预后评估和个体化治疗。通过对乳腺癌患者的临床数据进行聚类分析,可以识别出不同预后风险群体,并为患者提供个体化的治疗策略和建议。
综上所述,无监督学习在乳腺癌研究中具有广泛的应用潜力。通过无监督学习的方法,可以帮助研究人员更全面地了解乳腺癌的复杂性,并为乳腺癌的诊断、治疗和预后评估提供更多的信息和支持。
四、实用案例分析
4.1 已有无监督学习的乳腺癌研究案例
已有研究表明,无监督学习和R语言在乳腺癌研究中有广泛的应用。一些具体的案例包括:
-
基于基因表达数据的聚类分析:研究人员使用R语言中的无监督学习算法,如基于聚类的方法,对乳腺癌基因表达数据进行聚类分析[2]。这种分析方法可以将乳腺癌患者分为不同的亚型,从而帮助揭示乳腺癌的生物学特征和预后。 -
乳腺癌图像分析:无监督学习算法在乳腺癌图像分析中的应用也有所成果。研究人员借助R语言中的图像处理和机器学习工具,实现对乳腺癌图像的分割、特征提取和分类[3]。这些技术的采用有助于提高乳腺癌图像的诊断准确性和效率。
之后将会拜读文献[2]和[3],复现出他们的无监督学习过程和代码。感兴趣的可以关注和私信我,我们一起学习高分SCI的论文数据分析技巧。
4.2 乳腺癌病例分析
上篇文章乳腺癌预测的黑科技!一起探索 R 语言和随机森林算法的奇妙之处!,我们通过随机森林对疾病的结果做出了预测,但是结果不是很理想,auc值只有0.798,我们现在可以通过无监督学习来提示随机森林的预测准确度。
4.2.1 提升准确度思路
通过无监督学习来提升随机森林的预测准确度可以采用以下几种方法和技术:
-
「特征选择」:使用无监督学习方法如聚类、降维来进行特征选择。通过聚类分析,可以将相似的特征进行分组,并选择每个组中最具代表性的特征作为输入的一部分。降维方法如主成分分析(PCA)和因子分析可以减少高维特征空间中的冗余信息,提取主要的特征。 -
「异常检测和数据清洗」:无监督学习方法可以帮助识别和处理异常数据点,以减少对模型的干扰。通过异常检测技术,可以找到样本中与正常模式不一致的异常点,并进一步对其进行处理,包括删除、修复或重新标记。 -
「集成学习」:使用无监督学习方法来构建集成模型。例如,可以将多个无监督学习模型的输出作为输入,通过投票或加权的方式进行集成,以改进预测的稳定性和准确性。 -
「数据增强」:通过对无监督学习模型生成的合成样本进行数据增强,扩大训练集的规模和多样性。这可以减少过拟合,并提高随机森林模型的泛化能力。 -
「半监督学习」:利用有监督学习中的有标签数据和无监督学习中的无标签数据进行训练。半监督学习可以通过增加无标签数据的利用,提供更多的信息来改进模型的性能。
这里我们主要演示如何通过特征选择的方式来提升随机森林的预测准确度。如果想了解异常检测和数据清洗、集成学习、数据增强和半监督学习的方式,关注和私信我,我们一起讨论学习。当然如过想了解降维算法和关联规则挖掘的使用方法也可以和我交流。关注我,一起学习和进步!
4.2.2 数据准备与探索性分析
-
1.数据载入和解释
library(survival)
str(gbsg)
结果展示:
'data.frame': 686 obs. of 10 variables:
$ age : int 49 55 56 45 65 48 48 37 67 45 ...
$ meno : int 0 1 1 0 1 0 0 0 1 0 ...
$ size : int 18 20 40 25 30 52 21 20 20 30 ...
$ grade : int 2 3 3 3 2 2 3 2 2 2 ...
$ nodes : int 2 16 3 1 5 11 8 9 1 1 ...
$ pgr : int 0 0 0 0 0 0 0 0 0 0 ...
$ er : int 0 0 0 4 36 0 0 0 0 0 ...
$ hormon : int 0 0 0 0 1 0 0 1 1 0 ...
$ rfstime: int 1838 403 1603 177 1855 842 293 42 564 1093 ...
$ status : Factor w/ 2 levels "0","1": 1 2 1 1 1 2 2 1 2 2 ...
age:患者年龄
meno:更年期状态(0表示未更年期,1表示已更年期)
size:肿瘤大小
grade:肿瘤分级
nodes:受累淋巴结数量
pgr:孕激素受体表达水平
er:雌激素受体表达水平
hormon:激素治疗(0表示否,1表示是)
rfstime:复发或死亡时间(以天为单位)
status:事件状态(0表示被截尾,1表示事件发生)
-
2.探索性分析
summary(gbsg)
结果展示:
age meno size grade nodes pgr er hormon rfstime status
Min. :21.00 Min. :0.0000 Min. : 3.00 Min. :1.000 Min. : 1.00 Min. : 0.0 Min. : 0.00 Min. :0.0000 Min. : 8.0 0:387
1st Qu.:46.00 1st Qu.:0.0000 1st Qu.: 20.00 1st Qu.:2.000 1st Qu.: 1.00 1st Qu.: 7.0 1st Qu.: 8.00 1st Qu.:0.0000 1st Qu.: 567.8 1:299
Median :53.00 Median :1.0000 Median : 25.00 Median :2.000 Median : 3.00 Median : 32.5 Median : 36.00 Median :0.0000 Median :1084.0
Mean :53.05 Mean :0.5773 Mean : 29.33 Mean :2.117 Mean : 5.01 Mean : 110.0 Mean : 96.25 Mean :0.3586 Mean :1124.5
3rd Qu.:61.00 3rd Qu.:1.0000 3rd Qu.: 35.00 3rd Qu.:2.000 3rd Qu.: 7.00 3rd Qu.: 131.8 3rd Qu.: 114.00 3rd Qu.:1.0000 3rd Qu.:1684.8
Max. :80.00 Max. :1.0000 Max. :120.00 Max. :3.000 Max. :51.00 Max. :2380.0 Max. :1144.00 Max. :1.0000 Max. :2659.0
4.2.3 无监督学习
# 特征选择(这里可以t检验的结果进行特征选择)
selected_features <- c("age", "meno", "size", "grade","pgr","er","hormon")
# 标准化数据
scaled_data <- scale(gbsg[, selected_features], center = TRUE, scale = TRUE)
# 使用k-means聚类算法
kmeans_result <- kmeans(scaled_data, centers = 4)
# 聚类结果
cluster_labels <- kmeans_result$cluster
# 将聚类结果赋值给gbsg
gbsg$cluster <- cluster_labels
4.2.3 随机森林
library(randomForest)
set.seed(1234)
# 随机分出训练集和测试集
trainIndex <- sample(1:nrow(gbsg), 0.8 * nrow(gbsg))
train <- gbsg[trainIndex,]
test <- gbsg[-trainIndex,]
# 构建随机森林模型
train$status <- as.factor(train$status)
rf_mod <- randomForest(status ~ ., data = train, ntree = 100)
# 获取模型预测的概率
pred_prob <- predict(rf_mod, newdata = test, type = "prob")
# 计算真阳性率和假阳性率
roc <- pROC::roc(test$status, pred_prob[, 2])
# 绘制ROC曲线
plot(roc, main = "ROC Curve", print.auc = TRUE, auc.polygon = TRUE, grid = TRUE, legacy.axes = TRUE,col="blue")
从图中结果可以看出,AUC值比之前有所提升。补充说明一点,聚类的组数对结果影响很大,需要自己进行尝试。目前分类成四组的结果最佳。这里只是演示模型,不是真实的分析,真实的论文分析有很多的前置步骤和参数调优等!
五、总结
-
1.整合多模态数据:未来的研究可以将多模态数据(如基因表达数据、成像数据、临床数据等)结合起来,利用无监督学习方法来探索不同数据模态之间的关联,进一步提高乳腺癌的诊断和治疗水平。 -
2.增强学习和个性化治疗:利用强化学习的方法,可以根据患者的特定情况和治疗反应来制定个性化的治疗方案。通过利用无监督学习的特征提取能力,可以更好地预测患者响应不同治疗策略的机会。 -
3.数据隐私和安全:在使用乳腺癌数据时,需要注意保护患者的隐私和数据安全。未来的研究可以探索如何利用无监督学习方法来处理隐私敏感数据,以确保数据的安全和匿名性。 -
4.解释性和可解释性模型:尽管无监督学习方法能够对乳腺癌数据进行分析和建模,但其结果通常是难以解释和理解的。因此,未来的研究可以致力于开发能够提供解释性和可解释性的无监督学习模型,以揭示乳腺癌发展机制和相关因素,并为临床实践提供有用的解释和指导。 -
5.大规模和多中心数据整合:随着大规模乳腺癌数据集的积累和公开共享,未来的研究可以通过无监督学习方法来整合和分析多中心和大规模数据。这有助于提高乳腺癌研究的统计能力和数据驱动的发现,并推动精准医学的实现。 -
6.强调早期诊断和预防:无监督学习方法可以应用于乳腺癌早期诊断和预防的研究。通过对乳腺癌高风险群体的数据分析,可以发现早期乳腺癌的标志物和警示信号,并开发出更有效的筛查和预防策略。 -
7.整合临床和基础研究:将无监督学习方法与临床数据和基础研究相结合,有助于深入理解乳腺癌的发展和治疗反应过程。通过对乳腺癌组织样本的图像分析和临床数据的整合,可以揭示乳腺癌的细胞组织特征和患者预后情况。 -
8.跨领域合作:乳腺癌研究中的无监督学习方法需要跨越生物信息学、计算机科学和临床医学等多个领域的合作。未来的研究应强调跨学科的合作,促进乳腺癌研究中无监督学习的创新和应用。
因此,未来对乳腺癌研究中无监督学习的潜在研究方向和机会包括解释性模型、大规模数据整合、早期诊断、临床与基础研究的整合以及跨领域合作等,这些都将推动乳腺癌的早期诊断、个体化治疗和乳腺癌研究的进展。
总之,无监督学习在乳腺癌研究中具有广阔的应用前景。通过深入研究和发展无监督学习方法,可以更好地理解乳腺癌的复杂性,为乳腺癌的诊断和治疗提供理论依据。
参考文献:
-
Bray, F., Ferlay, J., Soerjomataram, I., Siegel, R. L., Torre, L. A., & Jemal, A. (2018). Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: A Cancer Journal for Clinicians, 68(6), 394-424. -
Weigelt, B., & Reis-Filho, J. S. (2009). Histological and molecular types of breast cancer: is there a unifying taxonomy? Nature Reviews Clinical Oncology, 6(12), 718-730. -
Davis, S., & Meltzer, P. S. (2007). GEOquery: a bridge between the Gene Expression Omnibus (GEO) and BioConductor. Bioinformatics, 23(14), 1846-1847.
*「未经许可,不得以任何方式复制或抄袭本篇文章之部分或全部内容。版权所有,侵权必究。」