目前,本人写的第二个R包pm3包的1.8版本已经正式在CRAN上线,用于3组倾向评分匹配,只能3组不能多也不能少。
可以使用以下代码安装
install.packages("pm3")

什么是倾向性评分匹配?倾向评分匹配(Propensity Score Matching,简称PSM)是一种统计学方法,用于处理观察研究(Observational Study)的数据,在SCI文章中应用非常广泛。在观察研究中,由于种种原因,数据偏差(bias)和混杂变量(confounding variable)较多,倾向评分匹配的方法正是为了减少这些偏差和混杂变量的影响,以便对实验组和对照组进行更合理的比较。
为什么需要做倾向评分匹配?
我们知道RCT的证据力度高,是因为对患者进行了严格的筛选。我们的回顾性研究都是过去的数据,很难像RCT一样进行严格的筛选出两组患者基线相近的基础资料,但我们可以通过倾向评分匹配把回归性的数据进行筛选,把基线资料相近的患者进行匹配,得到近似RCT的效果。
应用场景
1.基线资料不平
2.开展病例对照研究病阳性例数较少,如罕见病研究
3.将众多混杂因素变为一个变量:倾向值
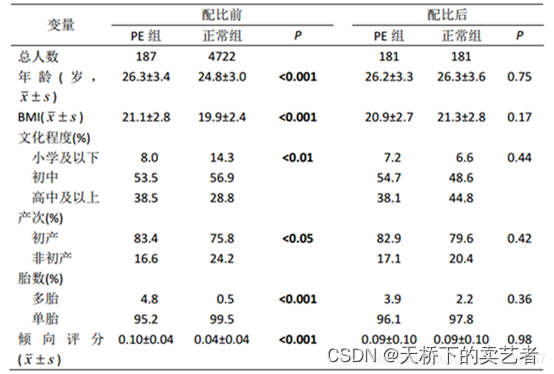
以下为一个实例,没进行匹配前两组患者基线资料相差很大,进行倾向评分匹配后,基线资料近似一致了
1.8版本修正了前面版本的一些错误,主要修正了两个问题:1是1.4版本的X变量如果是数字的话必须是1,2,3 ,1.8版本对变量进行了重编码,只要X是3分类就行,字符变量或者数字都可以,不在对数字有要求。2.是修正了对缺失数据的处理形式,进一步提高了上手性。下面我一一道来。
上一个版本中,X变量如果是数字来表示的话必须是1,2,3,使用0,1,2或者其他的数列都会报错。下面我们来试一下1.8版本,我先导入我们的早产数据,
bc<-read.csv("E:/r/test/zaochan.csv",sep=',',header=TRUE)
bc$race<-ifelse(bc$race=="black",0,ifelse(bc$race=="white",1,2))
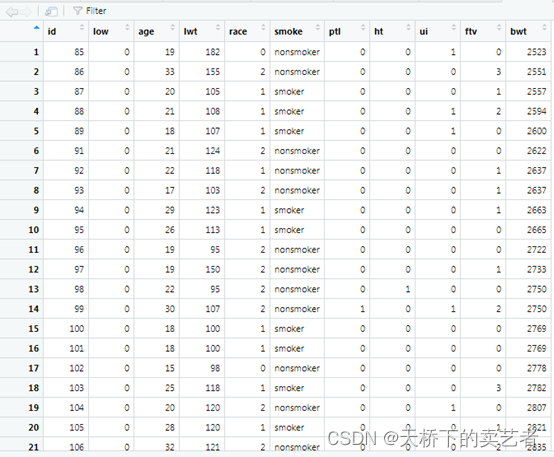
我们可以看到race这个变量现在是0,1,2,以往1.4版本是做不了的,必须改成1,2,3,现在新版本就没有这个问题了
g<-pm3(data=bc,x="race",y="low",covs=c("age","lwt","ptl"))
一句代码结果就出来了,较原来方便了许多。再来就是改变了缺失值的处理方式,因为粉丝收集的数据大都有缺失值,有些变量的缺失值还是挺多的, 1.4版本对缺失值的处理可以说是简单粗暴,直接删除,但是数据中如果不需要使用的变量缺失值很多,会导致了大量数据被删除,导致我们的分层变量数会减少,导致X变量的数变少,1.8版本引入了一个变量psid,先进行匹配,后期在进行数据匹配,这样使数据利用效率大大提高,下面一个例子说明,1.4版本:
library(pm3)
bc<-read.csv("E:/r/fensi/data8.csv",sep=',',header=TRUE)
g<-pm3(data=bc,x="group",y="solidfoodintroductionlessthan6m",
covs=c("femaleage","gestationalweeks","childgender"),factor=c("childgender"))
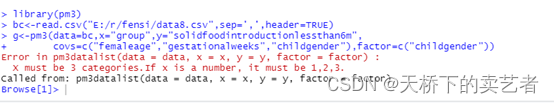
主要是这些用不到的变量缺失太多了
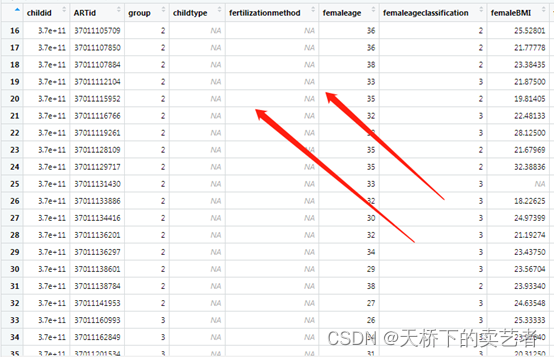
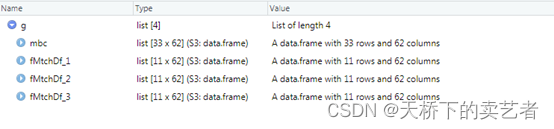
1.8版本避开了这个问题,还是同样的数据,同样的代码
bc<-read.csv("E:/r/fensi/data8.csv",sep=',',header=TRUE)
g<-pm3(data=bc,x="group",y="solidfoodintroductionlessthan6m",
covs=c("femaleage","gestationalweeks","childgender"),factor=c("childgender"))
再来说说factor这个参数,这个参数就是把数据中的变量变成因子,在上面的例子中,factor=c(“childgender”),其实就是相当于
bc$childgender<-as.factor(bc$childgender)
如果你在前面(计算前)就已经把分类变量转成因子了,factor不填就行了。
OK,新版本改动其实在构架来说还是蛮大的,但是做起来应该更简单了,如果有什么问题或建议,欢迎你来告诉我。