题目:Retrieval-based Controllable Molecule Generation
文章地址:https://openreview.net/pdf?id=vDFA1tpuLvk
代码地址:GitHub - NVlabs/RetMol: A new retrieval-based framework for controllable molecule generation.
主题:今天我们为大家分享一篇来自英伟达和莱斯大学研究团队的发表于“ICLR2023”的一篇分子生成的文章,作者提出了一种基于检索的分子生成框架“RetMol”,它可以使用仅满足部分目标属性的少量分子生成出满足所有所需属性的分子。
一、摘要
药物发现是一个复杂的多目标问题,生成出满足多种性质的分子在药物发现过程中非常重要。现有方法需要使用大型数据集进行的训练/微调,这在真实世界的生成任务中通常是不现实的。在本研究中,作者提出了一种新的基于检索的可控分子生成框架。不需要特定任务的微调,在数据稀缺的情况下(活性分子有限)容易推广到各种生成任务:仅仅需要使用一小组示例分子来引导模型就能生成出满足给定设计目标的分子。作者设计了一个新的自监督目标训练的检索机制,融合了检索库中的样本分子和输入分子,并预测输入分子附近的相似分子;提出了一个迭代改进过程,动态更新检索数据库以获得更好的泛化性能。从结果上看,在各种任务中,从简单的设计标准到用于设计与SARS-CoV-2主蛋白相结合的引物化合物的现实场景,作者提出的方法比之前的方法具有更好的性能和更广泛的适用性。
二、方法

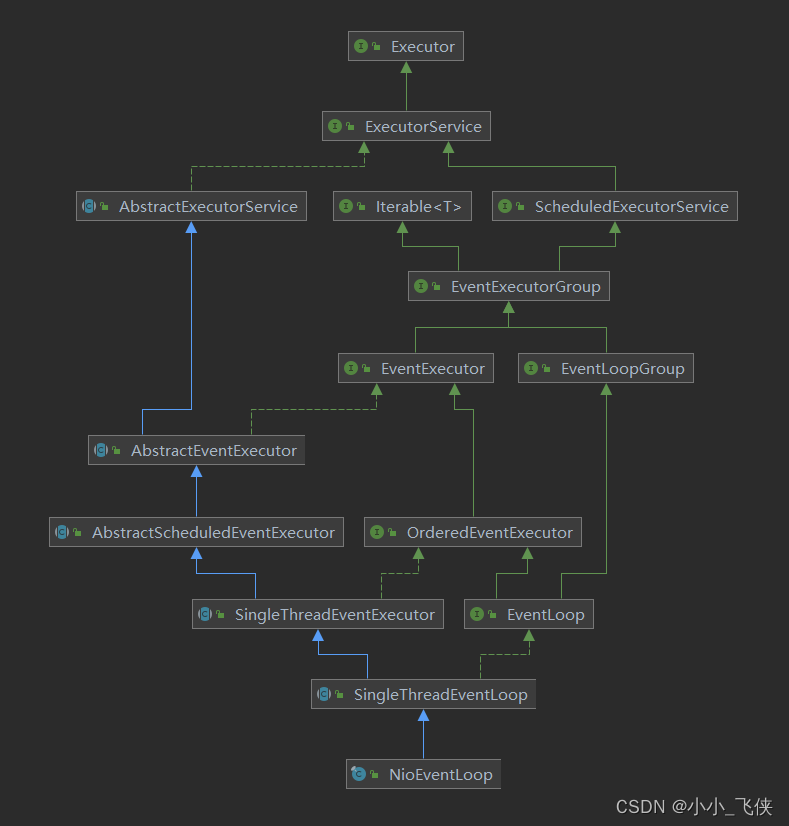
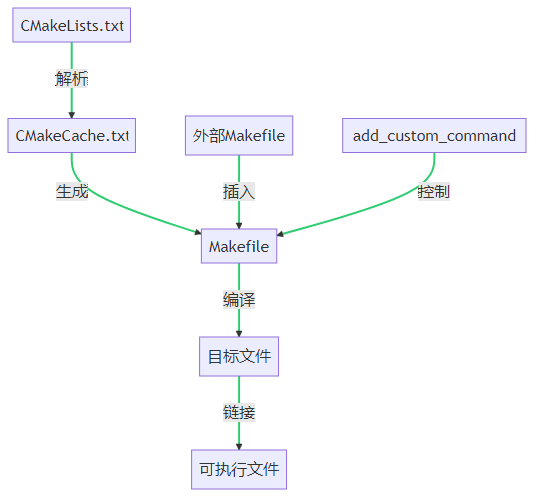
图1:RetMol框架。该框架结合了检索模块(分子检索器和信息融合)和预训练的生成模型(编码器Encoder和解码器Decoder)。上图示例展示了如何在各种设计条件下优化现有潜在药物法匹拉韦(Favipiravir)的结合力,以更好地治疗COVID-19病毒(SARS-CoV-2主要蛋白质,PDB ID:7L11)。
如上图所示,RetMol框架将一个轻量级检索机制Retriever插入预训练的编码器-解码器生成模型中。对于每个任务,作者首先构建了一个满足部分设计标准的样本分子组成的检索数据库Retrieval database。在给定待优化的输入分子Input molecule之后,检索器模块Retriever会从数据库中检索少量与之相关的示例分子,然后由预先训练好的生成模型的编码器Encoder将它们与输入分子转换为Retrieved embeddings 和 Input embedding。接下来,信息融合模块Information fusion将示例分子的嵌入与输入分子嵌入融合Fused embedding,以引导生成(通过预训练生成模型中的解码器)满足所需性质的分子Output molecule。
信息融合模块Information fusion是RetMol中唯一需要训练的部分。为了训练,作者提出了一个新的自监督目标(即预测输入分子的最近邻),以更新融合模块,使RetMol能够在没有特定任务训练/微调的情况下适用于各种生成任务。在模型的推理阶段,作者提出了一种通过迭代的推理过程,动态更新生成的分子和检索数据库,从而提高了生成质量和多样性。
1)编码器-解码器生成模型主干
预训练分子生成模型构成了RetMol的骨干,它连接了连续嵌入和原始分子表示。编码器将传入的分子编码成嵌入embedding,解码器从嵌入embedding中生成新分子。RetMol不关心底层编码器和解码器架构的设计,使其能够与各种生成模型和分子表示一起使用。在这项工作中,作者使用SMILES字符串表示分子和ChemFormer模型作为主干,其中ChemFormer模型是在十亿级别的ZINC数据集上训练的BART变体。
2)检索数据库(Retrieval database)
检索数据库Retrieval database包含满足各种所需性质的分子,可以引导输入分子向设计标准方向发展,从而生成出具有目标性质的分子,因此对于可控生成非常重要。了解哪些分子符合设计标准以及如何选择满足设计标准的分子在RetMol中发挥着重要作用。此外,作者发现仅有少量分子(例如23个)的数据库就已经可以提供强有力的控制信号,这使得作者的方法可以通过快速检索数据库来轻松适应各种任务。此外,在推理过程中可以动态更新检索数据库,即新产生的分子可以扩充检索数据库以实现更好的泛化效果。
3)分子检索
虽然在生成过程中可以使用整个检索数据库,但出于计算原因(例如内存和效率),选择少量最相关的分子来提供更准确的引导更为可行。因此作者设计了一个基于启发式的检索器,以检索出最适合给定控制设计标准的分子:首先构建一个“可行”分子集合,其中包含满足所有给定约束条件的分子;如果此集合大于常数K(希望检索的最大分子数,本文使用K=10),则选择具有最佳平均属性得分的K个分子,否则,通过一次移除一个约束条件来构造松弛可行集,直到松弛可行集大于K,选择最近删除的属性约束的最佳得分的K个分子。无论上述哪种情况,检索器都会检索比输入更理想的性质的例分子,并引导生成朝着给定的设计标准发展。一般来说,更复杂的检索器设计对生成更好性质的分子可能会更有效,作者把它留作未来的工作。
4)信息融合(Information fusion)
该模块可以使检索的分子将输入分子朝着目标设计标准变化,它通过一个轻量级的可训练标准交叉注意力机制来合并输入分子和检索到的分子的嵌入得到融合嵌入:
融合嵌入包含从检索到的样本分子和输入分子中提取的所需属性的信息,该信息用作解码器的输入。
5)通过预测输入分子最近的邻居进行自监督训练
传统的训练目标是重建输入分子,但在作者的情况下这种目标不适用,因为重建输入分子不会依赖于检索到的样本分子,这会导致输入分子无法利用到样本分子的优质属性信息。为了使RetMol学习使用样本分子进行可控生成,作者提出了一种新的自监督训练方案,其目标是预测输入分子的最近邻:
【\mathcal{L}(\theta)=\sum_{i=1}^{\mathcal{B}} \operatorname{CE}\left(\operatorname{Dec}\left(f_{\mathrm{CA}}\left(\boldsymbol{e}_{\mathrm{in}}^{(i)}, \boldsymbol{E}_{r}^{(i)} ; \theta\right)\right), x_{1 \mathrm{NN}}^{(i)}\right)】
其中CE是交叉熵损失函数,【x_{1NN}】代表输入分子的最近邻,B是批处理大小,i对输入分子进行索引。检索到的样本分子集合由输入分子的其余K-1个最近邻组成。在训练期间,作者冻结了预训练编码器和解码器的参数,只更新信息融合模块
【f_{CA}】中的参数,从而使训练轻量级和高效。此外,作者使用整个训练数据集作为检索数据库,并通过与输入分子的最佳相似度来检索样本分子,因此训练不是针对特定任务的,而是迫使融合模块
参与以相似性为代理标准的可控生成。在推理时,使用上述训练目标和使用相似度的代理准则训练的模型能够推广到具有不同设计准则的不同生成任务,并且比使用传统重建目标训练的模型性能更好。为了更加有效的训练,作者用高效的近似KNN算法预先计算了所有分子的嵌入及其两两之间的相似性
6)通过迭代改进策略进行推理
作者提出了一种迭代改进策略,以在利用样本分子的隐式指导进行可控生成时获得改进的结果。该策略通过替换输入分子并更新检索数据库以获取新生成的分子来工作。这种迭代更新在许多其他可控生成方法中是常见的,此外,如果在推理过程中固定而不进行更新检索数据库,则作者提出的方法会受到检索数据库中分子最佳性质值的限制,这极大地限制泛化能力以及在某些生成场景下的表现,例如无约束优化,因此作者建议在迭代中动态更新检索数据库。
首先通过添加高斯噪声来随机扰动融合嵌入M次,并从每个扰动嵌入中贪心地解码一个分子,以获得M个生成的分子,根据任务特定的设计标准对这些分子进行评分。然后,如果此集合中最佳分子的分数优于输入分子的分数,则将输入分子替换为最佳分子。同时,如果它们的分数比检索数据库中的最低分数更好,则将其余分子添加到检索数据库中。如果没有生成的分子的分数优于输入分子或检索数据库中最低分数,则输入分子和检索数据库保持不变,以供下一次迭代使用。如果达到最大允许的迭代次数或生成符合所需标准的成功分子便会停止。
Training objectives. We evaluate how the proposed self-training objective in Sec. 2.2 affects the quality of generated molecules in the unconditional generation setting. Table 4 (left) shows that, training with our proposed nearest neighbor objective (i.e., predict NN) indeed achieves better generation performance than the conventional input reconstruction objective (i.e., recon. input).
训练目标。我们评估了第2.2节中提出的自训练目标如何影响无条件生成设置下生成的分子质量。表格4(左)显示,使用我们提出的最近邻目标进行训练(即预测NN),确实比传统的输入重构目标进行训练(即重构输入),获得了更好的生成性能。
三、实验结果
1)QED和Penalized logP优化
QED优化:这是一个带约束的分子优化问题,目标是在满足与目标分子相似度大于一定阈值的条件下,优化目标分子的QED性质成功率。如图2(a)所示,RetMol的成功率远高于其他方法。
Penalized logP优化:这是一个带约束的分子优化问题,目标是在满足与目标分子相似度大于一定阈值的条件下,优化目标分子的Penalized logP性质。如图2(b)所示,RetMol能有效提高分子的此项性质。

表格1:(a)在相似性
【\delta】下满足QED条件的分子生成成功率;(b)在相似性
【\delta】下提升Penalized logP条件的分子生成成功率;
2)多目标优化
多目标优化:这是一个带约束的分子优化问题,目标是在满足分子的QED性质和SA性质符合阈值内的情况下提高生成分子对GSK3β和JNK3两个靶点的亲和性。
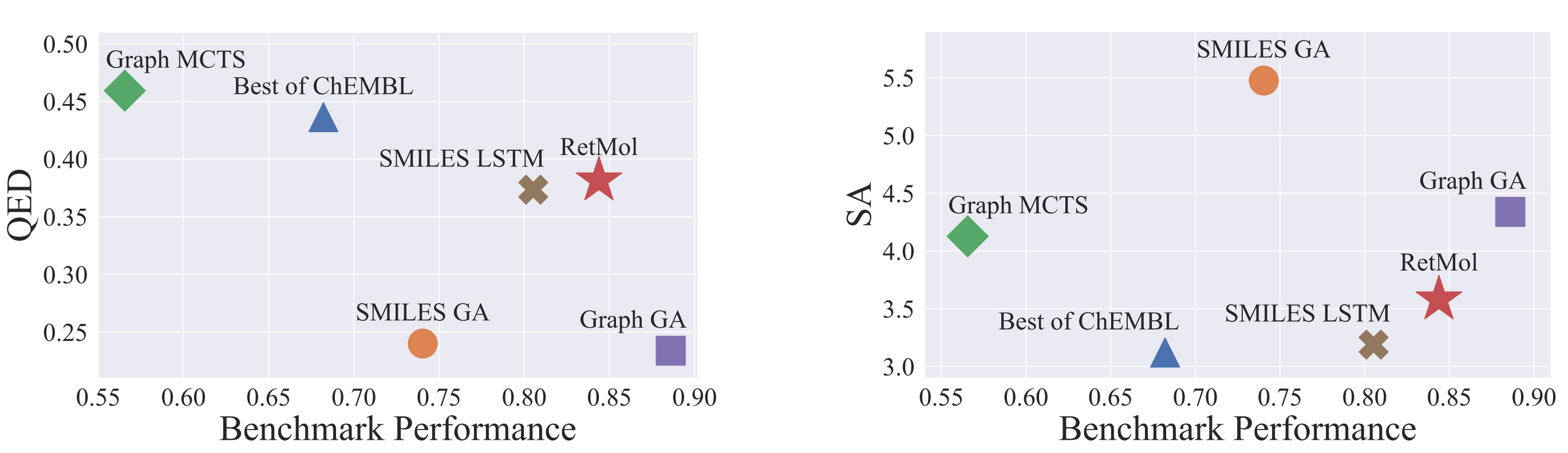
图2:在Guacamol基准测试上进行多属性优化(MPO)任务时与SOTA的比较。左图:QED(↑)与平均基准性能(↑)比较。右图:SA(↓)与平均基准性能(↑)之比较。RetMol在提高基准性能的同时保持所生成分子的可合成性(SA)和药物样性(QED)方面实现了最佳平衡。

表格2:在优化四个属性(QED、SA以及两种与GSK3β和JNK3靶点的结合亲和力)的任务中生成的分子的成功率、新颖性和多样性。
3)新冠药物优化
新冠药物优化:为了展示基于检索的框架在药物发现前沿的应用性,作者将其应用于改善对SARS-CoV-2主蛋白酶(Mpro,PDB ID:7L11)的现有弱抑制剂的抑制作用。作者使用23种已知抑制剂作为检索数据库,并选择8个最弱的抑制剂作为输入,设计了一个优化任务,以最大化生成分子与Mpro之间的docking score。具体结果如上表格所示,可以看到RetMol相比于对比方法,药物优化成功率以及优化力度都要高很多。
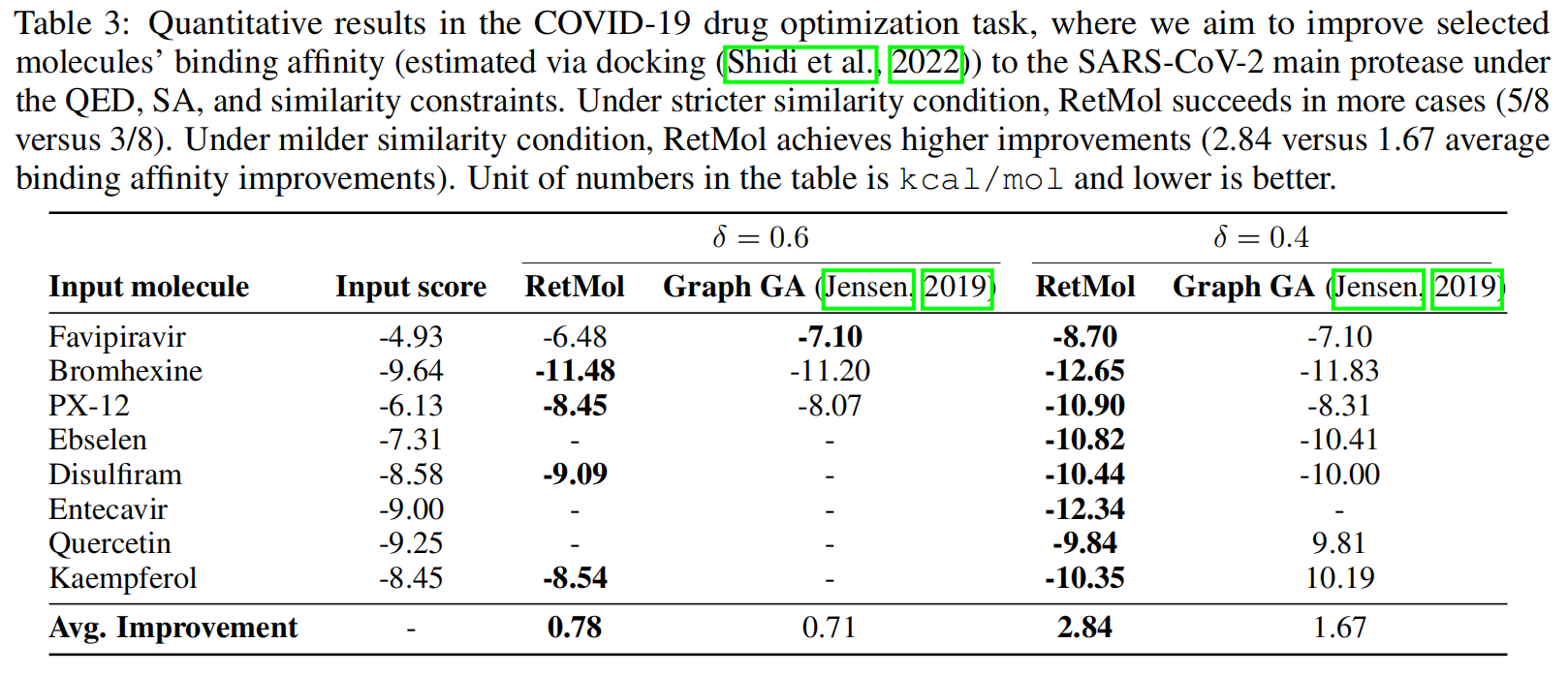
表格3:在COVID-19药物优化任务中,作者通过QED、SA和相似性约束来改善所选分子与SARS-CoV-2主要蛋白酶的结合亲和力(通过docking进行估计)。在更严格的相似性条件下,RetMol成功率更高(5/8与3/8相比)。在较轻的相似性条件下,RetMol实现了更高的改进(2.84与1.67平均结合亲和力改进值相比)。
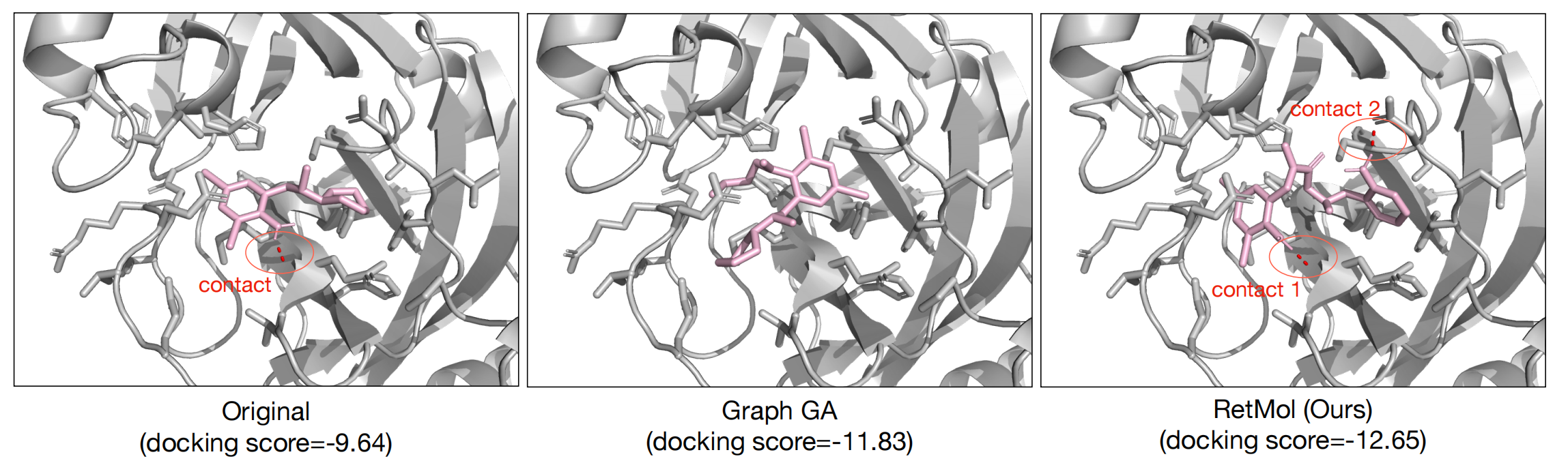
图3:比较RetMol和Graph GA方法在优化与SARS-CoV-2主要蛋白酶结合的原始抑制剂Bromhexine的δ=0.6情况下的三维可视化。RetMol优化的抑制剂具有更多的极性接触(红色线),与原化合物相比也具有更不同的结合模式,这与定量结果相一致。
四、结论:
作者提出了一种基于检索的可控分子生成框架RetMol。通过将检索模块与预训练的生成模型结合,RetMol利用从特定任务的检索数据库中检索的示例分子来引导生成模型生成满足所需设计标准的新分子。RetMol非常灵活,不需要任务特定的微调。作者展示了RetMol在各种基准任务和SARS-CoV-2病毒的真实抑制剂设计任务中的有效性,在每种情况下与现有方法相比均取得了最先进的性能。