rWCVP 的函数清单
- 1. get_area_name()
- 用法
- 参数
- 值
- 详介
- 例子
- 2. get_wgsrpd3_codes()
- 用法
- 参数
- 值
- 详介
- 例子
- 3. powo_map()
- 用法
- 参数
- 值
- 4. powo_pal(), scale_color_powo(), scale_colour_powo(), scale_fill_powo()
- 用法
- 参数
- 值
- 5. redlist_example
- 用法
- 格式
- 资源
- 6. taxonomic_mapping
- 用法
- 格式
- 资源
- 7. wcvp_checklist()
- 用法
- 参数
- 值
- 详介
- 8. wcvp_distribution()
- 用法
- 参数
- 值
- 详介
- 9. wcvp_distribution_map()
- 用法
- 参数
- 值
- 详介
- 例子
- 10. wcvp_match_exact()
- 用法
- 参数
- 值
- 另请参阅
- 例子
- 11. wcvp_match_fuzzy() phonetic_match() edit_match()
- 用法
- 参数
- 值
- 详介
- 另请参阅
- 例子
- 12. wcvp_match_names()
- 用法
- 参数
- 值
- 详介
- 另请参阅
- 例子
- 13. wcvp_occ_mat()
- 语法
- 参数
- 值
- 详介
- 例子
- 14. wcvp_reformat()
- 用法
- 参数
- 值
- 详介
- 15. wcvp_summary()
- 用法
- 参数
- 值
- 详介
- 例子
- 16. wcvp_summary_gt()
- 用法
- 参数
- 值
- 例子
- 17. wgsrpd3
- 用法
- 格式
- 资源
- 18. wgsrpd_mapping
- 用法
- 格式
- 资源
1. get_area_name()
根据地区编码获得地区名称
用法
get_area_name(area_codes)
参数
- area_codes:包含要映射到名称的代码集的字符向量
值
字符。长度为1的向量,带有3级区域集的名称,或者(如果该区域集没有名称)代码的输入向量。
详介
与get_wgsrpd3_codes函数执行相反的功能。适用于压缩代码集,例如文件名、绘图和表格格式。
例子
get_area_name(get_wgsrpd3_codes("Brazil"))
ℹ Matches to input geography found at Country (Gallagher) and Region (Level 2)
[1] "Brazil"
2. get_wgsrpd3_codes()
提取WGSRPD3级编码
用法
get_wgsrpd3_codes(geography, include_equatorial = NULL)
参数
- geography:字符变量。要转换为3级代码的地理位置。可能是WGSRPD区域(3级)、地区(2级)或洲(1级)、国家(政治)或半球(“北半球”、“南半球”或“赤道”)
- include_equatorial:逻辑变量。包括跨越赤道的3级区域?默认为NULL,这将生成一条消息并包括这些区域。如果地理不是半球,则忽略。
值
具有属于该地理区域的区号(级别3)的字符。
详介
国家绘图遵循加Gallagher et al. (2020)。重要的是,这意味着一些海外领土在此系统中不被视为国家的一部分,例如加那利群岛被指定为它们自己的 3 级区域,而不是此映射中西班牙的一部分。在不明确的地方,可以使用View(wgsrpd_mapping)来研究映射。
例子
get_wgsrpd3_codes("Brazil")
ℹ Matches to input geography found at Country (Gallagher) and Region (Level 2)
[1] "BZC" "BZE" "BZL" "BZN" "BZS"
3. powo_map()
为给定的范围和范围质心绘制 POWO 样式地图。
用法
powo_map(range_sf, centroids_sf)
参数
- range_sf:等高多边形的简单要素(sf)数据框
- centroids_sf:范围质心的简单要素(sf)数据框
值
该范围的ggplot地图
4. powo_pal(), scale_color_powo(), scale_colour_powo(), scale_fill_powo()
POWO网站上展示的范围地图有一个固定的、离散的调色板,它基于一个地区的分类群类型。
用法
powo_pal()
scale_color_powo(...)
scale_colour_powo(...)
scale_fill_powo(...)
参数
- …:传递给discrete_scale的参数
- palette:一个调色板函数,当用单个整数参数(标尺中的级别数)调用时,返回它们应该取的值(例如scales::hue_pal())
- limits:下列之一:
1. 默认为NULL
2. 定义了尺度可能的值和顺序的字符向量
3. 接受现有(自动)值并返回新值的函数。也接受rlang lambda函数表示法。 - drop:是否应从量表中省略未使用的因子水平?默认值TRUE使用数据中出现的级别;FALSE使用因子中的所有级别。
- na.translate:与连续刻度不同,离散刻度可以很容易地显示缺失值,默认情况下会这样做。如果要从离散刻度中删除缺失值,请指定na.translate = FALSE。
- scale_name:应用于与此刻度关联的错误消息的刻度的名称。
- name:尺度名称。用于轴或图例标题。如果是waiver(),默认值,尺度名称从首次绘图时获得。如果为NULL,图例标题将被忽略。
- labels:以下之一:
1. NULL表示没有标签
2. waiver() 用于转换对象计算的默认标签
3. 提供标签的字符向量(必须与 breaks 等长)
4. 表达式向量(必须与 breaks 等长)。详见 ?plotmath
5. 一个函数,它将breaks作为输入,并将标签作为输出返回。也接受rlang lambda函数表示法。 - guide:用于创建指南或其名称的函数。详见 guides()
- super:用于构建标尺的父类
值
字符。与POWO相匹配的名称和十六进制值的向量。
5. redlist_example
名称匹配的示例数据集。一个包含20个红名单评估样本的数据集,用于名称匹配
用法
redlist_example
格式
具有 20 行和 4 个变量的数据框:
- assessmentId:红色名录标识符
- scientificName:类群名称
- redlistCategory:红色名录威胁类别
- authority:类群命名人
资源
从 https://www.iucnredlist.org/ 下载并采样
6. taxonomic_mapping
用于将植物科映射到目或更高分类的数据。包含更高分类(被子植物、裸子植物、蕨类植物和石松植物)和 WCVP 中每个科的顺序的数据集。
用法
taxonomic_mapping
格式
一个包含457行和3个变量的数据框架:科、目和更高分类阶元
资源
PPG I 中的蕨类植物和石松类植物分类学。来自APG IV的被子植物分类学。Forest 等人的裸子植物分类学。
7. wcvp_checklist()
从 WCVP 生成物种清单
用法
wcvp_checklist(
taxon = NULL,
taxon_rank = c("species", "genus", "family", "order", "higher"),
area_codes = NULL,
synonyms = TRUE,
render_report = FALSE,
native = TRUE,
introduced = TRUE,
extinct = TRUE,
location_doubtful = TRUE,
hybrids = FALSE,
infraspecies = TRUE,
report_filename = NULL,
report_dir = NULL,
report_type = c("alphabetical", "taxonomic"),
wcvp_names = NULL,
wcvp_distributions = NULL
)
参数
- taxon:字符变量。指包含的类群。默认为NULL(不进行类群筛选,所有类群)
- taxon_rank:字符变量。“species”, “genus”, “family”, “order” or "higher"之一,指定 taxon的分类阶元。除非 taxon为 NULL,否则必须指定
- area_codes:字符变量。单个或多个WGSRPD3级区域代码。默认为NULL(全球范围)
- synonyms:逻辑变量。在清单中包含异名。默认为TRUE
- render_report:逻辑变量。将清单呈现为标记语言报告。默认为FALSE
- native:逻辑变量。包含未标记为 introduced,extinct或doubful的物种发现记录
- introduced:逻辑变量。包含标记为引入的物种发现记录。默认为TRUE
- extinct:逻辑变量。包含标记为灭绝的物种发现记录。默认为TRUE
- location_doubtful:逻辑变量。包含标记为产地存疑的物种发现记录。默认为TRUE
- hybrids:逻辑变量。清单包含杂交种。默认为FALSE
- infraspecies:逻辑变量。清单包含亚种。默认为TRUE
- report_dir:字符变量。HTML文件的存储路径。用户必须提供
- report_type:字符变量;alphabetical或taxonomic之一。生成的清单按字母顺序或分类顺序排列。默认为alphabetical
- wcvp_names:从WCVP 版本7或更新中获得的分类名称数据集。如果为NULL(默认),将从rWCVPdata::wcvp_names加载名称
- wcvp_distributions:从WCVP 版本7或更新中获得的分布区数据集。如果为NULL(默认),将从rWCVPdata::wcvp_distributions加载名称
值
返回筛选数据后的数据集,如果 render_report=TRUE,还有一个HTML报告文件
详介
使用synonyms参数可以只返回接受名。如果synonyms=TRUR,则废弃名、不合法名和其他非接受名都会返回(例如,清单并不限制名称:taxon_status==“Synonym”)。*rWCVP**提供两种清单类型:字母顺序型和分类顺序型。在字母顺序型清单中,所有名称都按照字母顺序排列,接受名加粗而异名接在接受名后。在分类顺序型清单中,名称按接受名分组,异名列在对应接受名下方。两种类型清单都包含命名人、参考文献和分布信息,注意科级标头仅在字母顺序清单中支持。
8. wcvp_distribution()
生成物种、属、科的空间分布对象*
用法
wcvp_distribution(
taxon,
taxon_rank = c("species", "genus", "family", "order", "higher"),
native = TRUE,
introduced = TRUE,
extinct = TRUE,
location_doubtful = TRUE,
wcvp_names = NULL,
wcvp_distributions = NULL
)
参数
- taxon:字符变量。要映射的分类单元。必需的。
- taxon_rank:字符变量。“species”, “genus”, “family”, “order” or "higher"之一。指定taxon的分类阶元
- native:逻辑变量。包含原生分布区?默认为TRUE
- introduced:逻辑变量。包含引种分布区?默认为TRUE
- extinct:逻辑变量。包含灭绝分布区?默认为TRUE
- location_doubtful:逻辑变量。包含存疑区?默认为TRUE
- wcvp_names:从WCVP 版本7或更新中获得的分类名称数据集。如果为NULL(默认),将从rWCVPdata::wcvp_names加载名称
- wcvp_distributions:从WCVP 版本7或更新中获得的分布区数据集。如果为NULL(默认),将从rWCVPdata::wcvp_distributions加载名称
值
包含分类单元范围多边形的简单特征 (sf) 数据框。
详介
当taxon_rank指定高于物种阶元,那么返回整个组的分布区,而不是该组中单个物种的分布区。当改变选项时也适用,比如,不管native=TURE或native=FALSE,如果在原生分布区外有引入发现记录,也会包含紧取。要分辨原生分布区内的灭绝、引种或存疑记录,可以使用wcvp_summary和wcvp_occ_mat
9. wcvp_distribution_map()
绘制物种、属、科的分布地区
用法
wcvp_distribution_map(
range,
crop_map = FALSE,
native = TRUE,
introduced = TRUE,
extinct = TRUE,
location_doubtful = TRUE
)
参数
- range:由wcvp_distribution返回的简单要素(sf)数据集
- crop_map:逻辑变量。裁剪地图至分布区?默认为FALSE
- native:逻辑变量。包含原生分布区?默认为TRUE
- introduced:逻辑变量。包含引种分布区?默认为TRUE
- extinct:逻辑变量。包含灭绝分布区?默认为TRUE
- location_doubtful:逻辑变量。包含存疑记录分布区?默认为TRUE
值
由ggplot2::ggplot绘制的分布区
详介
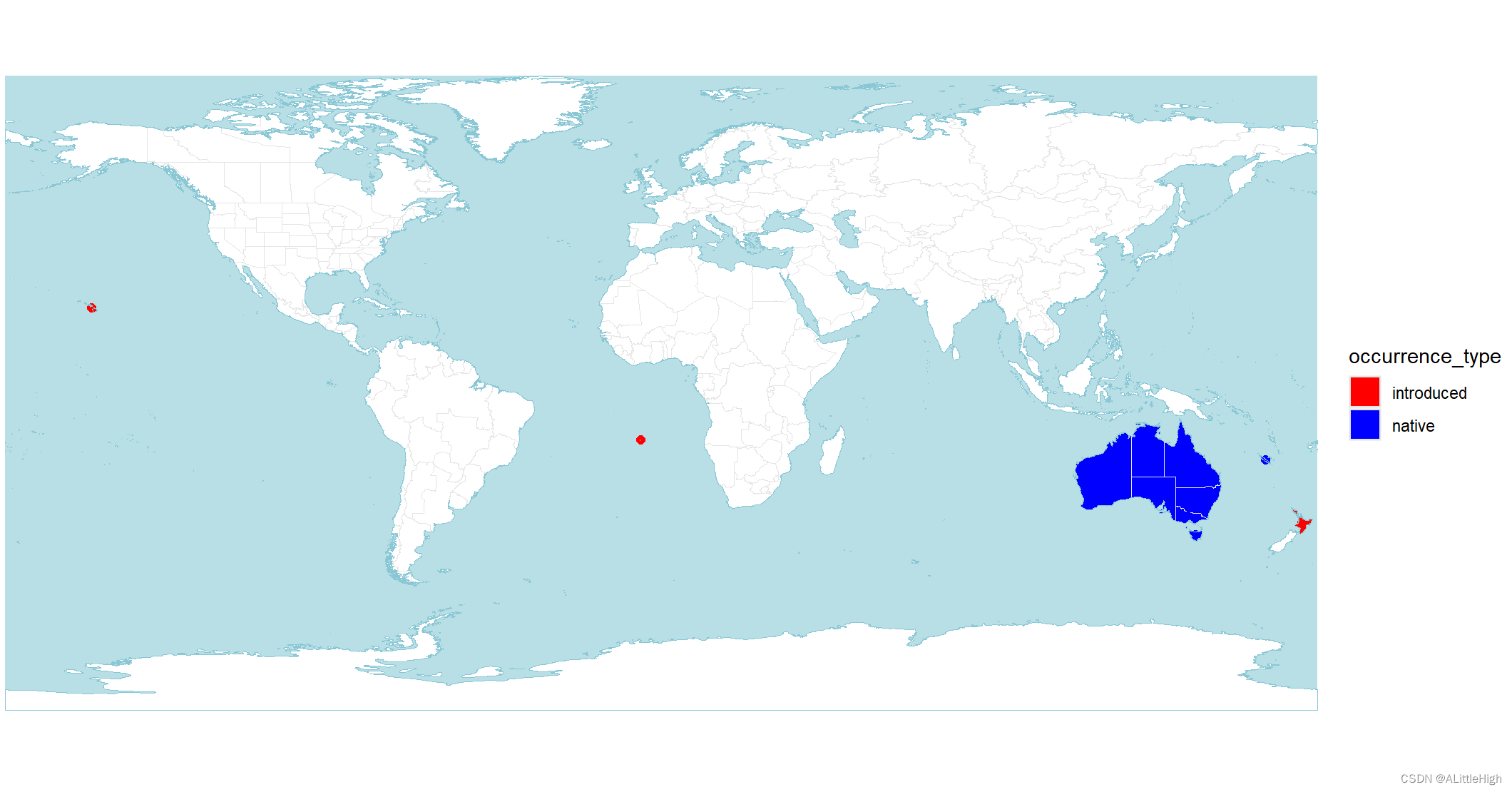
世界植物 (POWO; https://powo.science.kew.org/) 使用的配色方案镜像,其中绿色是原生的,紫色是引入的,红色是灭绝的,橙色是可疑的。有关如何使用自定义颜色的信息,请参见示例。
例子
wcvp_distribution_map(wcvp_distribution("Callitris", taxon_rank = "genus"))
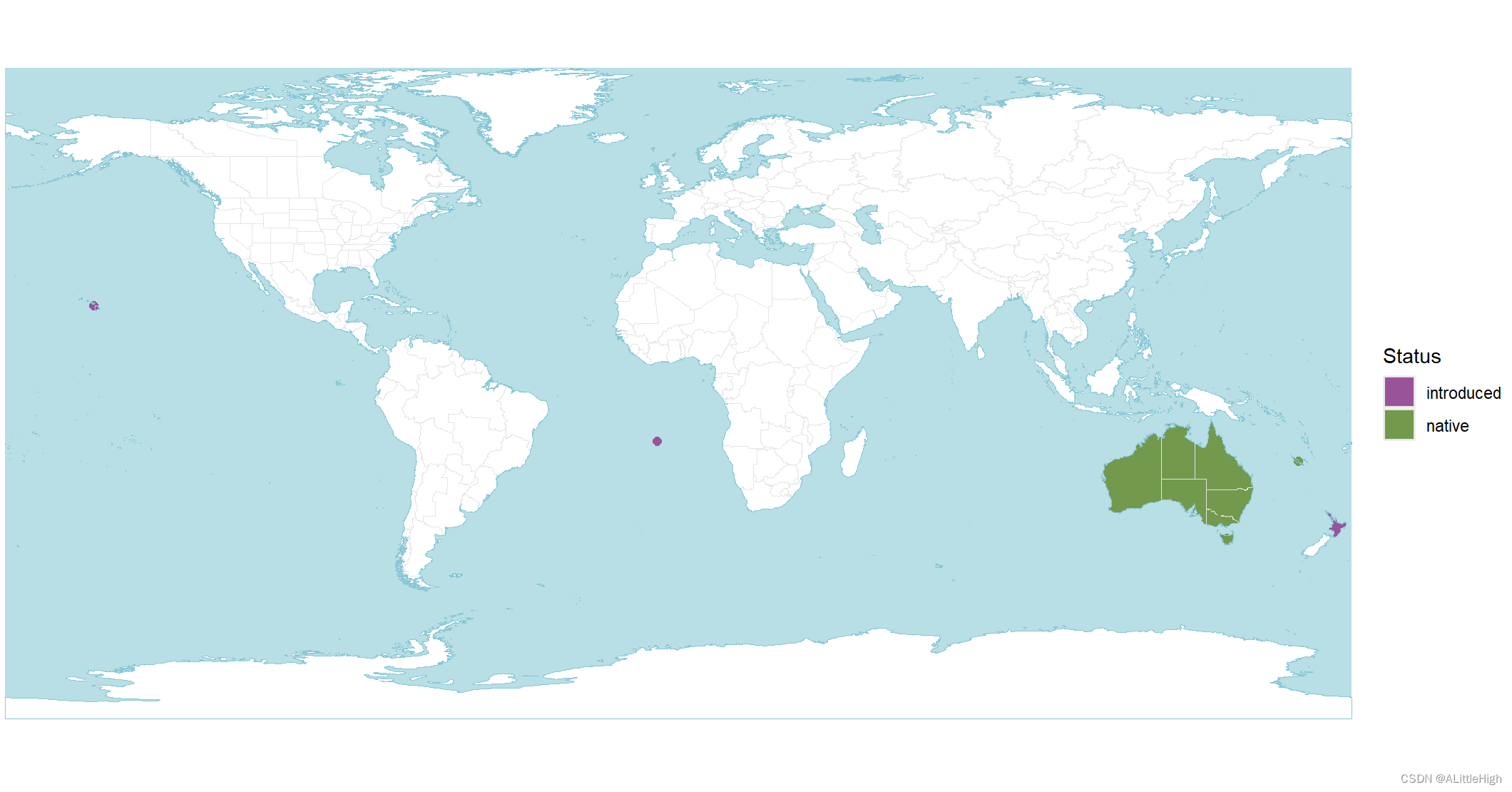
wcvp_distribution_map(wcvp_distribution("Callitris", taxon_rank = "genus"), crop_map = TRUE)
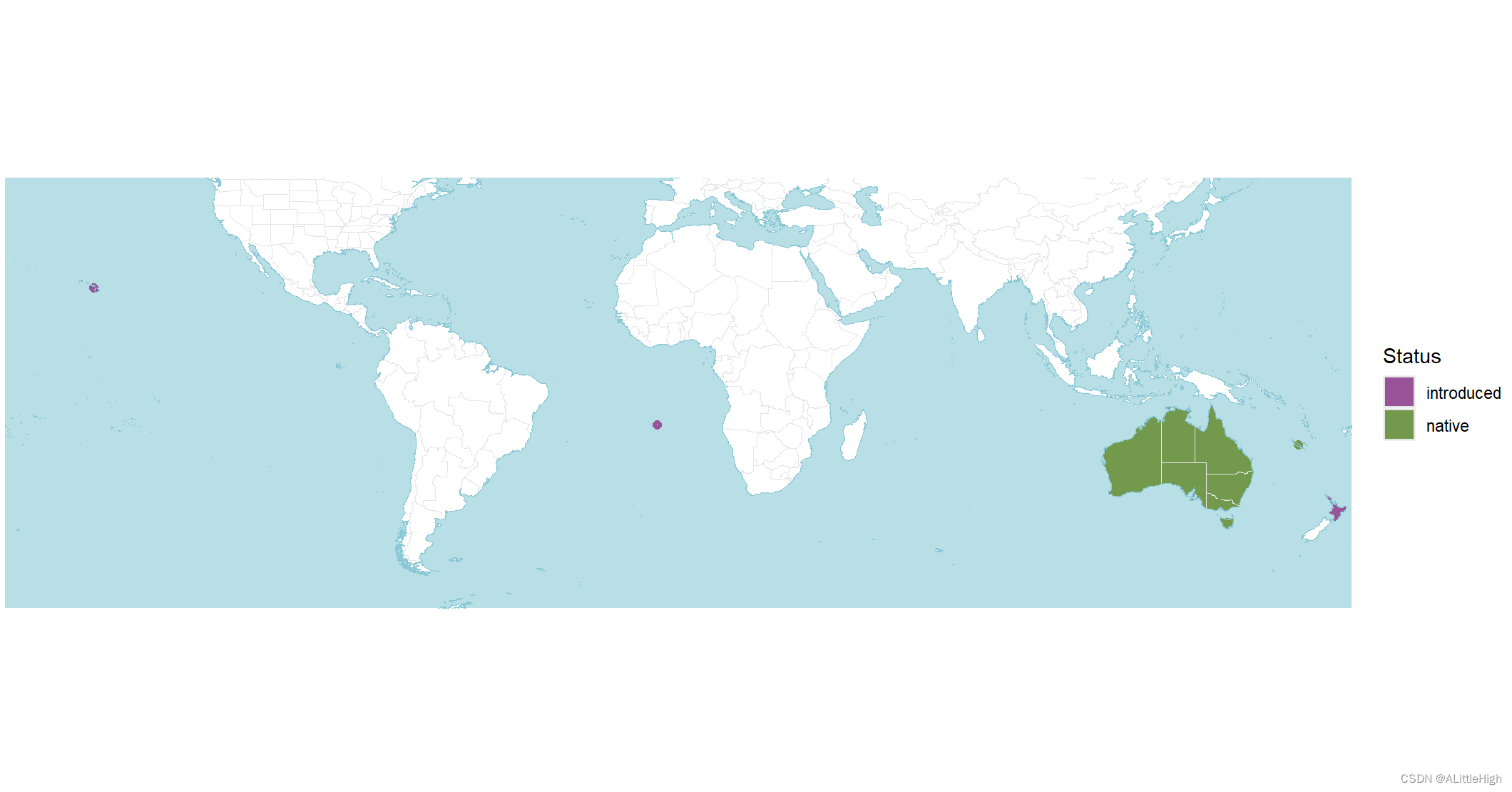
wcvp_distribution_map(wcvp_distribution("Callitris", taxon_rank = "genus")) +
ggplot2::scale_fill_manual(values = c("red", "blue")) +
ggplot2::scale_colour_manual(values = c("red", "blue"))
10. wcvp_match_exact()
与 WCVP 完全匹配。名称与 WCVP 的精确匹配,可选择使用作者字符串来优化结果。
用法
wcvp_match_exact(names_df, wcvp_names, name_col, author_col = NULL, id_col)
参数
- names_df:用于匹配的名称数据框。
- wcvp_names:从WCVP 版本7或更新中获得的分类名称数据集。如果为NULL(默认),将从rWCVPdata::wcvp_names加载名称
- name_col:字符变量。names_df中用于匹配类群名称的列
- author_col:names_df中含有名称命名人、用于辅助匹配的列。设置为NULL让匹配忽略命名人字符串
- id_col:names_df中含有发现记录id的列
值
将WCVP中的结果与names_df中的原始数据进行匹配。
另请参阅
** wcvp_match_fuzzy(), wcvp_match_names()**
例子
wcvp_names = rWCVPdata::wcvp_names
wcvp_match_exact(redlist_example, wcvp_names = wcvp_names, "scientificName", author_col = "authority", id_col = "assessmentId")
# A tibble: 20 × 16
assessmentId scientificName redlistCategory authority match_type
<dbl> <chr> <chr> <chr> <chr>
1 11081542 Antimima quart… Least Concern (Dinter)… NA
2 19395021 Avena hybrida Data Deficient Peterm. Exact (wi…
3 64135503 Citrus garrawa… Least Concern F.M.Bail… NA
4 189601563 Croton campanu… Endangered Caruzo &… Exact (wi…
5 115968141 Cynanchum free… Endangered (N.E.Br.… NA
6 11047751 Echinacanthus … Vulnerable H.S.Lo &… NA
7 11001316 Geissanthus pi… Endangered (Lundell… NA
8 126598076 Juglans pyrifo… Endangered Liebm. Exact (wi…
9 198678856 Leichhardtia v… Vulnerable (Guillau… Exact (wi…
10 135836392 Mouriri myrtil… Least Concern (Sw.) Po… NA
11 146459149 Neocussonia um… Least Concern (Sond.) … Exact (wi…
12 170239556 Papuodendron l… Least Concern C.T.White Exact (wi…
13 68117888 Plerandra sp. … Endangered Lowry & … NA
14 133242195 Psammisia scle… Least Concern A.C. Sm. NA
15 580432 Rebutia albipe… Endangered Rausch NA
16 133163169 Senecio canesc… Least Concern (Bonpl.)… NA
17 185585004 Serruria colli… Endangered Salisb. … NA
18 8379911 Sesbania brevi… Least Concern J.B.Gill… NA
19 122659437 Trichilia demi… Critically End… D.Penn. NA
20 1433575 Vicia mollis Least Concern Boiss. &… NA
# ℹ 11 more variables: multiple_matches <lgl>,
# match_similarity <dbl>, match_edit_distance <dbl>,
# wcvp_id <dbl>, wcvp_name <chr>, wcvp_authors <chr>,
# wcvp_rank <chr>, wcvp_status <chr>, wcvp_homotypic <lgl>,
# wcvp_ipni_id <chr>, wcvp_accepted_id <dbl>
wcvp_match_exact(redlist_example, wcvp_names = wcvp_names, "scientificName", id_col = "assessmentId")
# A tibble: 23 × 16
assessmentId scientificName redlistCategory authority match_type
<dbl> <chr> <chr> <chr> <chr>
1 11081542 Antimima quart… Least Concern (Dinter)… NA
2 19395021 Avena hybrida Data Deficient Peterm. Exact (wi…
3 64135503 Citrus garrawa… Least Concern F.M.Bail… NA
4 189601563 Croton campanu… Endangered Caruzo &… Exact (wi…
5 115968141 Cynanchum free… Endangered (N.E.Br.… NA
6 11047751 Echinacanthus … Vulnerable H.S.Lo &… Exact (wi…
7 11001316 Geissanthus pi… Endangered (Lundell… NA
8 126598076 Juglans pyrifo… Endangered Liebm. Exact (wi…
9 126598076 Juglans pyrifo… Endangered Liebm. Exact (wi…
10 198678856 Leichhardtia v… Vulnerable (Guillau… Exact (wi…
# ℹ 13 more rows
# ℹ 11 more variables: multiple_matches <lgl>,
# match_similarity <dbl>, match_edit_distance <dbl>,
# wcvp_id <dbl>, wcvp_name <chr>, wcvp_authors <chr>,
# wcvp_rank <chr>, wcvp_status <chr>, wcvp_homotypic <lgl>,
# wcvp_ipni_id <chr>, wcvp_accepted_id <dbl>
# ℹ Use `print(n = ...)` to see more rows
11. wcvp_match_fuzzy() phonetic_match() edit_match()
在WCVP中模糊匹配。使用语法匹配和编辑距离对 WCVP 中的名称进行模糊匹配。
用法
wcvp_match_fuzzy(names_df, wcvp_names, name_col, progress_bar = TRUE)
phonetic_match(names_df, wcvp_names, name_col)
edit_match(names_df, wcvp_names, name_col)
参数
- names_df:用于匹配的名称数据框。
- wcvp_names:从WCVP 版本7或更新中获得的分类名称数据集。如果为NULL(默认),将从rWCVPdata::wcvp_names加载名称
- name_col:字符变量。names_df中用于匹配类群名称的列
- progress_bar:逻辑变量。展示匹配进度条。默认为TRUE;如果选取了标记语言报告,请更改为FALSE
值
将WCVP中的结果与names_df中的原始数据进行匹配。
详介
wcvp_match_fuzzy函数首先使用语法匹配,种后再基于编辑距离查找最接近匹配结果
语法匹配使用了phonics::metaphoe,最大可以处理20个字符长度。
编辑距离匹配根据Levenshtein相似性查找最接近的匹配,使用RecordLinkage::levenshteinSim计算。
另请参阅
wcvp_match_exact(), wcvp_match_names()
例子
wcvp_match_fuzzy(redlist_example, wcvp_names = wcvp_names, "scientificName")
# A tibble: 23 × 16■■■■■■■■■■■■■■■ 80% | ETA: 4s
assessmentId scientificName redlistCategory authority match_type
<dbl> <chr> <chr> <chr> <chr>
1 19395021 Avena hybrida Data Deficient Peterm. Fuzzy (ph…
2 64135503 Citrus garrawa… Least Concern F.M.Bail… Fuzzy (ph…
3 189601563 Croton campanu… Endangered Caruzo &… Fuzzy (ph…
4 115968141 Cynanchum free… Endangered (N.E.Br.… Fuzzy (ph…
5 11047751 Echinacanthus … Vulnerable H.S.Lo &… Fuzzy (ph…
6 126598076 Juglans pyrifo… Endangered Liebm. Fuzzy (ph…
7 126598076 Juglans pyrifo… Endangered Liebm. Fuzzy (ph…
8 198678856 Leichhardtia v… Vulnerable (Guillau… Fuzzy (ph…
9 135836392 Mouriri myrtil… Least Concern (Sw.) Po… Fuzzy (ph…
10 146459149 Neocussonia um… Least Concern (Sond.) … Fuzzy (ph…
# ℹ 13 more rows
# ℹ 11 more variables: multiple_matches <lgl>,
# match_similarity <dbl>, match_edit_distance <dbl>,
# wcvp_id <dbl>, wcvp_name <chr>, wcvp_authors <chr>,
# wcvp_rank <chr>, wcvp_status <chr>, wcvp_homotypic <lgl>,
# wcvp_ipni_id <chr>, wcvp_accepted_id <dbl>
# ℹ Use `print(n = ...)` to see more rows
phonetic_match(redlist_example, wcvp_names = wcvp_names, "scientificName")
# A tibble: 24 × 16
assessmentId scientificName redlistCategory authority match_type
<dbl> <chr> <chr> <chr> <chr>
1 11081542 Antimima quart… Least Concern (Dinter)… NA
2 19395021 Avena hybrida Data Deficient Peterm. Fuzzy (ph…
3 64135503 Citrus garrawa… Least Concern F.M.Bail… Fuzzy (ph…
4 189601563 Croton campanu… Endangered Caruzo &… Fuzzy (ph…
5 115968141 Cynanchum free… Endangered (N.E.Br.… Fuzzy (ph…
6 11047751 Echinacanthus … Vulnerable H.S.Lo &… Fuzzy (ph…
7 11001316 Geissanthus pi… Endangered (Lundell… NA
8 126598076 Juglans pyrifo… Endangered Liebm. Fuzzy (ph…
9 126598076 Juglans pyrifo… Endangered Liebm. Fuzzy (ph…
10 198678856 Leichhardtia v… Vulnerable (Guillau… Fuzzy (ph…
# ℹ 14 more rows
# ℹ 11 more variables: multiple_matches <lgl>,
# match_similarity <dbl>, match_edit_distance <dbl>,
# wcvp_id <dbl>, wcvp_name <chr>, wcvp_authors <chr>,
# wcvp_rank <chr>, wcvp_status <chr>, wcvp_homotypic <lgl>,
# wcvp_ipni_id <chr>, wcvp_accepted_id <dbl>
# ℹ Use `print(n = ...)` to see more rows
edit_match(redlist_example, wcvp_names = wcvp_names, "scientificName")
# A tibble: 23 × 16■■■■■■■■■■■■■■■■■■■ 95% | ETA: 3s
assessmentId scientificName redlistCategory authority match_type
<dbl> <chr> <chr> <chr> <chr>
1 11081542 Antimima quart… Least Concern (Dinter)… Fuzzy (ed…
2 19395021 Avena hybrida Data Deficient Peterm. Fuzzy (ed…
3 64135503 Citrus garrawa… Least Concern F.M.Bail… Fuzzy (ed…
4 189601563 Croton campanu… Endangered Caruzo &… Fuzzy (ed…
5 115968141 Cynanchum free… Endangered (N.E.Br.… Fuzzy (ed…
6 11047751 Echinacanthus … Vulnerable H.S.Lo &… Fuzzy (ed…
7 11001316 Geissanthus pi… Endangered (Lundell… Fuzzy (ed…
8 126598076 Juglans pyrifo… Endangered Liebm. Fuzzy (ed…
9 126598076 Juglans pyrifo… Endangered Liebm. Fuzzy (ed…
10 198678856 Leichhardtia v… Vulnerable (Guillau… Fuzzy (ed…
# ℹ 13 more rows
# ℹ 11 more variables: multiple_matches <lgl>,
# match_similarity <dbl>, match_edit_distance <dbl>,
# wcvp_id <dbl>, wcvp_name <chr>, wcvp_authors <chr>,
# wcvp_rank <chr>, wcvp_status <chr>, wcvp_homotypic <lgl>,
# wcvp_ipni_id <chr>, wcvp_accepted_id <dbl>
# ℹ Use `print(n = ...)` to see more rows
12. wcvp_match_names()
在WCVP中匹配名称。将名称与 WCVP 匹配,首先使用精确匹配,然后对任何剩余的未匹配名称使用模糊匹配。
用法
wcvp_match_names(
names_df,
wcvp_names = NULL,
name_col = NULL,
id_col = NULL,
author_col = NULL,
join_cols = NULL,
fuzzy = TRUE,
progress_bar = TRUE
)
参数
- names_df:用于匹配的名称数据框。
- wcvp_names:从WCVP 版本7或更新中获得的分类名称数据集。如果为NULL(默认),将从rWCVPdata::wcvp_names加载名称
- name_col:字符变量。names_df中用于匹配类群名称的列
- progress_bar:逻辑变量。展示匹配进度条。默认为TRUE;如果选取了标记语言报告,请更改为FALSE
- author_col:names_df中含有名称命名人、用于辅助匹配的列。设置为NULL让匹配忽略命名人字符串
- id_col:names_df中含有发现记录id的列
- join_cols:字符变量。如果没有提供name_col,通过该向量各部分组建一个类群名称
- fuzzy:逻辑变量;是否应该对无法精确匹配的名称使用模糊匹配。
值
将WCVP中的结果与names_df中的原始数据进行匹配。
详介
一般来说,精确匹配只使用类群名称(name_col),除非提供了作者名(author_col)。
还可以通过join_cols来组建类群名称,但必须要确保它们的组建顺序正确(例如,c(“genus”, “species”, “infra_rank”, “infra”))。
模糊匹配使用语法和编辑距离匹配相结合,可以使用fuzzy=FALSE关闭模糊匹配。
另请参阅
wcvp_match_exact(), wcvp_match_fuzzy()
例子
wcvp_match_names(redlist_example, wcvp_names, name_col = "scientificName", id_col = "assessmentId")
── Matching names to WCVP ──────────────────────────────────────────
ℹ Using the `scientificName` column
! No author information supplied - matching on taxon name only
── Exact matching names ──
✔ Found 12 of names
── Fuzzy matching 8 names ──
✔ Found 7 of 8 names
── Matching complete! ──
✔ Matched 19 of 20 names
ℹ Exact (without author): 12
ℹ Fuzzy (edit distance): 4
ℹ Fuzzy (phonetic): 3
! Names with multiple matches: 3
# A tibble: 23 × 16■■■■■■■■■■■■■■■ 80% | ETA: 4s
assessmentId scientificName redlistCategory authority match_type
<dbl> <chr> <chr> <chr> <chr>
1 11081542 Antimima quart… Least Concern (Dinter)… Fuzzy (ed…
2 19395021 Avena hybrida Data Deficient Peterm. Exact (wi…
3 64135503 Citrus garrawa… Least Concern F.M.Bail… Fuzzy (ph…
4 189601563 Croton campanu… Endangered Caruzo &… Exact (wi…
5 115968141 Cynanchum free… Endangered (N.E.Br.… Fuzzy (ph…
6 11047751 Echinacanthus … Vulnerable H.S.Lo &… Exact (wi…
7 11001316 Geissanthus pi… Endangered (Lundell… Fuzzy (ed…
8 126598076 Juglans pyrifo… Endangered Liebm. Exact (wi…
9 126598076 Juglans pyrifo… Endangered Liebm. Exact (wi…
10 198678856 Leichhardtia v… Vulnerable (Guillau… Exact (wi…
# ℹ 13 more rows
# ℹ 11 more variables: multiple_matches <lgl>,
# match_similarity <dbl>, match_edit_distance <dbl>,
# wcvp_id <dbl>, wcvp_name <chr>, wcvp_authors <chr>,
# wcvp_rank <chr>, wcvp_status <chr>, wcvp_homotypic <lgl>,
# wcvp_ipni_id <chr>, wcvp_accepted_id <dbl>
# ℹ Use `print(n = ...)` to see more rows
13. wcvp_occ_mat()
生成类群和区域的发现记录矩阵
语法
wcvp_occ_mat(
taxon = NULL,
taxon_rank = c("species", "genus", "family", "order", "higher"),
area_codes = NULL,
native = TRUE,
introduced = TRUE,
extinct = TRUE,
location_doubtful = TRUE,
wcvp_names = NULL,
wcvp_distributions = NULL
)
参数
- taxon:字符变量。指包含的类群。默认为NULL(不进行类群筛选,所有类群)
- taxon_rank:字符变量。“species”, “genus”, “family”, “order” or "higher"之一,指定 taxon的分类阶元。除非 taxon为 NULL,否则必须指定
- area_codes:字符变量。单个或多个WGSRPD3级区域代码。默认为NULL(全球范围)
- synonyms:逻辑变量。在清单中包含异名。默认为TRUE
- render_report:逻辑变量。将清单呈现为标记语言报告。默认为FALSE
- native:逻辑变量。包含未标记为 introduced,extinct或doubful的物种发现记录
- introduced:逻辑变量。包含标记为引入的物种发现记录。默认为TRUE
- extinct:逻辑变量。包含标记为灭绝的物种发现记录。默认为TRUE
- location_doubtful:逻辑变量。包含标记为产地存疑的物种发现记录。默认为TRUE
- wcvp_names:从WCVP 版本7或更新中获得的分类名称数据集。如果为NULL(默认),将从rWCVPdata::wcvp_names加载名称
- wcvp_distributions:从WCVP 版本7或更新中获得的分布区数据集。如果为NULL(默认),将从rWCVPdata::wcvp_distributions加载名称
值
含有taxon_name和plant_name_id的一个数据集
详介
请参阅此处的示例,了解如何将此输出格式化以供发表。
例子
wcvp_occ_mat(taxon = "Poa", taxon_rank = "genus",
area = c("TAS","VIC","NSW"),
introduced = FALSE)
# A tibble: 37 × 5
plant_name_id taxon_name NSW TAS VIC
<dbl> <chr> <dbl> <dbl> <dbl>
1 435044 Poa affinis 1 0 1
2 469193 Poa amplexicaulis 0 0 1
3 435565 Poa cheelii 1 0 0
4 435598 Poa clelandii 0 1 1
5 435599 Poa clivicola 1 1 1
6 435658 Poa costiniana 1 1 1
7 435661 Poa crassicaudex 0 0 1
8 435797 Poa drummondiana 0 0 1
9 435841 Poa ensiformis 1 0 1
10 435878 Poa fawcettiae 1 1 1
# ℹ 27 more rows
# ℹ Use `print(n = ...)` to see more rows
14. wcvp_reformat()
WCVP本地版本的重新格式化
用法
wcvp_reformat(wcvp_local, version = NULL)
参数
- wcvp_local:数据集。WCVP的本地副本。
- version:9或”v9“。
值
返回相同结构的一个数据集
详介
请注意,并非所有原始变量在重新格式化期间都会保留。例如,publication 在 v9 中是单个变量,但在数据包中拆分为多个变量。因此不可能简单地重命名这个变量。数据包中存在但 v9 中不存在的变量用 NA 填充。
15. wcvp_summary()
从WCVP中生成一个概要表
用法
wcvp_summary(
taxon = NULL,
taxon_rank = c("species", "genus", "family", "order", "higher"),
area_codes = NULL,
grouping_var = c("area_code_l3", "genus", "family", "order", "higher"),
hybrids = FALSE,
wcvp_names = NULL,
wcvp_distributions = NULL
)
参数
- taxon:字符变量。指包含的类群。默认为NULL(不进行类群筛选,所有类群)
- taxon_rank:字符变量。“species”, “genus”, “family”, “order” or "higher"之一,指定 taxon的分类阶元。除非 taxon为 NULL,否则必须指定
- area_codes:字符变量。单个或多个WGSRPD3级区域代码。默认为NULL(全球范围)
- grouping_var:字符变量;“area_code_l3”, “genus”, “family”,“order” or "higher"之一,指定摘要如何排序。默认为area_code_l3
- hybrids:逻辑变量。是否统计杂交种。默认为FALSE
-
- wcvp_names:从WCVP 版本7或更新中获得的分类名称数据集。如果为NULL(默认),将从rWCVPdata::wcvp_names加载名称
- wcvp_distributions:从WCVP 版本7或更新中获得的分布区数据集。如果为NULL(默认),将从rWCVPdata::wcvp_distributions加载名称
值
筛选后的数据集,或一个gt表格
详介
分类阶元higher的可用值有 Angiosperms, Gymnosperms, Ferns and Lycophytes 。请注意,分组变量(如果是分类学的)应低于 taxon 和 taxon_rank 的级别,以生成有意义的摘要(即,按属、科或更高分类对属进行分组没有意义)。此外,如果分组变量是分类变量,则物种出现在整个输入区域。这意味着如果一个物种是任何输入区域的本地物种(即使它是在其他地区引入或灭绝的),它也被视为“本地”。同样,引入的事件优先于灭绝的事件。请注意,在此类汇总表中,“地方性”表示输入区域特有,不一定是输入区域内的单个 WGSRPD 第 3 级区域。
例子
ferns = wcvp_summary("Ferns", "higher", get_wgsrpd3_codes("New Zealand"), grouping_var = "family")
wcvp_summary_gt(ferns)
ℹ Matches to input geography found at Country (Gallagher) and Region (Level 2)
ℹ Aggregating occurrence types across input area ("New Zealand") - see `?wcvp_summary()` for details.
Matching ■■■■■■■■■■■■■■■■■■■■■■■■■ 80% | ETA: 4s
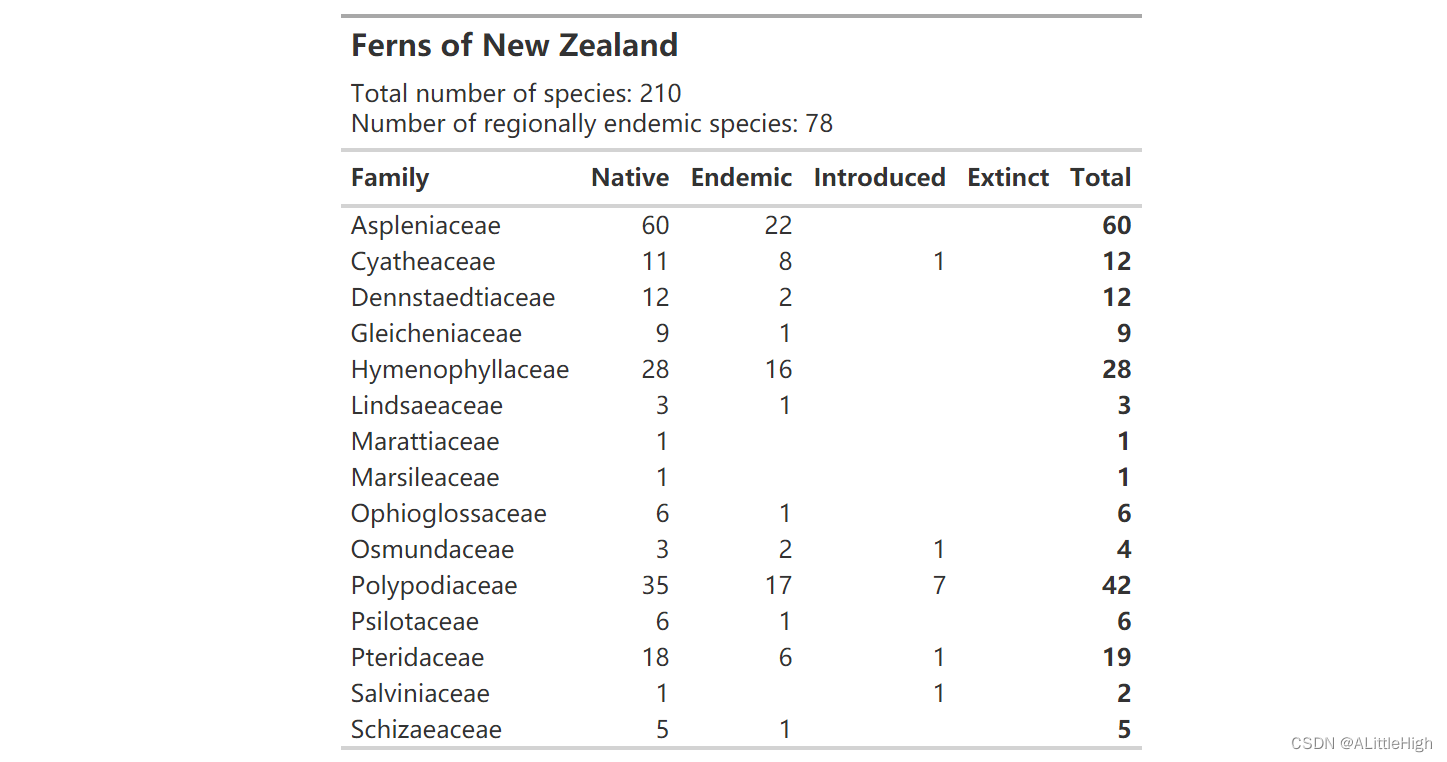
16. wcvp_summary_gt()
从wcvp_summary结果中呈现摘要表
用法
wcvp_summary_gt(x)
参数
- x:列表
值
gt表格
例子
见15. 例子
17. wgsrpd3
生物多样性信息标准 (TDWG) 记录植物分布的世界地理计划 (WGSRPD)。WGSRPD 3级的空间数据,用于绘制地图
用法
wgsrpd3
格式
一个包含20行和4个变量的“sf”对象:
- LEVEL3_NAM:区域名称
- LEVEL3_COD:区域代码
- LEVEL2_COD:2级代码
- LEVEL1_COD:1级(大陆)
geometry:sf几何
fillcol:用于映射
资源
https://github.com/tdwg/wgsrpd/tree/master/level3添加链接描述
18. wgsrpd_mapping
将 WGSRPD 地理映射到其他级别的数据。一个数据集,包含每个3级地区的地区(3级)、#'地区(2级)、大陆(1级)、国家(政治)和半球类别。
用法
wgsrpd_mapping
格式
一个包含370行和7个变量的数据集:
- HEMISPHERE:北、南或赤道(跨赤道)
- LEVEL1_COD:大陆代码
- LEVEL1_NAM:大陆
- LEVEL2_COD:区域代码
- LEVEL2_NAM:区域
- COUNTRY:国家(行政)
- LEVEL3_COD:地区代码
- LEVEL3_NAM:地区
资源
https://github.com/tdwg/wgsrpd