扫码关注下方公粽号,回复推文合集,获取400页单细胞学习资源!

本文共计1887字,阅读大约需要6分钟,目录如下:
-
SingleR基本介绍
-
SingleR包安装
-
SingleR包使用
-
- 1.使用已有的参考数据集进行细胞定义
- 2.使用自定义数据集进行细胞定义
-
小结
-
获取代码
-
代码参考
-
往期单细胞系统教程
SingleR基本介绍
SingleR最早发表在2019年Nature Immunology杂志的一篇文章上,文章题目为"Reference-based analysis of lung single-cell sequencing reveals a transitional profibrotic macrophage"。截止至2023年3月10日,引用量已经达到了947。SingleR的基本原理是利用已知类型细胞的基因表达谱和单个细胞的基因表达谱的相关性进行细胞类型鉴定。
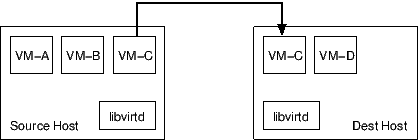
SingleR的具体流程是(图1):

图1:SingleR工作示意图(来源:Nat Immunol. 2019 Feb;20(2):163-172.)
1.计算参考数据集中各个细胞类型基因表达谱的可变基因,对于每一个需要鉴定的细胞,计算单个细胞与参考数据集中各个细胞类型基因在可变基因上的Spearman相关性;
2.根据参考基因集的命名注释聚合每个细胞类型的多重相关系数,每种细胞类型的评分默认选择相关系数值的80%,此时可以筛选得到每个细胞最可能的细胞定义类型;
3.计算来自参考数据集中最可能的细胞类型基因表达谱的可变基因,并计算单个细胞与最可能细胞类型在可变基因上的Spearman相关性,去除最低值的细胞(或是比最高值低于0.05的细胞),重复此步骤,直到只保留两种细胞类型。最后一次运行后,相关系数值最高的细胞类型则作为需要鉴定的细胞的定义。
此外,SingleR还提供了网页版工具(图2)用于在线注释细胞类型(https://comphealth.ucsf.edu/app/singler)。

图2:SingleR网页版
SingleR包安装
使用devtools包或者BiocManager进行安装。
devtools::install_github('dviraran/SingleR')
#BiocManager::install("SingleR")
SingleR包使用
1.使用已有的参考数据集进行细胞定义
celldex(http://bioconductor.org/packages/release/data/experiment/vignettes/celldex/inst/doc/userguide.html)提供了来自bulk RNA-seq或者是芯片测序的7个常用的参考数据库:
①General-purpose references:
Human primary cell atlas (HPCA);
Blueprint/ENCODE;
Mouse RNA-seq。
②Immune references:
Immunological Genome Project (ImmGen);
Database of Immune Cell Expression/eQTLs/Epigenomics (DICE);
Novershtern hematopoietic data;
Monaco immune data。
可用HumanPrimaryCellAtlasData()函数从Human Primary Cell Atlas中获取参考数据,建议将参考数据本地保存,便于每次分析时加载。
library(celldex)
hpca.se <- HumanPrimaryCellAtlasData()
hpca.se
save(hpca.se,file="hpca.se.RData")
加载SingleR和待注释单细胞数据集(待注释数据集来自10X官网的PBMC数据,已经按照官方流程整理成pbmc3k.rds),保证两个数据集的基因相同。由于参考集里面是logcounts矩阵,后面对于单细胞数据集也要做类似的处理。
library(SingleR)
library(scater)
library(SummarizedExperiment)
library(cowplot)
test.seu<-readRDS('pbmc3k.rds')
test.count=as.data.frame(test.seu[["RNA"]]@counts)
load(file="hpca.se.RData")
common_hpca <- intersect(rownames(test.count), rownames(hpca.se))
hpca.se <- hpca.se[common_hpca,]
test.count_forhpca <- test.count[common_hpca,]
test.count_forhpca.se <- SummarizedExperiment(assays=list(counts=test.count_forhpca))
test.count_forhpca.se <- logNormCounts(test.count_forhpca.se)
定义主要的细胞类型,这里使用的是label.main参数进行细胞大类注释。
pred.main.hpca <- SingleR(test = test.count_forhpca.se, ref = hpca.se, labels = hpca.se$label.main)
result_main_hpca <- as.data.frame(pred.main.hpca$labels)
result_main_hpca$CB <- rownames(pred.main.hpca)
colnames(result_main_hpca) <- c('HPCA_Main', 'CB')
head(result_main_hpca)

图3:result_main_hpca数据结构
整合到meta.data中,利用UMAP展示结果(图4)。
test.seu@meta.data$CB=rownames(test.seu@meta.data)
test.seu@meta.data=inner_join(test.seu@meta.data,result_main_hpca,by="CB")
rownames(test.seu@meta.data)=test.seu@meta.data$CB
DimPlot(test.seu, reduction = "umap", group.by = "HPCA_Main",label = TRUE,repel = TRUE,ncol=1)+
DimPlot(test.seu, reduction = "umap", group.by = "ident",label = TRUE,repel = TRUE,ncol=1)&
theme(axis.line = element_blank(), axis.ticks= element_blank(),axis.text = element_blank())

图4:SingleR鉴定结果
SingleR细胞注释结果与典型的marker基因表达结果对比,可见在单核细胞(LYZ、CD14)、B细胞(MS4A1)、T细胞(CD3E、CD8A)、NK细胞(GNLY)、血小板(PPBP)大群上注释比较准确(图5)。
FeaturePlot(test.seu, features = c("MS4A1", "GNLY", "CD3E", "CD14", "FCER1A", "FCGR3A", "LYZ", "PPBP", "CD8A"))

图5:细胞典型标记物的表达情况
我们也可以将参数修改为label.fine进行再次分析,与典型标记物结果对比可见,比如表达FCER1A的树突状细胞并没有区分出来,但是也根据FCGR3A的表达区分出来两群单核细胞(图6)。
pred.detail.hpca <- SingleR(test = test.count_forhpca.se, ref = hpca.se, labels = hpca.se$label.fine)

图6:SingleR鉴定结果2
2.使用自定义数据集进行细胞定义
使用自己定义/找到的单细胞数据集做注释,演示用到的数据集来自10X官网的PBMC数据。用PBMC5k数据集作为参考(已根据典型细胞标记物定义好细胞类型)来注释PBMC3k的细胞。两套数据集,已经跑完Seurat标准流程并整理成rds文件:pbmc3k.rds和pbmc5k.rds。首先进行参考数据集的构建。
library(Seurat)
library(tidyverse)
library(SummarizedExperiment)
library(scuttle)
pbmc5k=readRDS("pbmc5k.rds")
pbmc5k_count=pbmc5k[["RNA"]]@counts
pbmc5k@meta.data$Index =rownames(pbmc5k@meta.data) #给metadata增加一列Index,代表每个细胞名字
pdata=pbmc5k@meta.data[,c("Index","celltype")]
rownames(pdata)=pdata$Index
pdata$Index=NULL
colnames(pdata)="ref_label"
pbmc5k_SE <- SummarizedExperiment(assays=list(counts=pbmc5k_count),colData = pdata)
pbmc5k_SE <- logNormCounts(pbmc5k_SE)
saveRDS(pbmc5k_SE,"pbmc5k_SE.ref2.rds")
数据整理完毕后就可以导入待注释数据集和参考数据集进行细胞注释(图7),和前面用已有的参考数据集鉴定的结果是还是有点差异的。
library(tidyverse)
library(Seurat)
library(SingleR)
library(scuttle)
library(SummarizedExperiment)
pbmc5k_SE=readRDS("pbmc5k_SE.ref2.rds")
pbmc3k=readRDS("pbmc3k.rds")
pbmc3k_count=pbmc3k[["RNA"]]@counts
common_gene <- intersect(rownames(pbmc3k_count), rownames(pbmc5k_SE))
pbmc5k_SE <- pbmc5k_SE[common_gene,]
pbmc3k_count <- pbmc3k_count[common_gene,]
pbmc3k_SE <- SummarizedExperiment(assays=list(counts=pbmc3k_count))
pbmc3k_SE <- logNormCounts(pbmc3k_SE)
singleR_res <- SingleR(test = pbmc3k_SE, ref = pbmc5k_SE, labels = pbmc5k_SE$ref_label)
anno_df <- as.data.frame(singleR_res$labels)
anno_df$Index <- rownames(singleR_res)
colnames(anno_df)[1] <- 'ref_label_from_pbmc5k'
pbmc3k@meta.data=pbmc3k@meta.data%>%inner_join(anno_df,by="Index")
rownames(pbmc3k@meta.data)=pbmc3k@meta.data$Index
DimPlot(pbmc3k, reduction = "umap", group.by = "HPCA_Main",label = TRUE,repel = TRUE,ncol=1)+
DimPlot(pbmc3k, reduction = "umap", group.by = "ref_label_from_pbmc5k",label = TRUE,repel = TRUE,ncol=1)&
theme(axis.line = element_blank(), axis.ticks= element_blank(),axis.text = element_blank())

图7:自定义数据集鉴定结果
小结
使用SingleR自带的数据集在进行细胞的大类定义时还是比较准确的,但在更细致的亚群区分中往往表现不佳,后续我们也将介绍其他一些在单细胞转录组测序中常用细胞注释软件。
获取代码
代码和测试数据请关注公粽号获取

代码参考
GitHub - dviraran/SingleR: SingleR: Single-cell RNA-seq cell types Recognition (legacy version)
https://satijalab.org/seurat/articles/pbmc3k_tutorial.html
往期单细胞系统教程
单细胞分析实录(1): 认识Cell Hashing
单细胞分析实录(2): 使用Cell Ranger得到表达矩阵
单细胞分析实录(3): Cell Hashing数据拆分
单细胞分析实录(4): doublet检测
单细胞分析实录(5): Seurat标准流程
单细胞分析实录(6): 去除批次效应/整合数据
单细胞分析实录(7): 差异表达分析/细胞类型注释
推荐几个细胞注释网站
如何批量查询marker基因(对应的蛋白)会不会在膜上表达?
单细胞分析实录(8): 展示marker基因的4种图形(一)
单细胞分析实录(9): 展示marker基因的4种图形(二)
单细胞分析实录(10): 消除细胞周期的影响
单细胞分析实录(11): inferCNV的基本用法
单细胞分析实录(12): 如何推断肿瘤细胞
单细胞分析实录(13): inferCNV结合UPhyloplot2分析肿瘤进化
单细胞分析实录(14): 细胞类型注释的另一种思路 — CellID
单细胞分析实录(15): 基于monocle2的拟时序分析
单细胞分析实录(16): 非负矩阵分解(NMF)检测细胞异质性
单细胞分析实录(17): 非负矩阵分解(NMF)代码演示
单细胞分析实录(18): 基于CellPhoneDB的细胞通讯分析及可视化 (上篇)
单细胞分析实录(19): 基于CellPhoneDB的细胞通讯分析及可视化 (下篇)
一个对接CellPhoneDB的R包
【代码更新】单细胞分析实录(20): 将多个样本的CNV定位到染色体臂,并画热图
【代码更新】单细胞分析实录(21): 非负矩阵分解(NMF)的R代码实现,只需两步,啥图都有