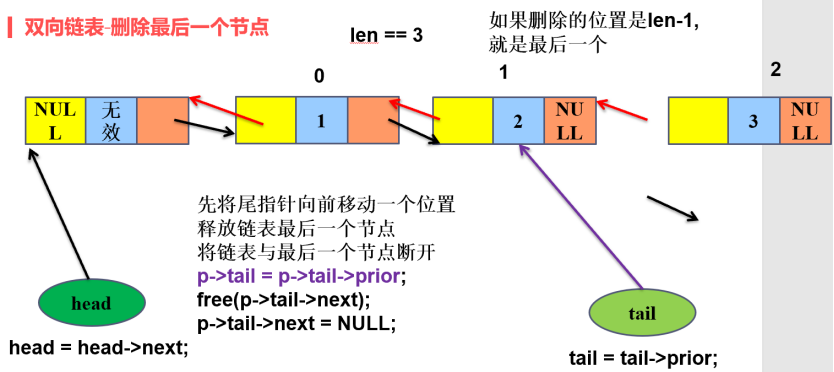
一、比赛地址和背景
1.1 比赛地址
比赛地址
1.2 数据说明及任务
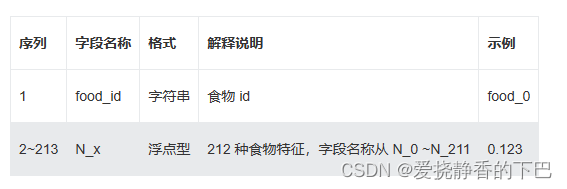
本次算法赛将提供超过 23.5W 的食物、疾病对应关系及其量化得分,其中食物特征超过 200 个,疾病特征由 3 种不同的方式抽取,累积超过 4000 个特征信息。初赛为 0、1 二分类预测,提供食物、疾病特征,与食物疾病的关系标签。
疾病特征

食物特征
1.2.1 初赛任务
本赛道将提供脱敏后的食物与疾病特征,参赛团队根据主办方提供数据,在高度稀疏数据的场景中,进一步挖掘、融合特征并设计模型,以预测食物与疾病的关系。初赛阶段为二分类问题,分类标签分别为 0(无关)、1(存在正面或负面的影响)。

1.2.2 复赛任务
在初赛的基础上,增加对食物与疾病相关性评级维度的评估。
复赛阶段同时评估 0、1 二分类与相关性评级,在原训练集中增加食物疾病相关性评级的标签数据。

其实还是二分类,不过评价方面考虑了相关性的排序
三、总体思路分享
初赛做的比较简单,基本就是在baseline基础上调了一下参数就混进了复赛
复赛多少做了一些工作,下面是主要内容分享
3.1 总体流程
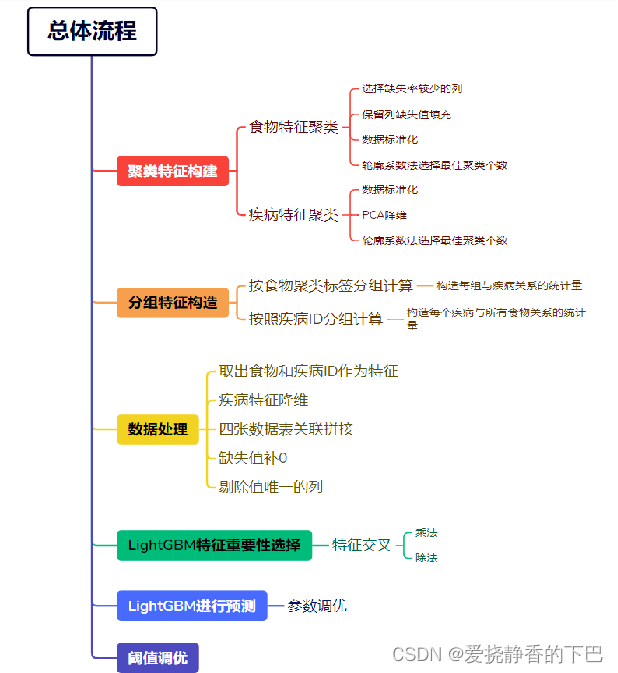
整个流程如图所示
主要采用了高斯聚类、PCA、特征分组衍生、LightGBM等方法
虽然树模型可以处理缺失值,但是这里发现补0可以提高一个多点,由于是匿名特征,所以这里也没办法解释。
另外线上线下差距极大,比较容易过拟合。
3.2 高斯聚类
高斯聚类的思路和流程如下:
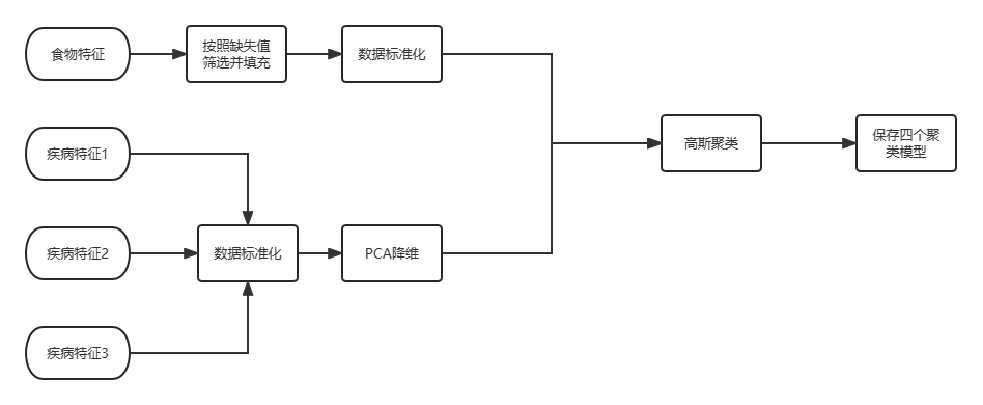
数据特点:所有特征经过脱敏处理。食物特征缺失率较大,三个疾病特征维度高,而且疾病数量各不相同,总体情况较为复杂。
食物的高斯聚类:由于测试集食物是全新的数据,在训练集中没有出现,所以我们想通过聚类算法来打标签。这样就获得了食物的类别标签。也方便进行下一步的特征挖掘。具体方法是找出缺失率较少的列(<10%),然后将这些列的缺失值进行填充(-1,因为部分特征值有0,容易混淆。)
疾病的高斯聚类:虽然疾病特征数据是固定的,但是我们也想要通过聚类算法来找出疾病可以分为几个类别,来获得更多的疾病类别信息。
3.3 特征挖掘
总体思路如下:

分组统计量特征
计算每个食物组在训练集中与疾病相关性的统计量:均值、标准差、偏度、峰度。
计算每个疾病ID在训练集中与食物相关性的统计量:均值、方差、偏度、峰度。
特征交叉
按照LGBM的特征重要性排序,将重要性靠前的特征进行特征交叉,由于复赛进行交叉过拟合严重,所以只选取前5的特征(食物特征)进行乘法和除法的交叉。
3.4 尝试过的其他方法
聚类方法:K均值聚类、层次聚类等,效果最优的是高斯聚类。
降维方法:稀疏PCA效果略优,但是资源消耗较大,其他方法(比如tsvd)替换后效果略差。总体来说差距不大。
分组特征统计量:最大值、最小值、极差、中位数等。
其他分组:尝试将疾病聚类后类别进行分组计算,效果不佳.
特征交叉:初赛将food_id作为交叉项效果较好,但是放在复赛线上线下差距较大。
特征重要性:尝试删除一些特征重要性较低的特征,效果不佳。
模型选择:XGB、Catboost等,但是经过实验,初始效果较差,参数略微调整后没有提升。
模型融合:由于其他模型效果相对较差、模型融合(比如stacking)效果不佳,且模型比较大。
3.5 未尝试的想法
食物与疾病的相关性:
食物和疾病由于不属于一个种类,特征提取方式也有所不同,其中数据间也会存在差异性。
经过多表拼接之后,其实就是将食物疾病特征融合,最后得到一个得分,也就是相关性。
在本赛题中nn模型大多使用的也是先拼接融合,在进行计算,线上线下差距很大。可能是由于模型隐层提供更多的交叉特征信息,导致了过拟合严重。
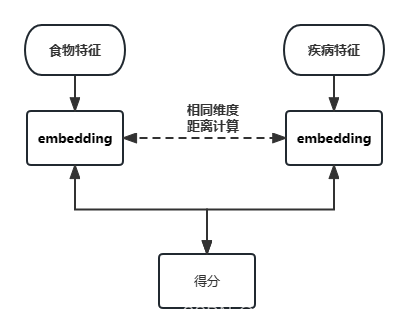
所以双塔模型其实可能会有更好的效果。
双塔模型通过施加以下约束可能会达到更优效果:
1.消除食物和疾病特征间的差异性,这部分差异主要是由特征提取方式不同导致的。L2范数最小化特征矩阵距离,对抗学习等等。
2.在公共特征空间放缩特征距离,有相关性的特征距离缩小,无相关性的特征距离放大
3.其他约束。
四、代码分享
和鲸社区:点击跳转
github:点击跳转
如果觉得有用,可以给个fork和点赞