paleobioDB详解(paleobiology database)
- PBDB
- 初步认识paleobioDB
- 一个简单的例子
- 所有函数详解
- 1. pbdb_collection
- 描述
- 用法
- 参数
- 细节
- 值
- 例子
- 2. pbdb_collections
- 描述
- 用法
- 参数
- 值
- 例子
- 3. pbdb_collections_geo
- 描述
- 用法
- 参数
- 值
- 例子
- 4. pbdb_interval
- 描述
- 用法
- 参数
- 值
- 例子
- 5. pbdb_intervals
- 描述
- 用法
- 参数
- 值
- 例子
- 6. pbdb_map
- 描述
- 用法
- 参数
- 细节
- 值
- 例子
- 7. pbdb_map_occur
- 描述
- 用法
- 参数
- 细节
- 值
- 例子
- 8. pbdb_map_richness
- 描述
- 用法
- 参数
- 细节
- 值
- 例子
- 9. pbdb_occurrence
- 描述
- 用法
- 参数
- 细节
- 值
- 例子
- 10. pbdb_occurrences
- 描述
- 用法
- 参数
- 细节
- 值
- 例子
- 11. pbdb_orig_ext
- 描述
- 用法
- 参数
- 值
- 例子
- 12. pbdb_reference
- 描述
- 用法
- 参数
- 值
- 例子
- 13. pbdb_references
- 描述
- 用法
- 参数
- 值
- 例子
- 14. pbdb_ref_collections
- 描述
- 用法
- 参数
- 值
- 例子
- 15. pbdb_ref_occurrences
- 描述
- 用法
- 参数
- 值
- 例子
- 16. pbdb_ref_taxa
- 描述
- 用法
- 参数
- 值
- 例子
- 17. pbdb_richness
- 描述
- 用法
- 参数
- 值
- 例子
- 18. pbdb_scale
- 描述
- 用法
- 参数
- 值
- 例子
- 19. pbdb_scales
- 描述
- 用法
- 参数
- 值
- 例子
- 20. pbdb_strata
- 描述
- 用法
- 参数
- 值
- 例子
- 21. pbdb_strata_auto
- 描述
- 用法
- 参数
- 值
- 例子
- 22. pbdb_subtaxa
- 描述
- 用法
- 参数
- 值
- 例子
- 23. pbdb_taxa
- 描述
- 用法
- 参数
- 值
- 例子
- 24. pbdb_taxa_auto
- 描述
- 用法
- 参数
- 值
- 例子
- 25. pbdb_taxon
- 描述
- 用法
- 参数
- 值
- 例子
- 26. pbdb_temporal_resolution
- 描述
- 用法
- 参数
- 值
- 例子
- 27. pbdb_temp_range
- 描述
- 用法
- 参数
- 值
- 例子
PBDB
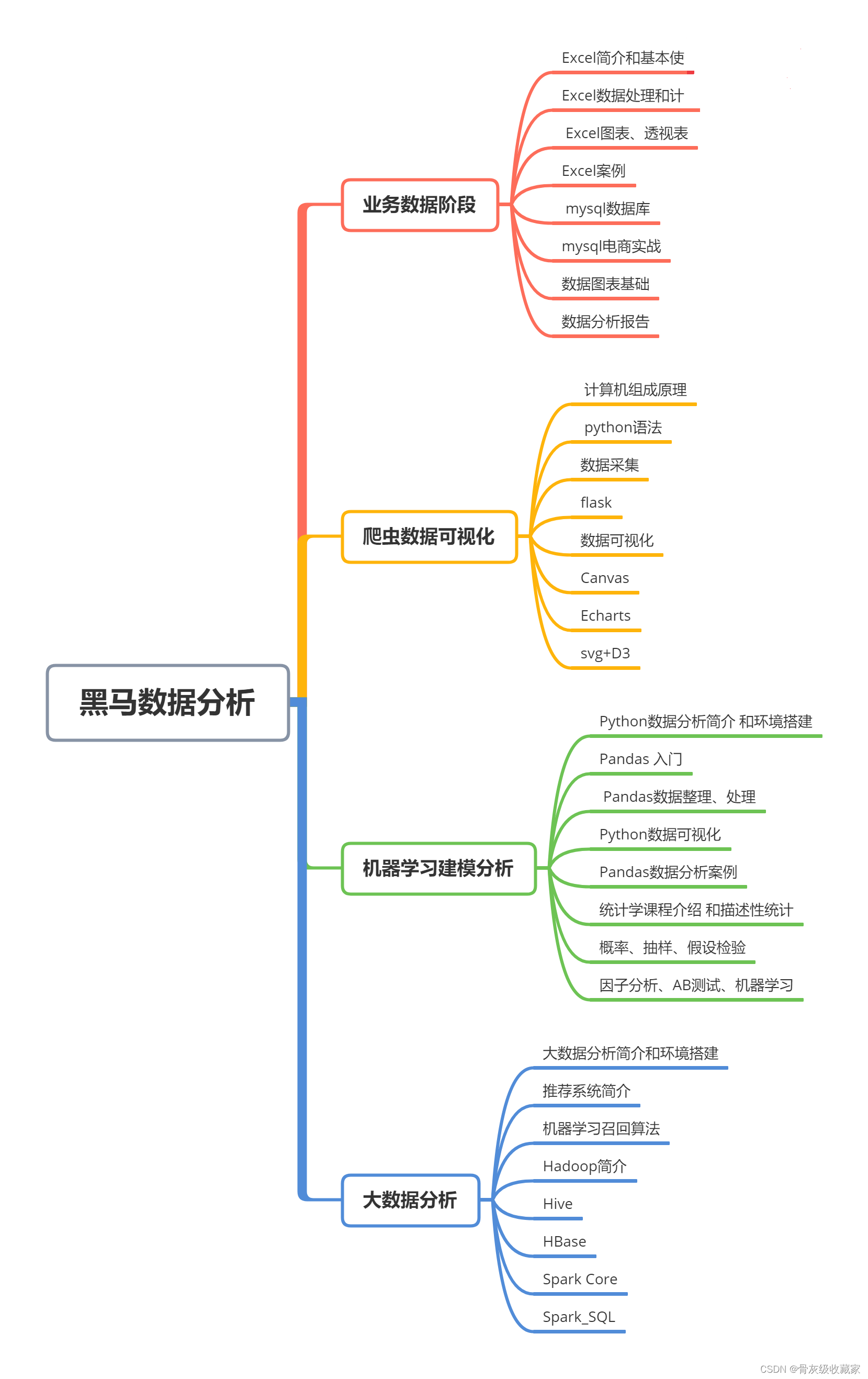
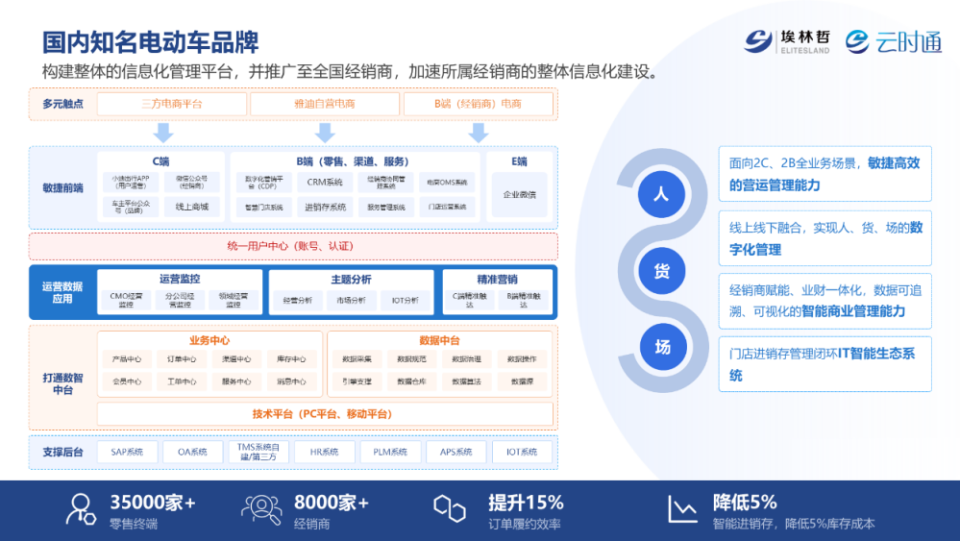
古生物数据库是全球性的古生物学数据库,在古生物学研究中有着举足轻重的地位,为该领域的研究发挥了巨大作用。该数据库提供了优质的DPI以供调用,前人据此利用R语言编写了paleobioDB库。paleobioDB不仅包含了PBDB提供的19个搜索API,还额外提供了8个用于可视化和分析的功能函数。由于该库作者并没有提供官方的介绍文档,所以下面我将通过实践该库进行细致讲解,并在力所能及的范围内,用python编写用于PBDB的库,并进一步写出可视化的操作界面,以供小同行使用。
初步认识paleobioDB
paleobioDB的作者初衷在于:更便捷、更有效地从PBDB获取数据,并进一步可视化和下载目标数据;从而在R语言中更有效对数据进行处理、分析。
paleobioDB的功能函数可分为两组。首先是,对PBDB API进行封装,提供常用且高效的功能。其次是,在以上功能的基础上,从地理、时空和分类三个维度对化石记录进行挖掘分析并可视化。
一个简单的例子
这里有一个不愉快的小插曲,如果你和我遇到了同样的问题,请查看此处解决报错:
Error in function (type, msg, asError = TRUE) :
schannel: CertGetCertificateChain trust error CERT_TRUST_IS_NOT_TIME_VALID
解决了上面的小坑后,让我们通过下面的示例来初步了解并使用paleobioDB(例子中功能函数在后面详解)
library(paleobioDB)
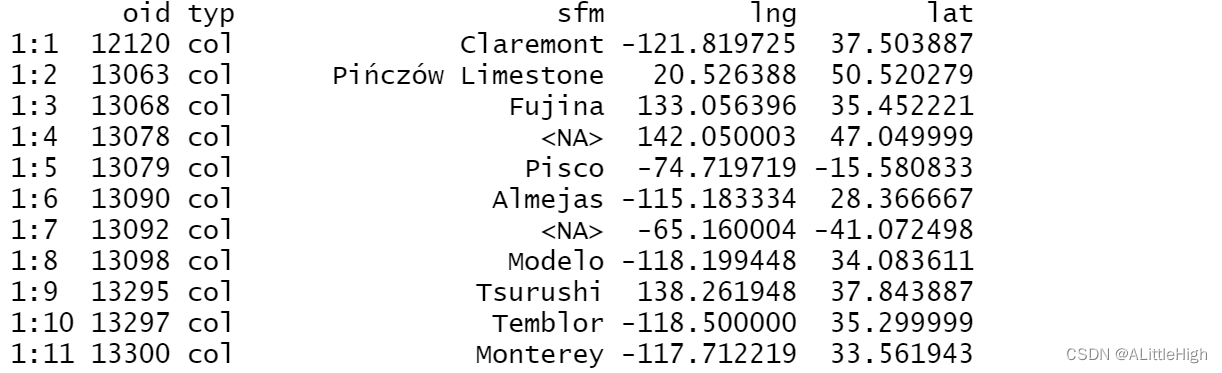
canidae = pbdb_occurrences (limit="all", base_name="canidae",
interval="Quaternary", show=c("coords", "phylo", "ident"))
稍作等待后,它会返回一个数据:

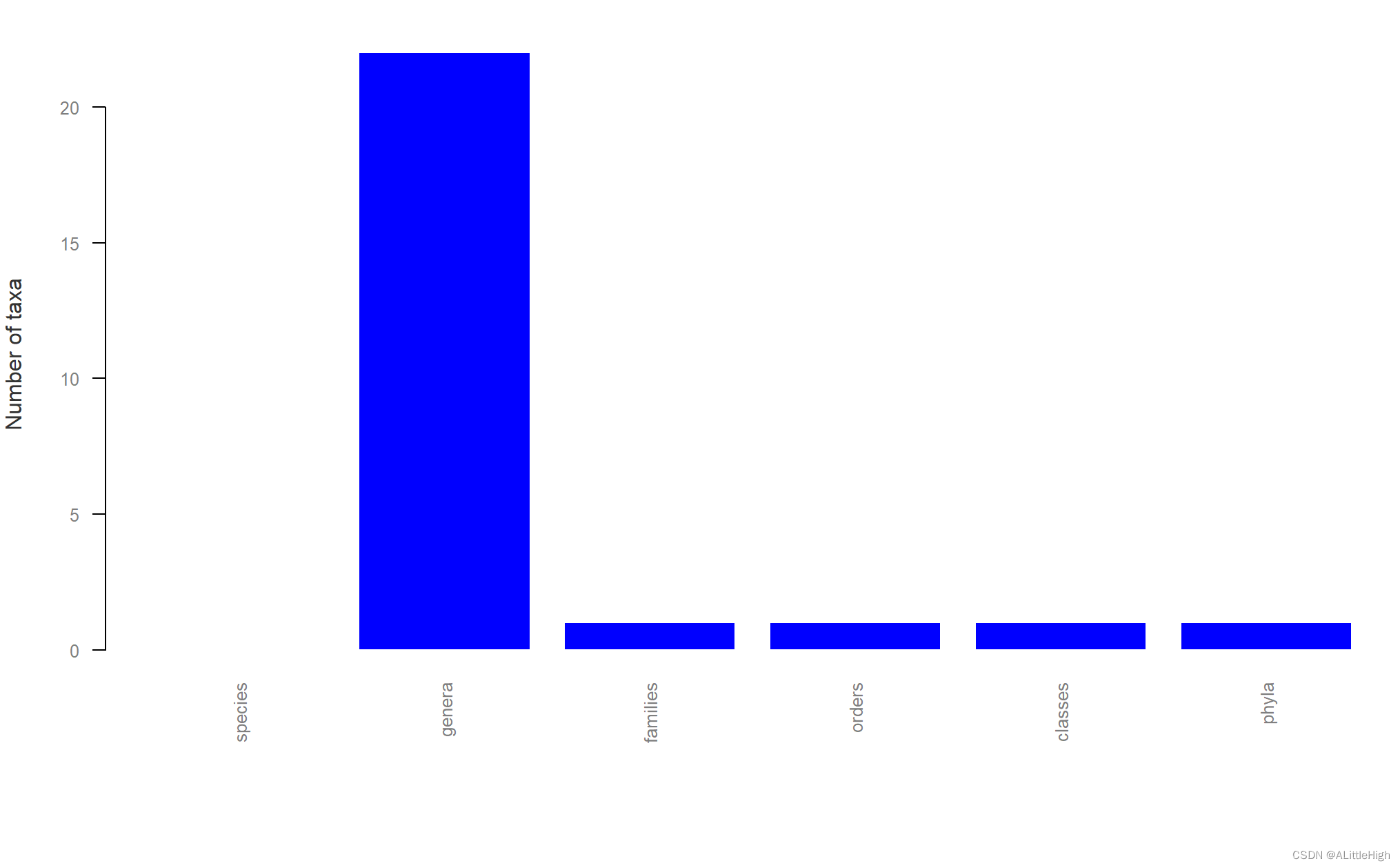
# to explore the number of subtaxa 显示各级分类单元的数量
pbdb_subtaxa(canidae)
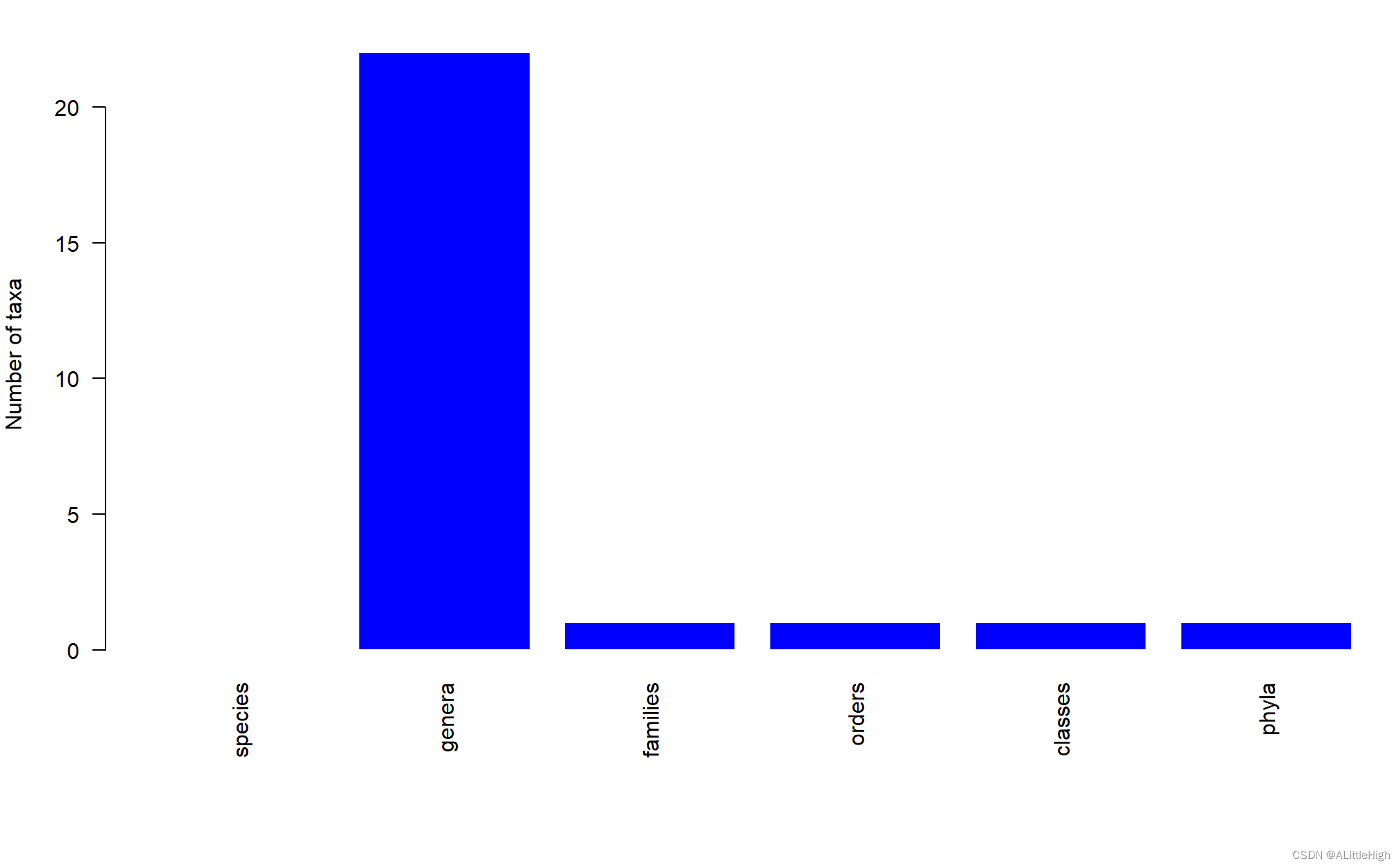
pbdb_subtaxa()既在控制台输出结果,还会将结果可视化:
标题行从左到右依次为:种、属、科、目、纲、门。

# to explore the temporal resolution of the fossil records
# 分析化石记录的时间分布
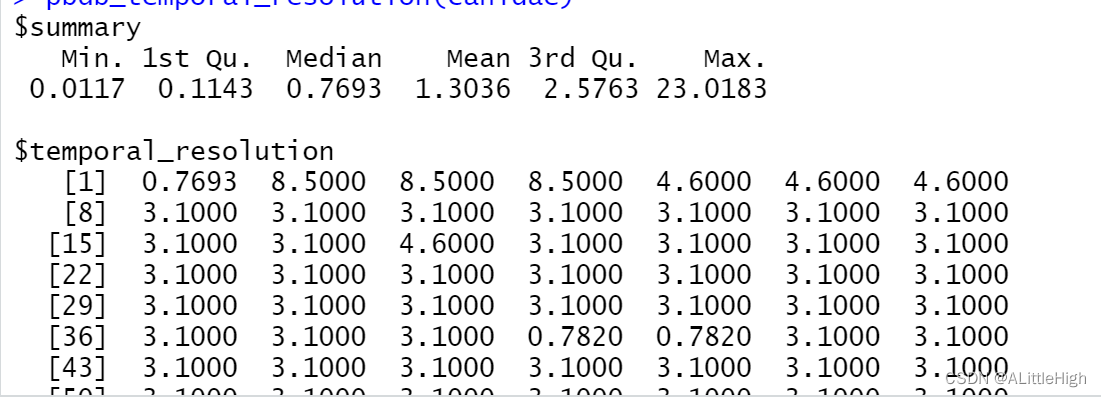
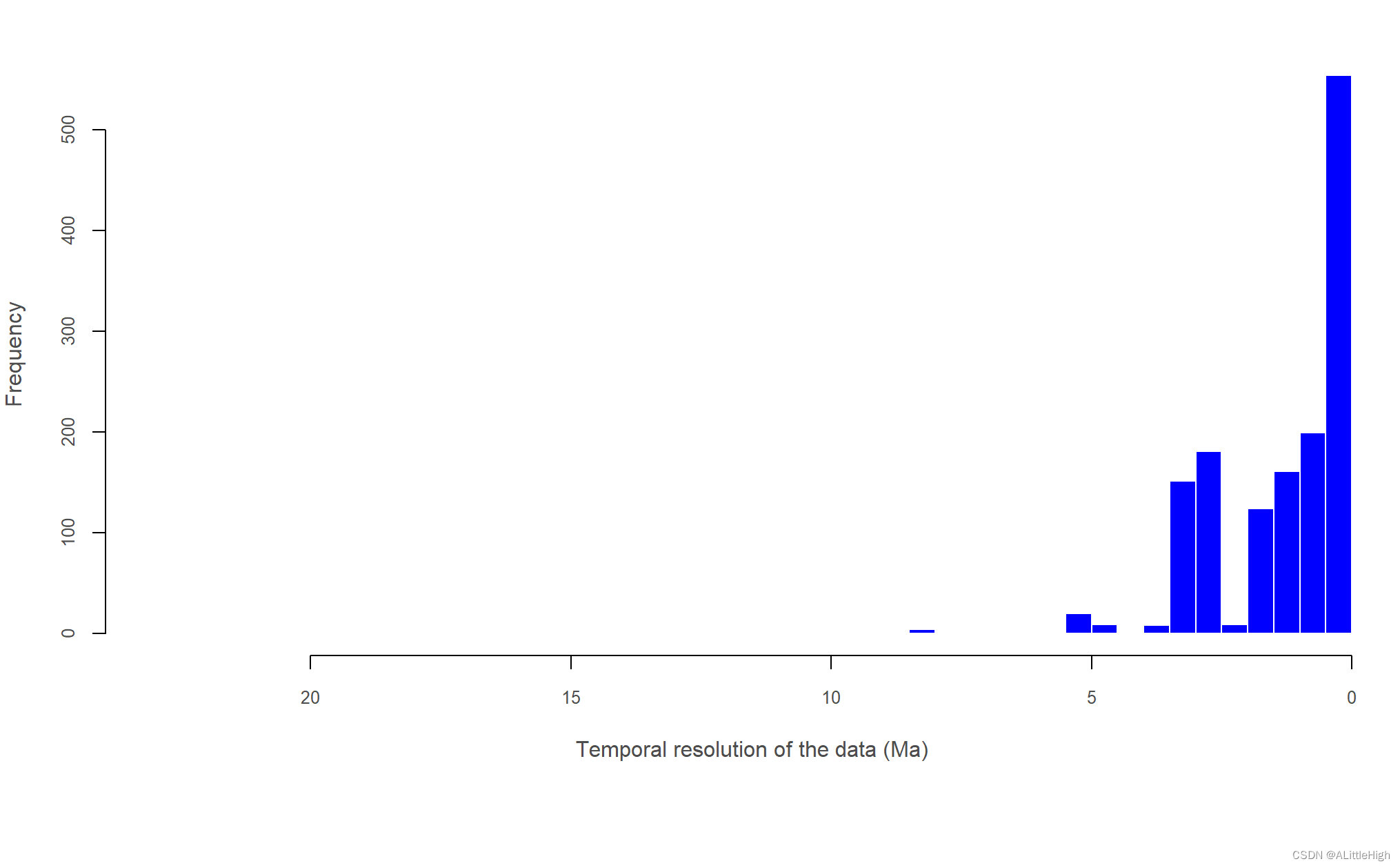
pbdb_temporal_resolution(canidae)
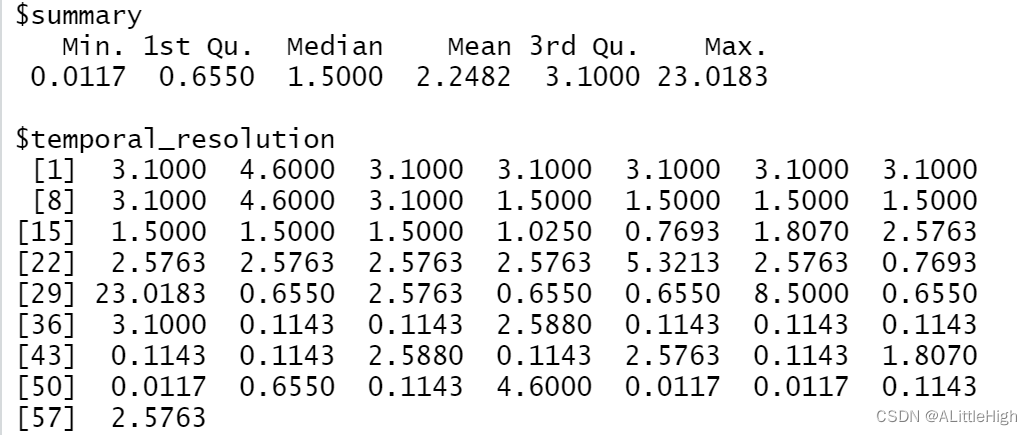
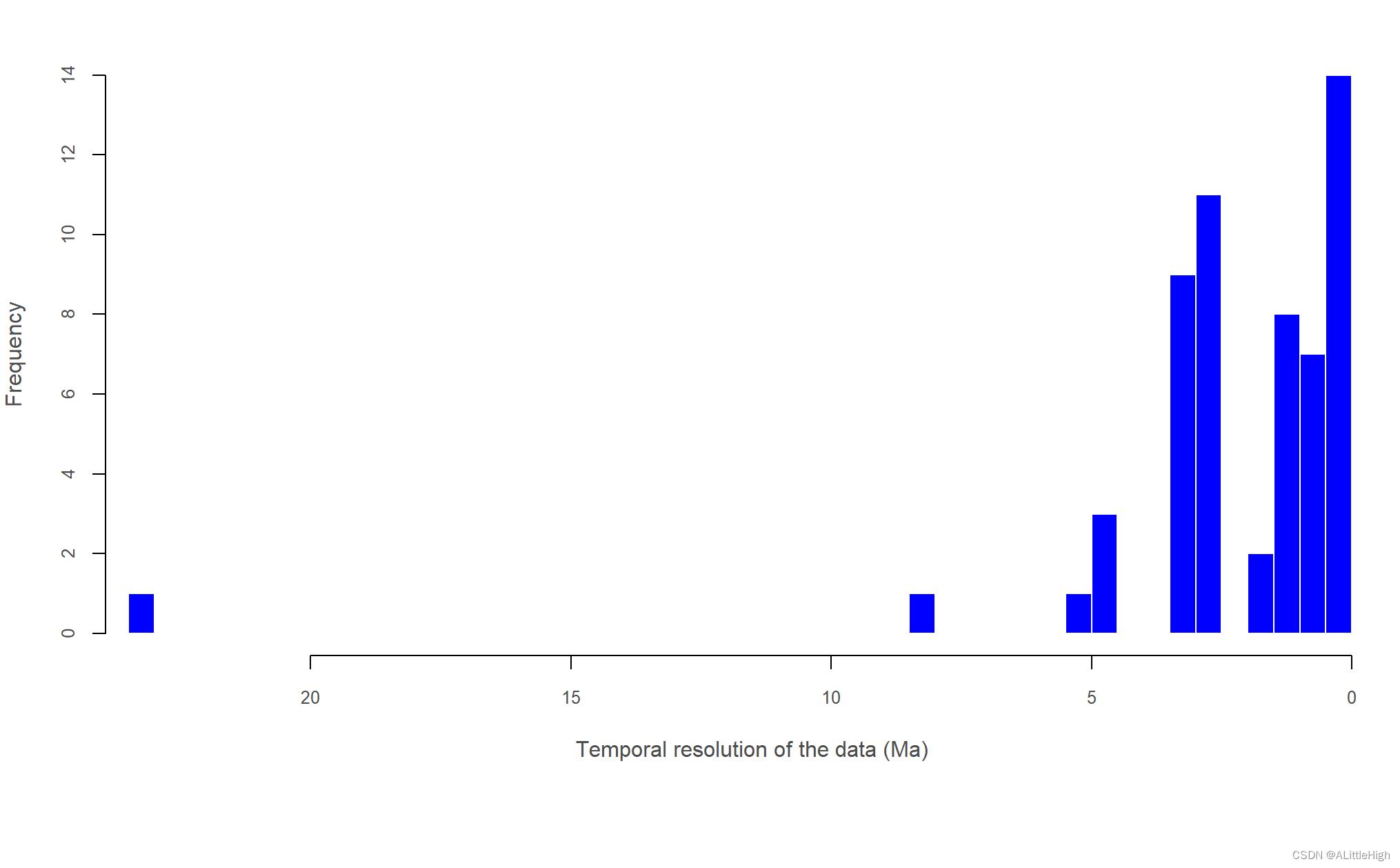
pbdb_temporal_resolution()会在控制台输出一个总览数据:最小值、上四分位值、中值、均值、下四分位值、最大值。随后会输出详细的值,和可视化图形:
# returns a dataframe and a plot with the temporal span of the species, genera, etc.
# 分析特定分类单元的年龄范围并图形化展示
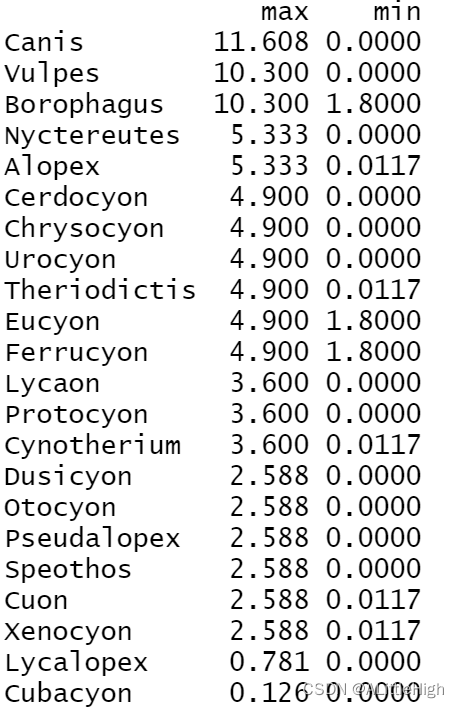
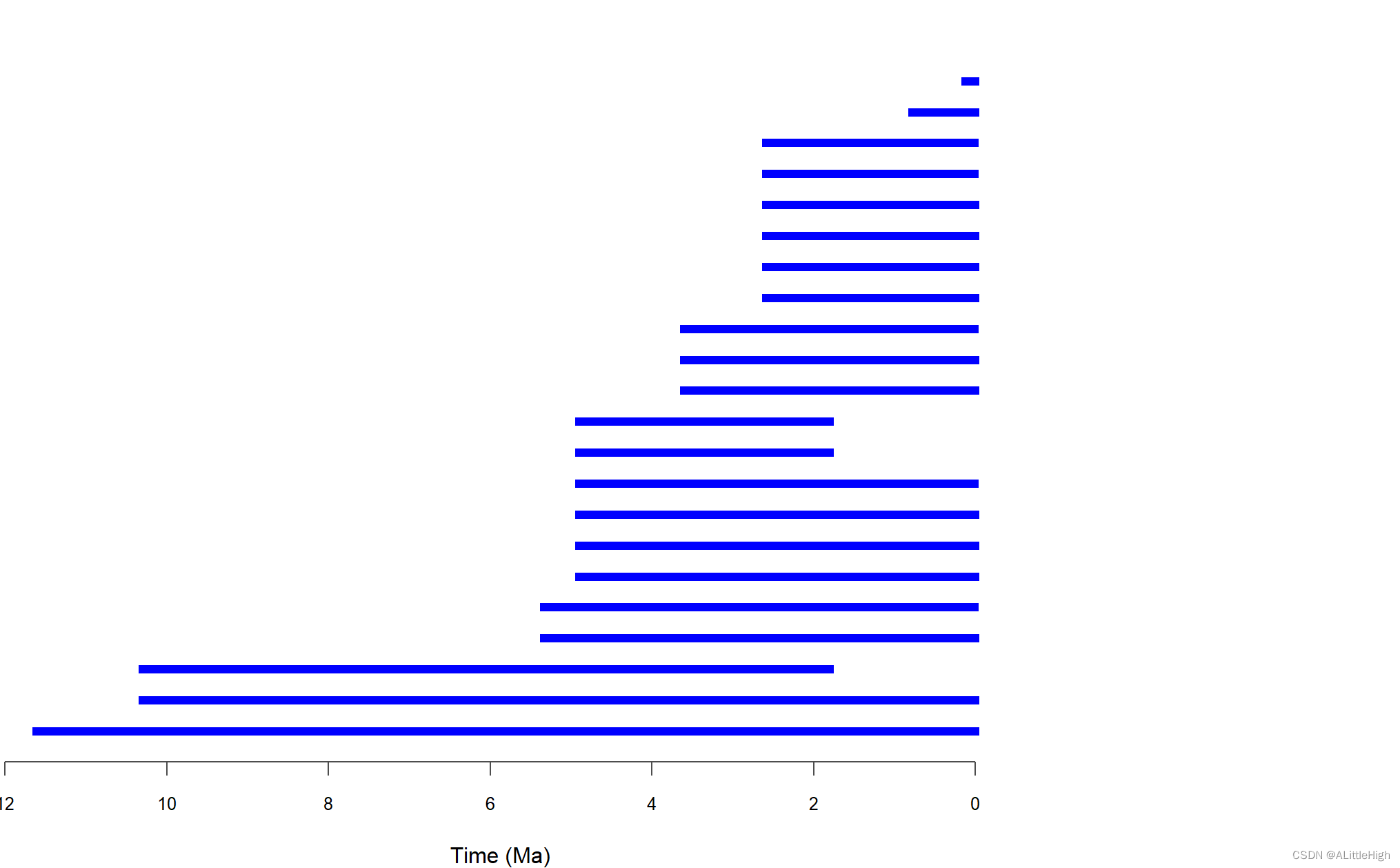
pbdb_temp_range(canidae, rank="genus", names=FALSE)
dataframe和图形的展示顺序相反:
# returns a dataframe and a plot showing the species, genera, etc. richness acrsoss time
pbdb_richness (canidae, rank="species", res=1, temporal_extent=c(0,10))
我在尝试pbdb_richness()时报错,但本人对R语言并不熟悉,查看了源码也不知如何修改,todo:
Error in data.frame(temporal_intervals, richness) :
参数值意味着不同的行数: 10, 11
# returns a dataframe and a plot showing the evolutionary and extinction rates across time
# evolutionary rates = evo_ext=1
pbdb_orig_ext(canidae, rank="genus", temporal_extent = c(0,10), res=1, orig_ext = 1)
pbdb_orig_ext()将展示指定分类阶元的演化和灭绝速率:
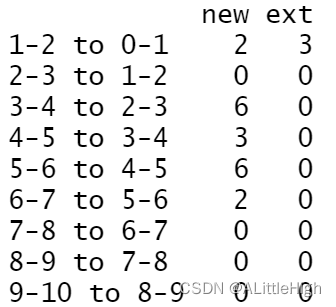
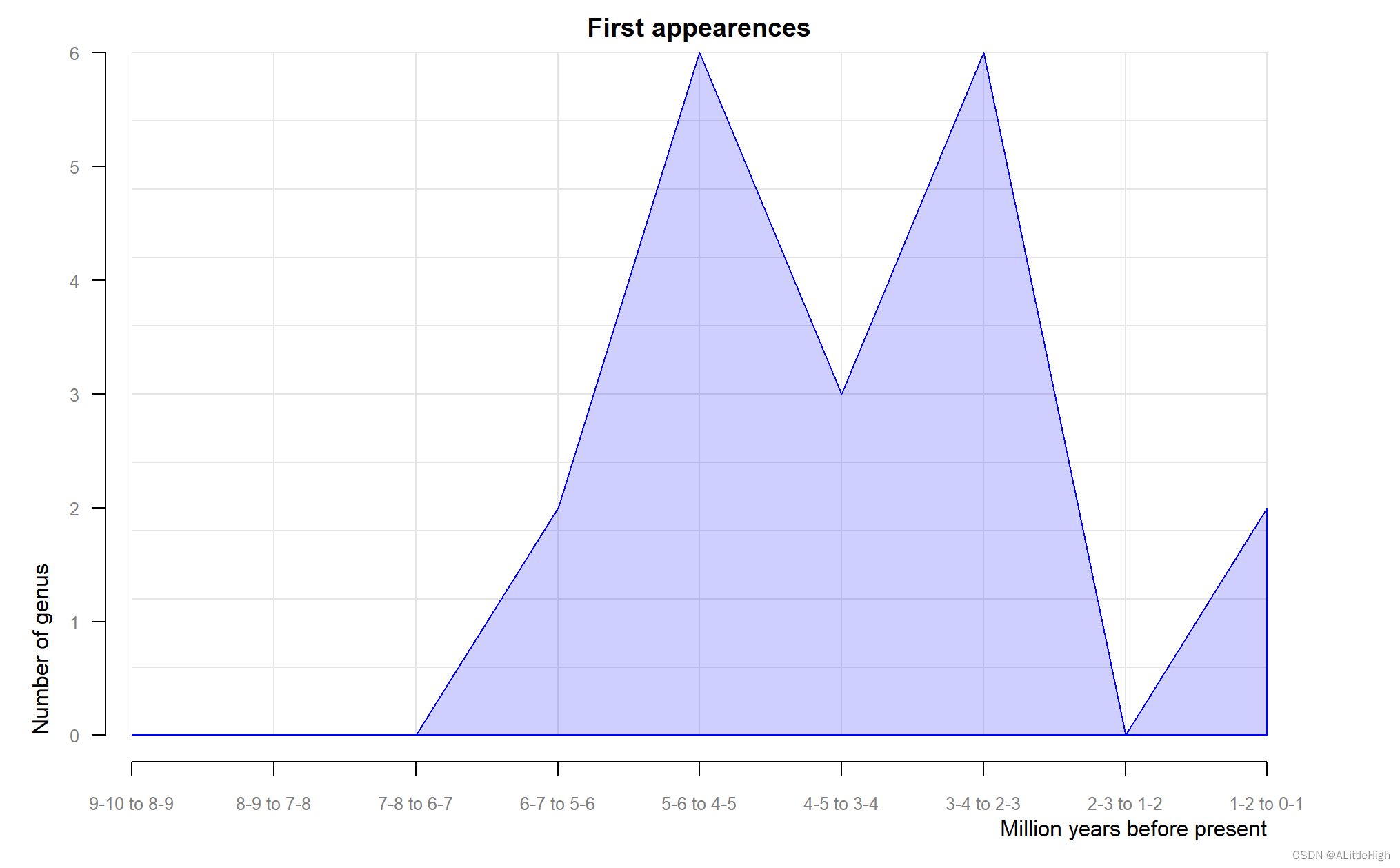
设置不同的演化速率会导致不同的结果:
# evolutionary rates = evo_ext=2
pbdb_orig_ext(canidae, rank="genus", temporal_extent = c(0,10), res=1, orig_ext = 2)

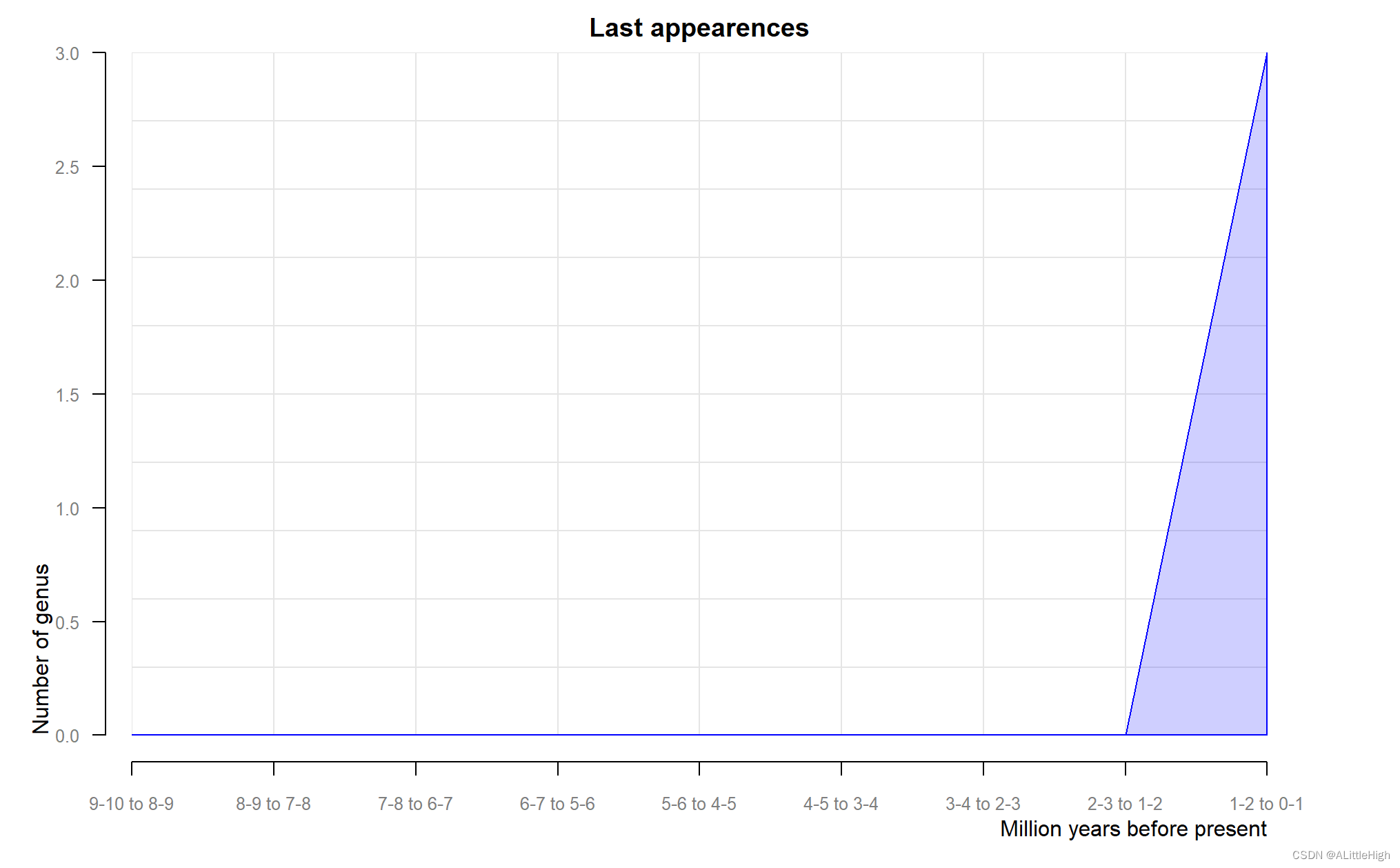
# maps the fossil occurrences
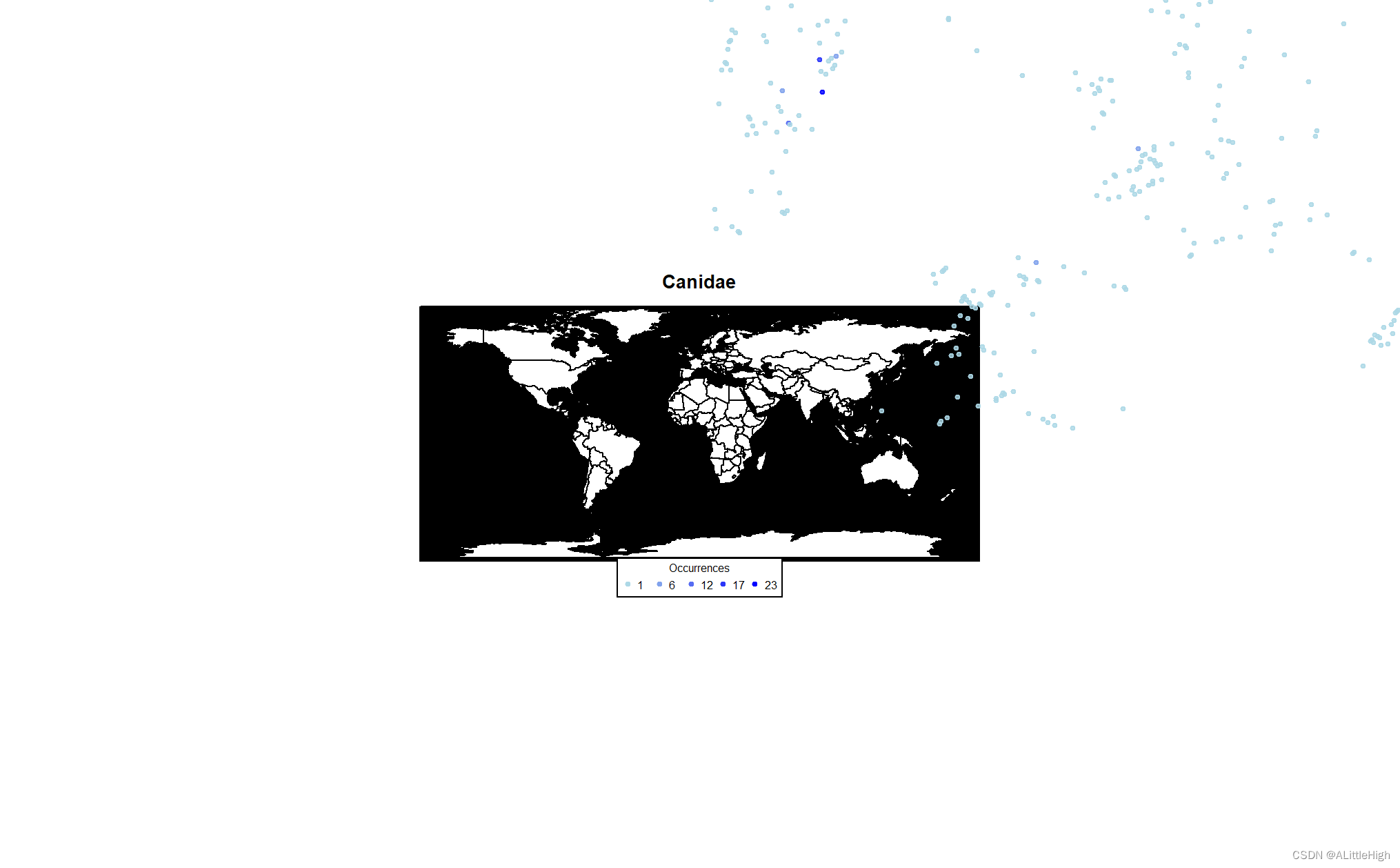
pbdb_map(canidae, main="Canidae", pch=19, cex=0.7)
该函数将化石记录绘制在地图上,下图可以看到结果错得离谱:
# maps the sampling effort
pbdb_map_occur(canidae, res=5)
此功能函数用于计算抽样误差,但会直接报错:
Error in .doCellFromXY(object@ncols, object@nrows, object@extent@xmin, :
Not compatible with requested type: [type=character; target=double].
# maps the species, genera, etc. richness
pbdb_map_richness(canidae, rank="species", res=5)
此功能函数用于展示指定分类阶元的丰度,但会直接报错:
Error in .doCellFromXY(object@ncols, object@nrows, object@extent@xmin, :
Not compatible with requested type: [type=character; target=double].
所有函数详解
1. pbdb_collection
描述
此函数会从PBDB返回一个化石记录信息。
用法
pbdb_collection (id, ...)
参数
- id:化石记录的标识符。此参数是必需的。
- …:传递给 API 的其他参数。查看 http://paleobiodb.org/data1.1/colls/single 中的所有可用参数。例如:
- vocab:设置 vocab=“pbdb” 以显示变量的完整名称(默认情况下变量具有简短的 3 个字母名称)
- show:显示额外变量
- …
细节
转到pbdb_occurrences以查看有关主要筛选参数的说明。
值
单个化石记录数据框
例子
pbdb_collection (id=1003, vocab="pbdb", show="loc")

2. pbdb_collections
描述
返回有关根据您提供的参数选择的多个化石记录的信息。
用法
pbdb_collections (...)
参数
- …:有关所有参数的文档,请参阅 http://paleobiodb.org/data1.1/colls/list 请转到pbdb_occurrences以查看有关主要筛选参数的说明
值
包含与查询值匹配的化石记录的数据框
例子
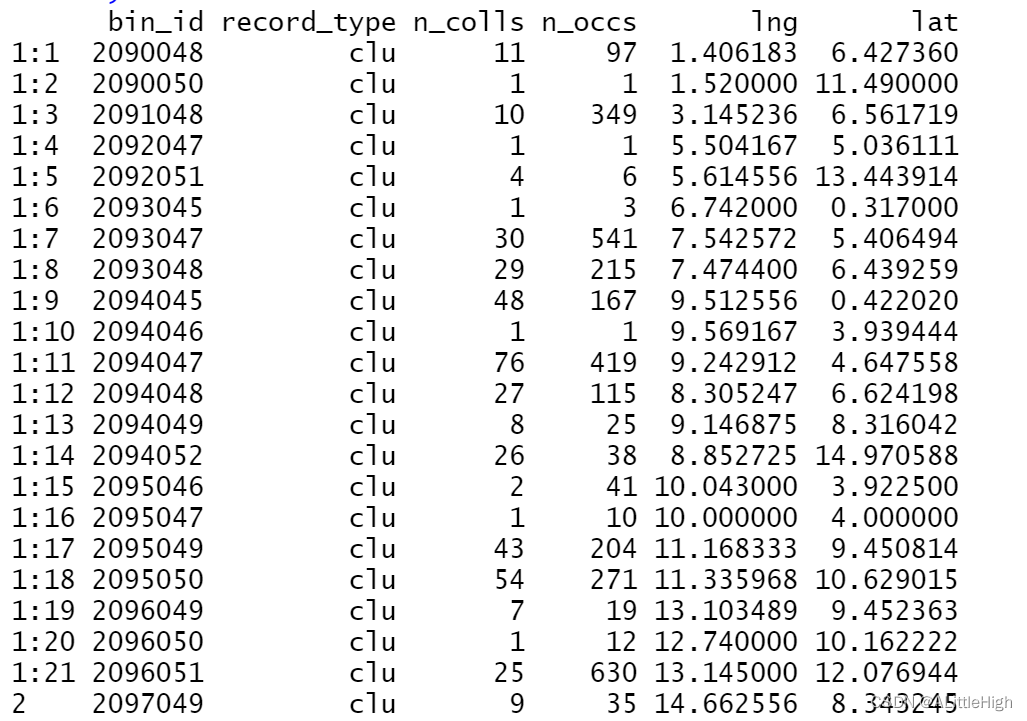
pbdb_collections (base_name="Cetacea", interval="Miocene")
3. pbdb_collections_geo
描述
此路径从PBDB中返回相关化石记录的地理集的信息。
用法
pbdb_collections_geo (...)
参数
- …:有关所有参数的文档,请参阅 http://paleobiodb.org/data1.1/colls/summary 请转到pbdb_occurrences以查看有关主要筛选参数的说明
值
包含与查询值匹配的化石记录的数据框
例子
pbdb_collections_geo (vocab="pbdb", lngmin=0.0,
lngmax=15.0, latmin=0.0, latmax=15.0, level=2)
4. pbdb_interval
描述
返回有关按标识符选择的单个时间间隔的信息。
用法
pbdb_interval (id, ...)
参数
- id:化石记录的标识符。此参数是必需的。
- …:传递给 API 的其他参数。有关 http://paleobiodb.org/data1.1/intervals/single 中接受的参数,请参阅文档。例如:
- vocab:设置 vocab=“pbdb” 以显示变量的完整名称(默认情况下变量具有简短的 3 个字母名称)
值
包含来自单个时间间隔的信息的数据框
例子
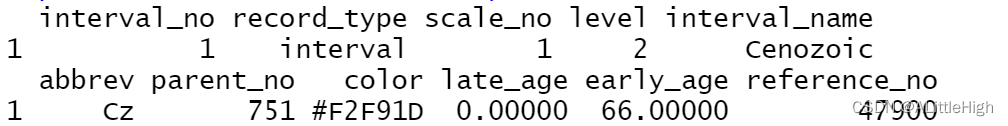
pbdb_interval (id=1, vocab="pbdb")
5. pbdb_intervals
描述
返回有关根据您提供的参数选择的多个间隔的信息。
用法
pbdb_intervals (...)
参数
- …:传递给 API 的参数。有关 http://paleobiodb.org/data1.1/intervals/list 中接受的参数,请参阅文档。例如:
- min_ma:仅返回不晚于给定间隔的结果
- max_ma:仅返回不早于给定间隔的结果
- order:按指定顺序返回间隔。可能的值包括较老、较年轻。默认为更年轻
- vocab:设置 vocab=“pbdb” 以显示变量的完整名称(默认情况下变量具有简短的 3 个字母名称)
- …
值
包含来自多个时间间隔的信息的数据框
例子
pbdb_intervals (min_ma= 0, max_ma=2, vocab="pbdb")

6. pbdb_map
描述
绘制化石记录地图分布
用法
pbdb_map (data, col.int="white" ,pch=19, col.ocean="black", main=NULL,
col.point=c("light blue","blue"), ...)
参数
- data:输入数据框。此数据框是pbdb_occurrences函数设置参数show="coords"的输出结果
- col.int:大陆的颜色
- pch:参见graphics库的par。输入一个整数或字符来设置散点图标。
- col.ocean:大洋的颜色
- main:参见graphics库的par。设置地图的标题。
- col.point:两个或更多颜色。生成用于显示地图中每个单元格出现次数的颜色渐变
- …:其余参数。参见graphics库的par和maps库的map。
细节
该函数将打开一个地图新窗口。
==注意:==pbdb_occurrences函数的参数设置show="corrds"是必需的。为了更好的可视化图形,开发者建议使用Cairo库。
值
根据每个网格的出现次数显示化石记录分布的地图,其中点带有颜色渐变。
例子
data = pbdb_occurrences (limit="all", vocab= "pbdb",
base_name="Canis", show="coords")
X11(width=12, height=8)
pbdb_map(data,pch=19,col.point=c("pink","red"), col.ocean="light blue",
main="canis")
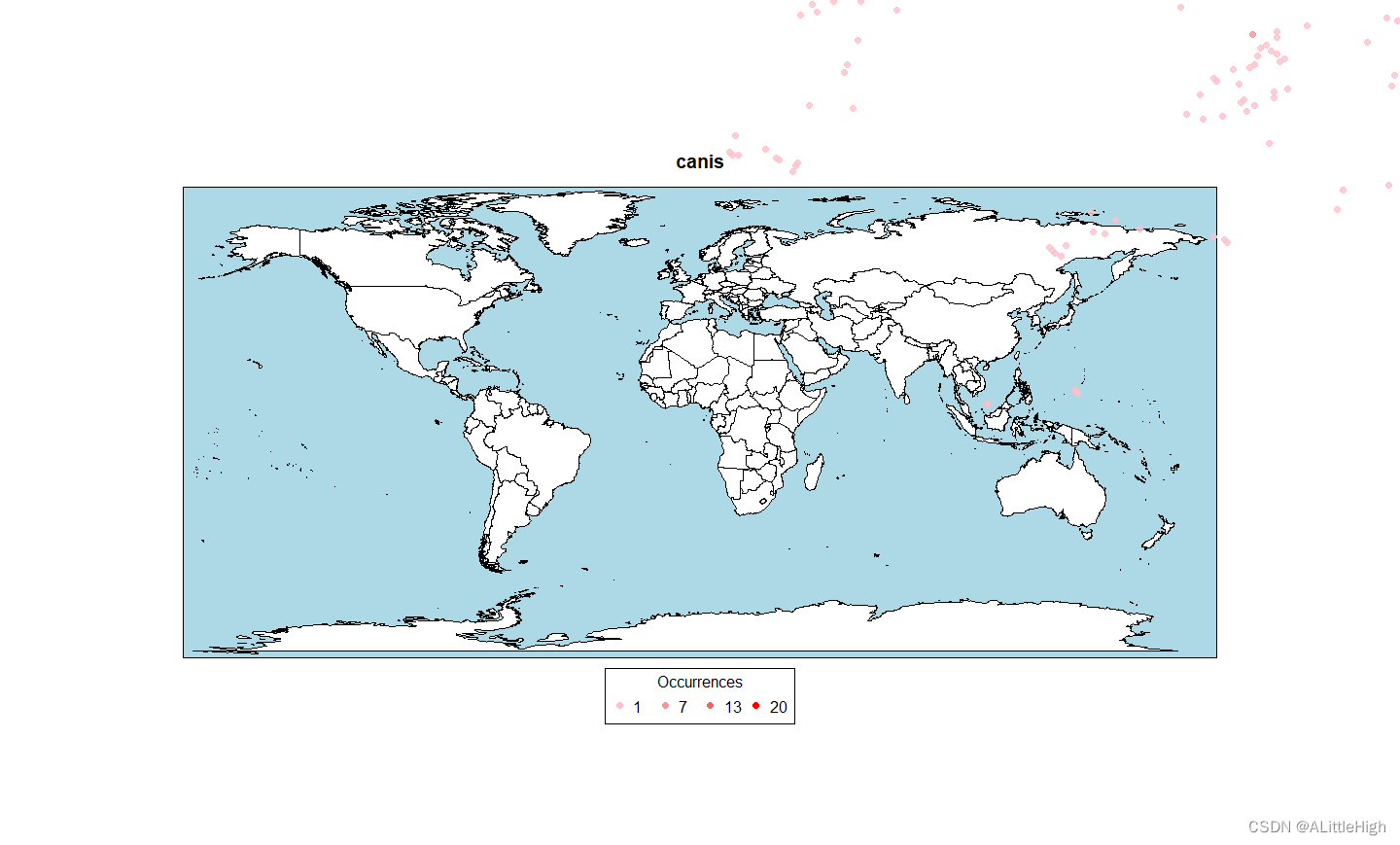
图像仍然是错误的
7. pbdb_map_occur
描述
创建栅格图层对象和采样工作图(每个网格的化石记录数)。
用法
pbdb_map_occur (data, res=5, col.int="white", col.ocean="black",
col.eff=c("light blue","blue"), do.plot=TRUE, ...)
参数
- data:输入数据框。此数据框是pbdb_occurrences函数设置参数show="coords"的输出结果
- res:栅格图层对象的分辨率。参见raster库的raster。
- col.int:大陆的颜色。
- col.ocean:大洋的颜色。
- col.eff:两个或更多颜色。生成用于显示地图中每个单元格出现次数的颜色渐变
- do.plot:逻辑变量;TRUE允许该函数返回一个栅格图层对象和一个图像。
- …:其余参数。参见graphics库的par和maps库的map。
细节
==注意:==pbdb_occurrences函数的参数设置show="corrds"是必需的。为了更好的可视化图形,开发者建议使用Cairo库。
值
一个栅格图层对象和一个显示取样量的图形(每个网格中的化石记录数)。通过设置参数res改变栅格图层的分辨率。默认为res=1。
例子
data = pbdb_occurrences (limit="all", vocab= "pbdb", base_name="Canis",
show="coords")
X11(width=13, height=7.8)
pbdb_map_occur (data,res=2)
## to obtain the raster file without plotting it
pbdb_map_occur (data,res=3,do.plot=F)
仍然出现报错:
Error in .doCellFromXY(object@ncols, object@nrows, object@extent@xmin, :
Not compatible with requested type: [type=character; target=double].
8. pbdb_map_richness
描述
创建一个栅格图层对象,绘制种、属、科等分类阶元在每个网格上的丰富度的图像。
用法
pbdb_map_richness (data, rank="species", do.plot=TRUE, res=5,
col.int="white", col.ocean="black",
col.rich=c("light blue","blue"),...)
参数
- data:输入数据框。此数据框是pbdb_occurrences函数设置参数show=c(“phylo”, “coords”, “ident”)的输出结果
- rank:设置为您想计算的分类阶元丰富度。可选项有:“species”, “genus”, “family”, “order”, “class” or “phylum”。
- do.plot:逻辑变量;TRUE允许该函数返回一个栅格图层对象和一个图像。
- res:栅格图层对象的分辨率。参见raster库的raster。
- col.int:大陆的颜色。
- col.ocean:大洋的颜色。
- col.rich:两个或更多颜色。生成用于显示地图中每个单元格出现次数的颜色渐变
- …:其余参数。参见graphics库的par和maps库的map。
细节
==注意:==pbdb_occurrences函数的参数设置show="corrds"是必需的。为了更好的可视化图形,开发者建议使用Cairo库。
值
一个栅格图层对象和一个显示指定分类阶元丰富度的图形(每个网格中的化石记录数)。通过设置参数res改变栅格图层的分辨率。默认为res=1。
例子
data = pbdb_occurrences (limit=1000, vocab= "pbdb", base_name="mammalia",
show=c("phylo","coords","ident"))
X11(width=13, height=7.8)
pbdb_map_richness (data,res=8,rank="genus")
pbdb_map_richness (data,res=8,rank="family")
## to obtain the raster file and not plot the map
pbdb_map_richness (data,res=8,rank="family",do.plot=F)
毫无悬念地报错了
Error in .doCellFromXY(object@ncols, object@nrows, object@extent@xmin, :
Not compatible with requested type: [type=character; target=double].
9. pbdb_occurrence
描述
从古生物学数据库返回有关单个产出记录的信息。
用法
pbdb_occurrence (id, ...)
参数
- id:产出记录的标识符。此参数是必需的。
- …:传递给 API 的参数。查看 http://paleobiodb.org/data1.1/occs/single 中的所有可用参数。例如:
- vocab:词汇:设置 vocab=“PBDB” 以显示变量的完整名称(默认情况下变量具有 3 个字母的短名称)
细节
有关所有参数的文档,请访问 http://paleobiodb.org/data1.1/occs/single。在上面的参数列表中,我们描述了古生物学家和生态学家可能使用的最常见的过滤器。
值
单个产出记录的数据框
例子
pbdb_occurrence (id=1001, vocab="pbdb", show="coords")

10. pbdb_occurrences
描述
返回有关存储在PBDB中的物种产出记录的信息。
用法
pbdb_occurrences(...)
参数
- …:传递给 API 的参数。查看 http://paleobiodb.org/data1.1/occs/list 中的所有可用参数
- limit:将限制设置为“all”以下载所有匹配项。默认情况下,限制为 500。
- taxon_name:仅返回与指定分类名称关联的记录。您可以指定多个名称,用逗号分隔。
- base_name:返回与指定分类学名称及其任何子项关联的记录
- lngmin:数值类型。经度边界将被规范化为介于 -180 和 180 之间。请注意,如果指定 lngmin,则还必须指定 lngmax。仅返回地理位置在给定边界框(由 lngmin、lngmax、latmin、latmax 定义)内的记录。如果范围与反计时符相交,它将生成两个相邻的边界框。
- lngmax:数值类型。经度边界将被规范化为介于 -180 和 180 之间。
- latmin:数值类型。介于 -90 和 90 之间。请注意,如果指定 latmin,则还必须指定 latmax。
- latmax:数值类型。介于 -90 和 90 之间。
- min_ma:仅返回不晚于给定间隔的结果
- max_ma:仅返回不早于给定间隔的结果
- interval:仅返回其时间段在指定地质时间间隔内的记录(例如“Miocene”)。
- continent:仅返回地理位置在指定大洲内的记录。
- show:展示额外的变量信息(例如coords,phylo,ident)
细节
有关所有参数的文档,请访问 http://paleobiodb.org/data1.1/occs/list。我们描述了古生物学家和生态学家可能在上面的参数列表中使用的最常见的过滤器。
值
物种产出记录的数据框
例子
pbdb_occurrences (id=c(10, 11), show=c("coords", "phylo", "ident"))

pbdb_occurrences (limit="all", vocab= "pbdb",
taxon_name="Canis", show=c("coords", "phylo", "ident"))
11. pbdb_orig_ext
描述
绘制新分类群随时间的变化。
用法
pbdb_orig_ext (data, rank,
temporal_extent, res, orig_ext,
colour="#0000FF30", bord="#0000FF", do.plot=TRUE)
参数
- data:使用pbdb_occurrences函数获取的数据框。重要的是,需要显示科、目等名称。在数据帧中,要执行该集合:show=c(“phylo”, “ident”)
- rank:设置您感兴趣的分类单元排名。默认情况下rank= “species”
- temporal_extent:用于设置时间段(最小值、最大值)的矢量
- res:数值类型。设置temporal_extent的时间段
- orig_ext:1代表起源,2代表灭绝
- colour:改变图像条形的颜色,默认是skyblue2.
- bord:设置多边形边缘颜色
- do.plot:TRUE/FALSE(默认为TRUE)
值
包含所选分类单元随时间推移的首次出现和灭绝次数的数据框,以及包含所选分类单元随时间推移的首次出现或灭绝的图。
例子
canidae = pbdb_occurrences (limit="all", vocab="pbdb",
+ base_name="Canidae", show=c("phylo", "ident"))
pbdb_orig_ext (canidae, rank="genus", temporal_extent=c(0, 10),
+ res=1, orig_ext=1)
pbdb_orig_ext (canidae, rank="species", temporal_extent=c(0, 10),
res=1, orig_ext=2)

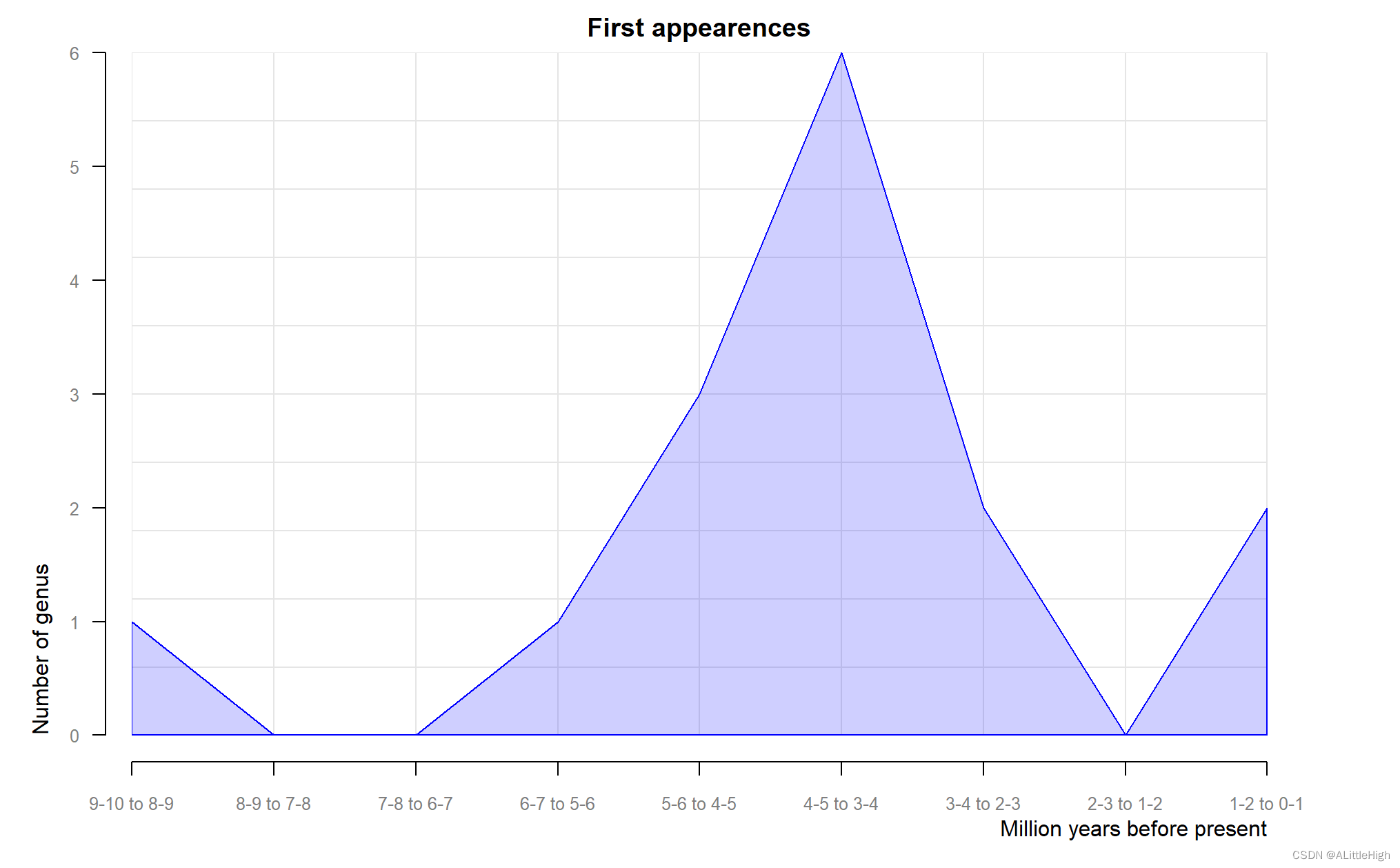

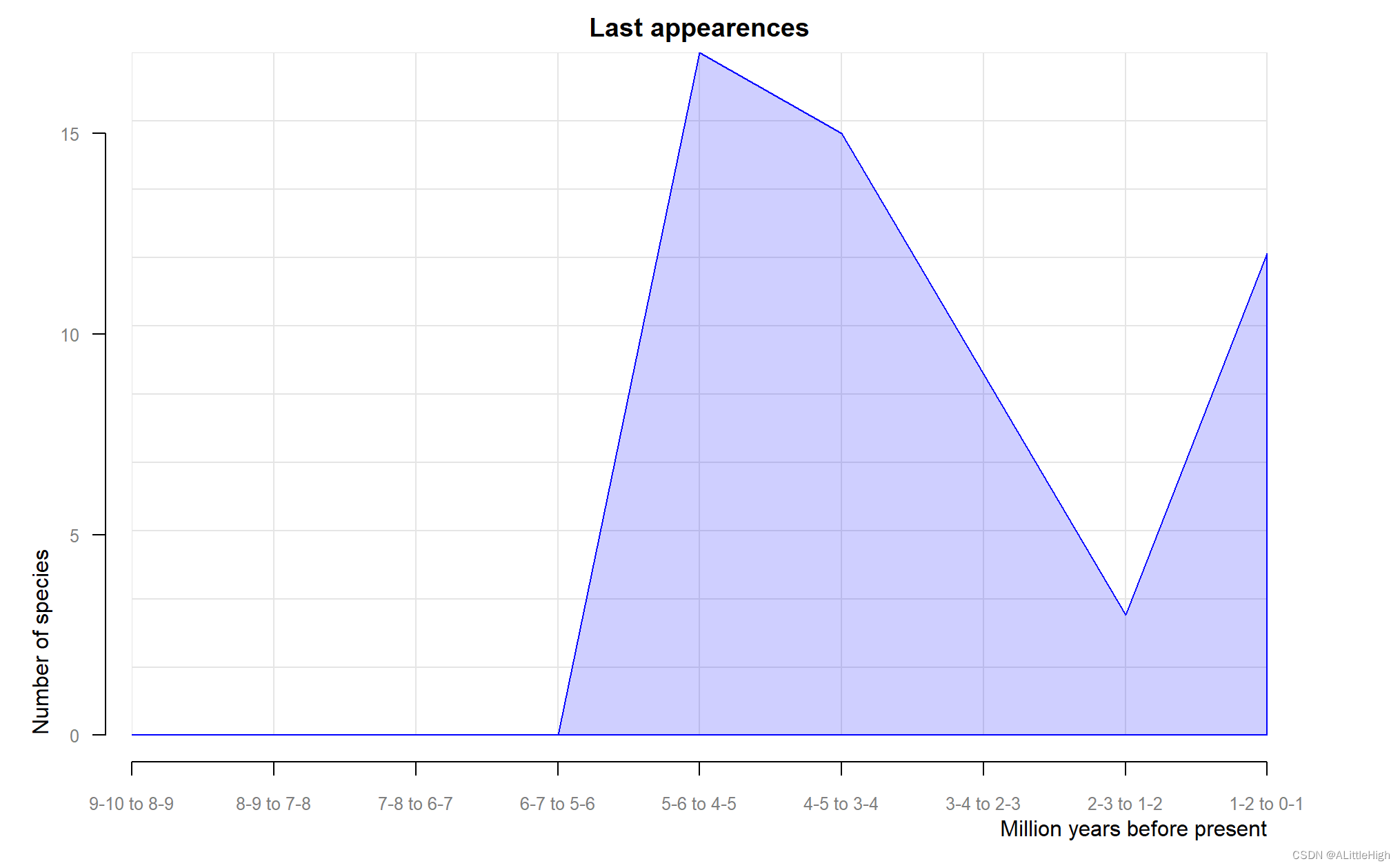
12. pbdb_reference
描述
返回有关按标识符选择的单个引用的信息。转到pbdb_occurrences以查看有关主要筛选参数的说明
用法
pbdb_reference (id, ...)
参数
- id:引用的标识符。此参数是必需的。
- …:传递给 API 的参数。有关 http://paleobiodb.org/data1.1/refs/single 中接受的参数,请参阅文档。
值
具有单个引用的数据框
例子
pbdb_collection (id=1003, vocab="pbdb", show="loc")
13. pbdb_references
描述
返回有关根据您提供的参数选择的多个引用的信息。
用法
pbdb_references (...)
参数
- …:传递给 API 的参数。有关 http://paleobiodb.org/data1.1/refs/list 中接受的参数,请参阅文档。
值
一个数据框,其中包含有关与查询匹配的引用的信息
例子
pbdb_references (author= "Polly")
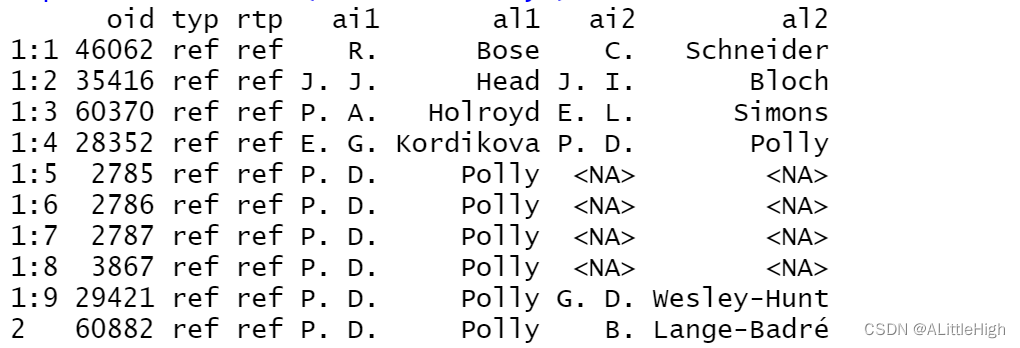
14. pbdb_ref_collections
描述
返回有关从中输入所选集合数据的引用的信息。
用法
pbdb_ref_collections (...)
参数
- …:传递给 API 的参数。有关 http://paleobiodb.org/data1.1/colls/refs 中接受的参数,请参阅文档。
值
一个数据框,其中包含有关与查询匹配的引用的信息
例子
pbdb_ref_collections (id=1)
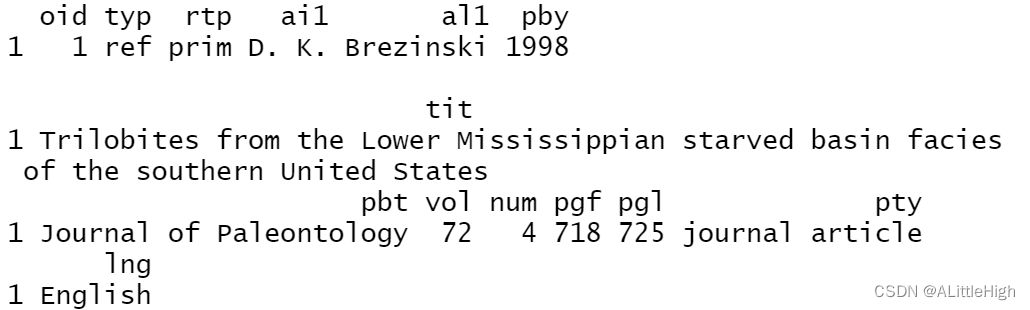
15. pbdb_ref_occurrences
描述
从数据库中返回有关与化石事件关联的书目参考的信息。
用法
pbdb_ref_occurrences (...)
参数
- …:传递给 API 的参数。查看 http://paleobiodb.org/data1.1/occs/refs 中的所有可用参数
值
一个数据框,其中包含有关与查询匹配的引用的信息
例子
pbdb_ref_occurrences (vocab="pbdb",
base_name="Canis", year=2000)
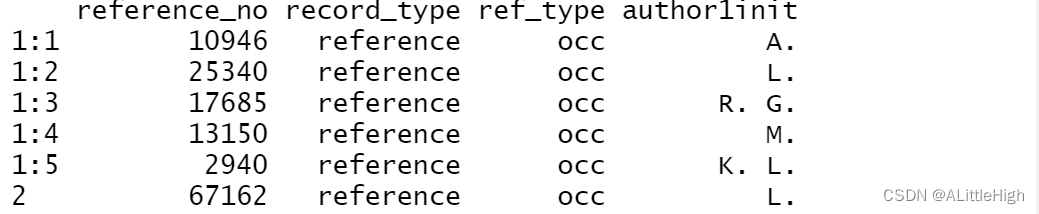
16. pbdb_ref_taxa
描述
此 URL 路径返回有关与古生物学数据库中的分类群关联的源引用的信息。您可以使用与pbdb_taxa相同的参数,但返回引用记录而不是分类记录。每个引用返回一条记录,即使它与多个分类群相关联也是如此。
用法
pbdb_ref_taxa (...)
参数
- …:传递给 API 的参数。查看 http://paleobiodb.org/data1.1/taxa/refs 中的所有可用参数
值
包含来自分类群列表的引用的数据框
例子
pbdb_ref_taxa (name="Canidae", vocab="pbdb", show=c("attr", "app", "size", "nav"))
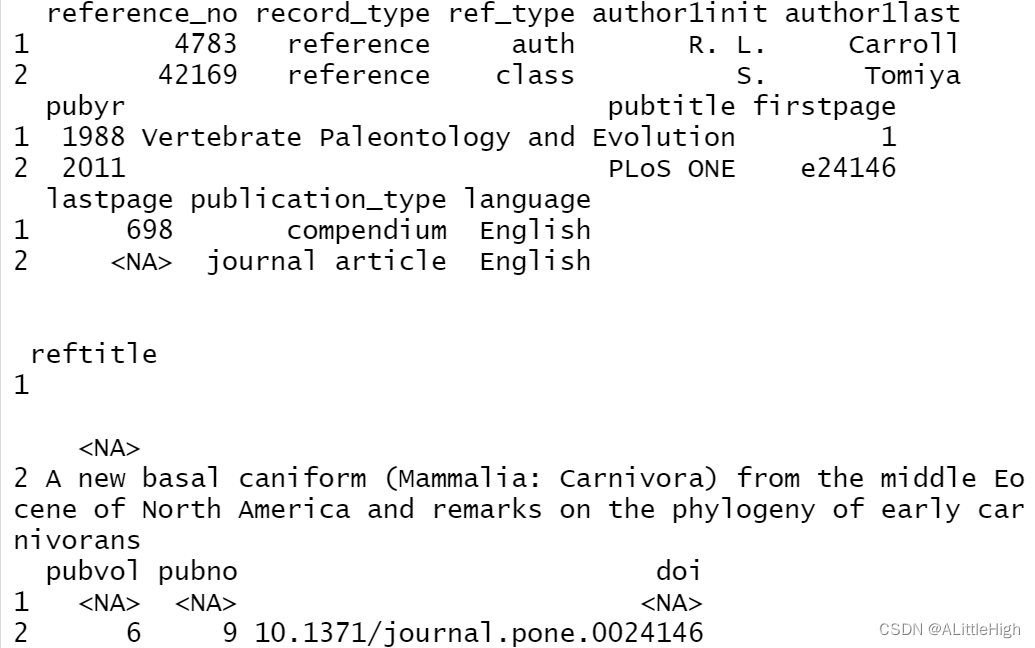
17. pbdb_richness
描述
绘制目标分类阶元的数量图
用法
pbdb_richness (data, rank, res, temporal_extent, colour, bord, do.plot)
参数
- data:数据框与我们对pbdb_occurrences的查询。重要的是,需要显示科、目等名称。在数据帧中,要执行该集合:show=c(“phylo”, “ident”)
- rank:设置您感兴趣的分类单元排名。默认情况下rank= “species”
- temporal_extent:用于设置时间段(最小值、最大值)的矢量
- res:数值类型。设置temporal_extent的时间段
- orig_ext:1代表起源,2代表灭绝
- colour:改变图像条形的颜色,默认是skyblue2.
- bord:设置多边形边缘颜色
- do.plot:TRUE/FALSE(默认为TRUE)
值
一个图和数据框,其丰富度由指定时间段和分辨率下的分类单元阶元聚合。
例子
data = pbdb_occurrences (limit="all", vocab="pbdb",
base_name="Canidae", show=c("phylo", "ident"))
pbdb_richness (data, rank="species", res=1, temporal_extent=c(0,3))

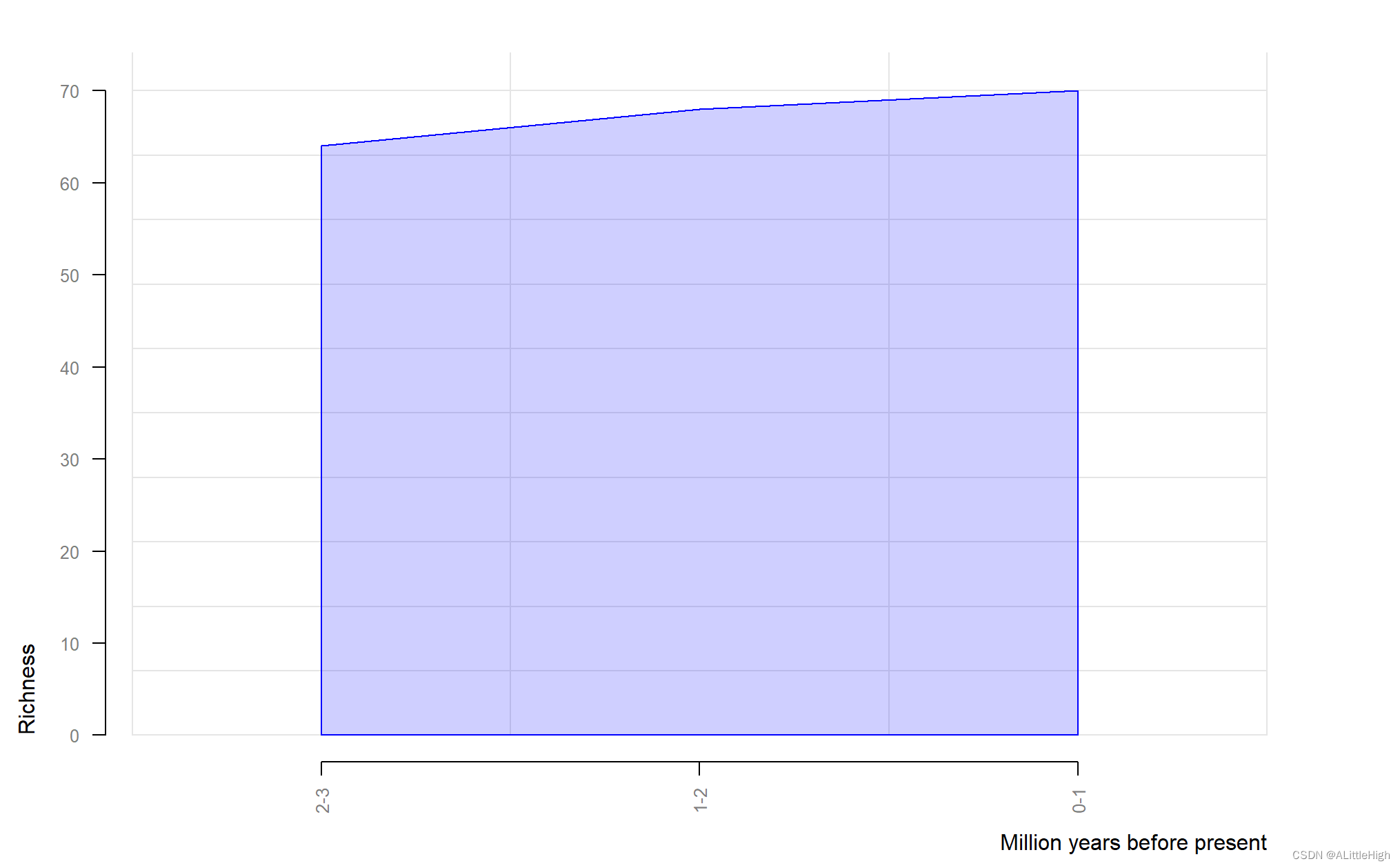
18. pbdb_scale
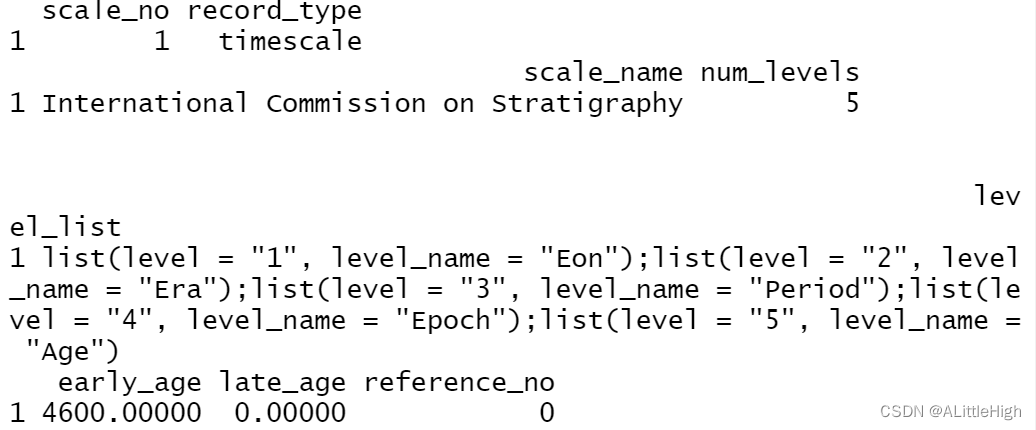
描述
返回有关按标识符选择的单个时间刻度的信息。
用法
pbdb_scale (id, ...)
参数
- id: 时间间隔的标识符。此参数是必需的。
- …:传递给 API 的其他参数。有关 http://paleobiodb.org/data1.1/scales/single 中接受的参数,请参阅文档。
值
包含来自单个时间刻度的信息的数据框
例子
pbdb_scale (id=1, vocab="pbdb")
19. pbdb_scales
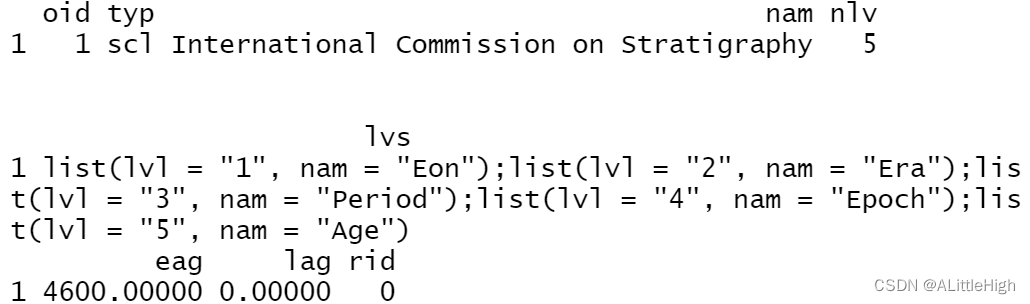
描述
返回有关多个时间刻度的信息。
用法
pbdb_scales(...)
参数
- …:传递给 API 的参数。有关 http://paleobiodb.org/data1.1/scales/list 中接受的参数,请参阅文档。
值
包含所选刻度信息的数据框
例子
pbdb_scales ()
20. pbdb_strata
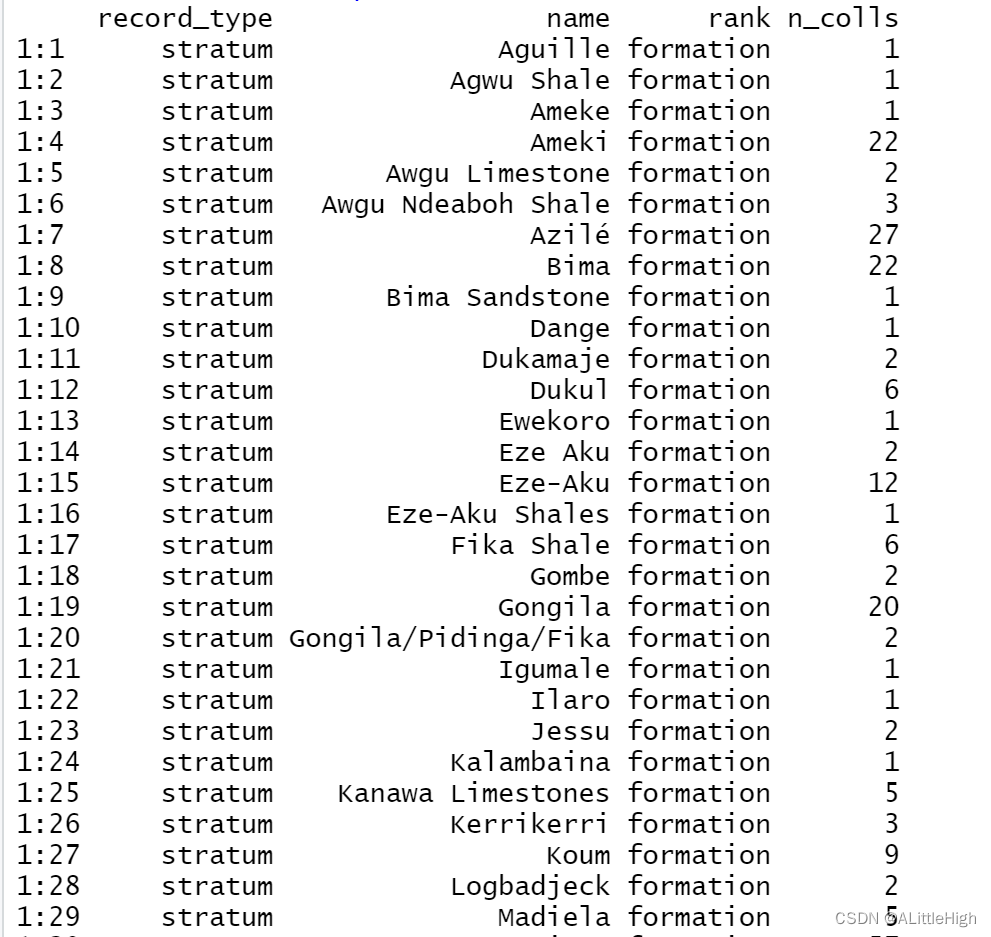
描述
返回有关按名称、等级和/或地理位置选择的地质地层的信息。
用法
pbdb_strata (...)
参数
- …:传递给 API 的参数。有关 http://paleobiodb.org/data1.1/strata/list 中接受的参数,请参阅文档。
值
包含来自所选地层的信息的数据框
例子
pbdb_strata (lngmin=0, lngmax=15, latmin=0, latmax=15, rank="formation", vocab="pbdb")
21. pbdb_strata_auto
描述
返回与给定前缀或部分名称匹配的地层列表。这可用于实现地层名称的自动完成,如果需要,可以受地理位置的限制。
用法
pbdb_strata_auto (...)
参数
- …:传递给 API 的参数。有关 http://paleobiodb.org/data1.1/strata/auto 中接受的参数,请参阅文档。
值
一个数据框,其中包含与我们的字母匹配的地层信息。
例子
pbdb_strata_auto (name= "Pin", vocab="pbdb")

22. pbdb_subtaxa
描述
计算给定分类群中的子分类群数量。例如,一个属内的物种数量。
用法
pbdb_subtaxa (data, do.plot, col)
参数
- data:数据框,其中包含我们对古BD pbdb_occurrences的查询
- do.plot:默认情况下,此函数制作一个图来可视化分类群的分布。设置为 FALSE 可跳过绘图。
- col:设置直方图的颜色。默认情况下为skyblue2。
值
包含数据中子分类群数的绘图和数据框。
例子
canidae_quat = pbdb_occurrences (limit="all",
base_name="Canidae", interval="Quaternary",
show=c("coords", "phylo", "ident"))
pbdb_subtaxa (canidae_quat)
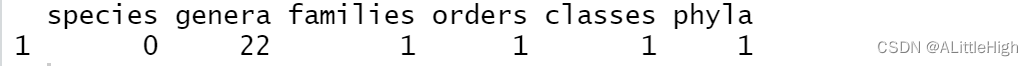
23. pbdb_taxa
描述
返回有关多个分类名称的信息。此函数可用于查询给定分类单元的所有子项或父项,以及其他操作。
用法
pbdb_taxa (...)
参数
- …:传递给 API 的参数。查看 http://paleobiodb.org/data1.1/taxa/list 中的所有可用参数
值
包含来自分类群列表的信息的数据框
例子
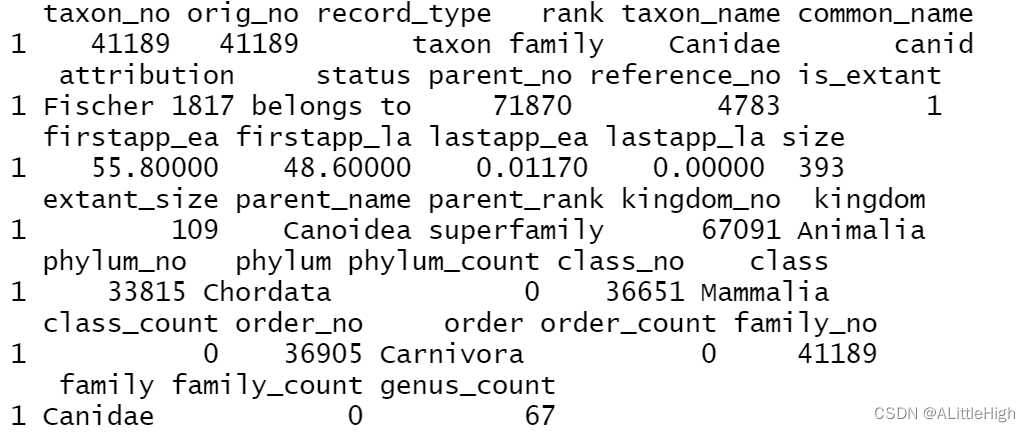
pbdb_taxa (name="Canidae", vocab="pbdb",
show=c("attr", "app", "size", "nav"))
24. pbdb_taxa_auto
描述
返回与给定前缀或部分名称匹配的名称列表。
用法
pbdb_taxa_auto (...)
参数
- …:传递给 API 的参数。有关 http://paleobiodb.org/data1.1/taxa/auto_doc.html 中接受的参数,请参阅文档。
值
包含有关匹配项的信息的数据框(分类单元排名和数据库中的出现次数)
例子
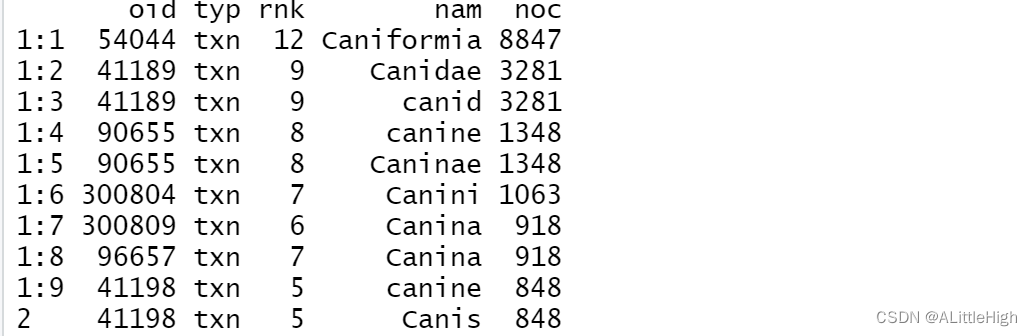
pbdb_taxa_auto (name="Cani", limit=10)
25. pbdb_taxon
描述
返回有关单个分类名称(按名称或标识符标识)的信息。
用法
pbdb_taxon (...)
参数
- …:传递给 API 的参数。有关 http://paleobiodb.org/data1.1/taxa/single 中接受的参数,请参阅文档。
值
包含来自单个分类单元的信息的数据框
例子
pbdb_taxon (name="Canis", vocab="pbdb",
show=c("attr", "app", "size"))
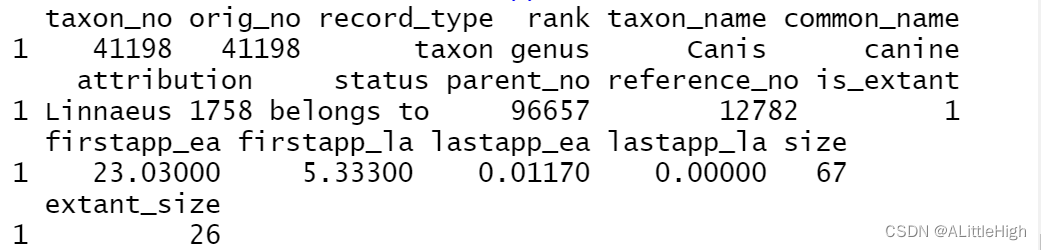
26. pbdb_temporal_resolution
描述
显示化石数据的时间分辨率
用法
pbdb_temporal_resolution (data, do.plot=TRUE)
参数
- data:数据框,其中包含我们对pbdb_occurrences的查询
- do.plot:TRUE/FALSE。显示数据的频率图(默认为 TRUE)。
值
一个图和一个列表,其中包含数据的时间分辨率(最小值、最大值、第 1 和第 3 个四分位数、中位数和平均值)以及每个化石记录的时间分辨率 (Ma)。
例子
data = pbdb_occurrences (taxon_name= "Canidae", interval= "Quaternary")
pbdb_temporal_resolution (data)
27. pbdb_temp_range
描述
构建一个图和一个数据框,其中包含所选高等分类单元中分类群(物种、属、科等)的时间范围。
用法
pbdb_temp_range (data, rank, col = "#0000FF",
names = TRUE, do.plot =TRUE)
参数
- data:数据框与我们对pbdb_occurrences的查询。重要的是,需要显示科、目等名称。在数据帧中,要执行该集合:show=c(“phylo”, “ident”)
- rank:设置您感兴趣的分类单元排名。
- do.plot:TRUE/FALSE。(默认为 TRUE)。
- col:设置直方图的颜色。默认情况下为skyblue2。
- names:TRUE/FALSE(默认为TRUE)。图中是否显示类群的名称。
值
包含所选分类群(物种、属等)的时间跨度的图和数据框
例子
canis_quaternary = pbdb_occurrences (limit="all", base_name="Canis",
interval="Quaternary", show=c("coords", "phylo", "ident"))
pbdb_temp_range (canis_quaternary, rank="species", names=FALSE)
结果报错:
Error in data.frame(..., check.names = FALSE) :
参数值意味着不同的行数: 0, 2