预测药物-靶点相互作用是药物发现的关键。最近基于深度学习的方法表现出良好的性能,但仍然存在两个挑战:
- 如何明确地建模和学习药物和靶标之间的局部相互作用,以更好地预测和解释;
- 如何优化新型药物-靶标对预测的泛化性能。
在这里,作者提出了一种具有域自适应的深度双线性注意力网络框架DrugBAN,用于明确地学习"药物-靶标pair"之间的局部相互作用,并适应于分布外数据。DrugBAN对药物分子图和靶蛋白序列进行预测,使用条件领域对抗学习在不同分布上对齐学习到的相互作用表示,以便更好地泛化到新的"药物-靶pair"。在域内和跨域设置下的三个基准数据集上的实验表明,与五个最先进的基线模型相比,DrugBAN获得了最佳的总体性能。此外,将学习到的双线性注意力图可视化,可以从预测结果中提供可解释的见解。
来源:Interpretable bilinear attention network with domain adaptation improves drug–target prediction
目录
- 背景介绍
- 方法
- 双线性注意力网络
- 领域自适应
- 可解释性可视化
背景介绍
尽管目前出现了很多优秀的DTI预测方法,但现有的基于DL的方法仍存在两个挑战。
第一个挑战是如何明确了解药物和蛋白质的局部结构之间的相互作用。DTI基本上由药物化合物中的重要分子亚结构和蛋白质序列中的结合位点之间的相互作用决定。然而,许多先前的研究在各自的编码器中只学习全局表示,而没有明确地学习药物和靶标之间的局部交互。单独的全局表示学习往往会限制建模能力和预测性能。此外,如果没有对局部交互的显式学习,即使模型的预测是准确的,其预测结果也很难解释。
第二个挑战是跨域泛化预测性能,即预测分布外(out of learned distribution)数据。由于化学空间和基因组空间巨大,实际应用中需要被预测的"药物-靶标pair"通常是不可见的,并且它们与训练数据中的任何pair分布都不同,所以需要跨域建模。一个鲁棒的模型应该能够将学习到的知识转移到一个未标记数据的新领域。在这种情况下,作者需要通过学习可转移表示(例如从source到target)来调整分布并提高跨域泛化性能。目前,这是药物发现的一个尚未探索的方向。
关于药物-药物相互作用预测,也有一篇预测分布外数据的工作:DSIL-DDI: A Domain-Invariant Substructure Interaction Learning for Generalizable Drug–Drug Interaction Prediction
区别:DrugBAN需要领域自适应的学习过程(源域有标签,目标域无标签,但需要对抗学习对齐两个域的表示),DSIL-DDI直接在源域学习到通用的表示,无需领域自适应学习。
方法
DrugBAN属于CPI中的Y型架构,靶标蛋白记为 P = ( a 1 , . . . , a n ) P=(a_{1},...,a_{n}) P=(a1,...,an),其中 a i a_{i} ai为23个氨基酸中的某一个。药物需要将SMILES转为2D分子图 G = ( V , E ) G=(V,E) G=(V,E)。

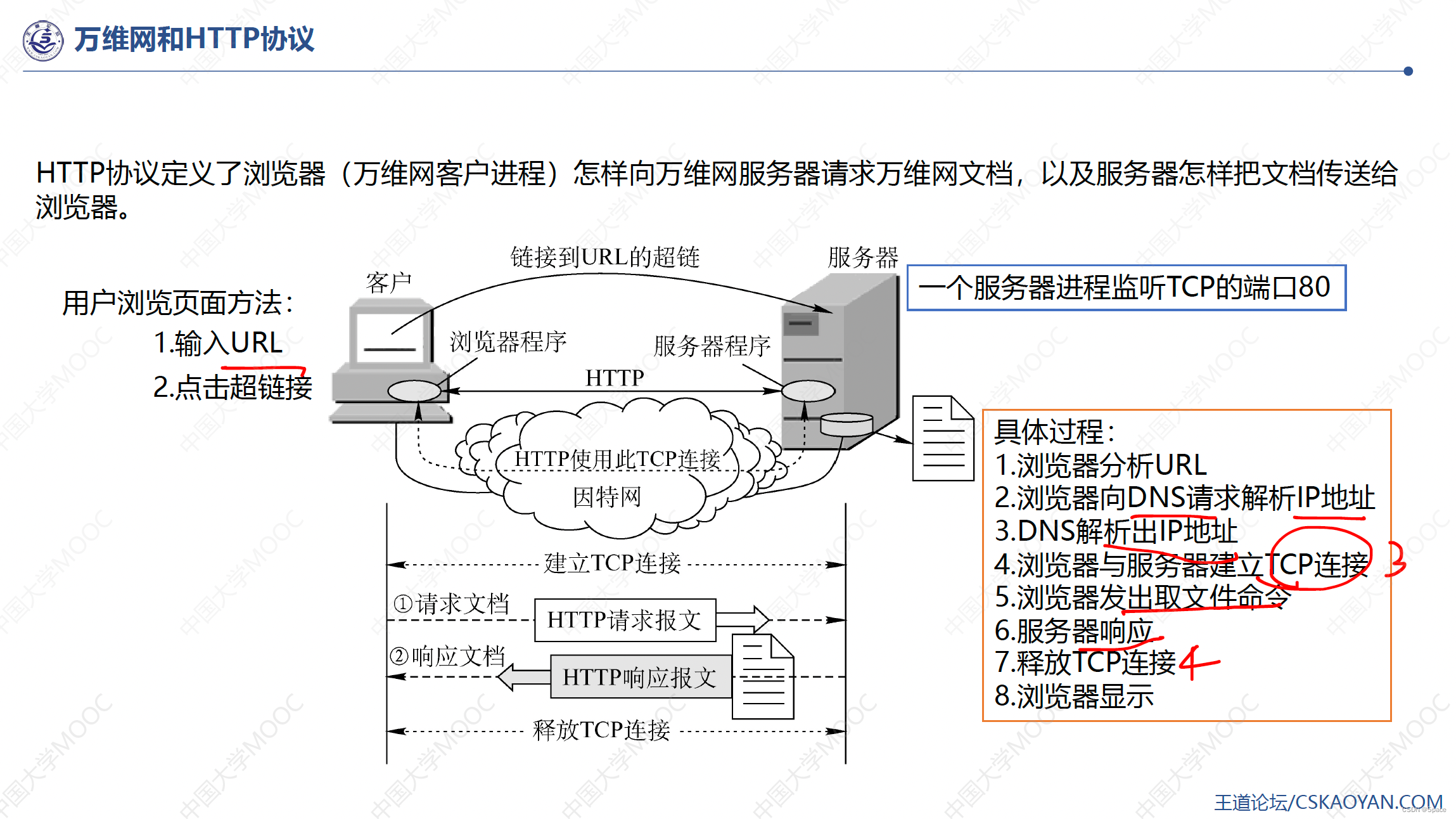
- 图1a:DrugBAN框架。药物分子使用GCN编码,蛋白质序列由1D CNN编码。药物编码表示的每一行是子结构的向量。蛋白质编码表示的每一行是子序列的向量。药物和蛋白质的表示被输入双线性注意力模块,用于学习它们的成对局部相互作用。联合表示 f \textbf{f} f由全连接解码器预测DTI概率 p p p。如果预测任务是跨域的,使用CDAN(Bilinear Attention Networks,NeurIPS,2018)模块在源域和目标域对齐学习的表示,再输入解码器预测概率。
- 图1b:双线性注意力网络。 H d \textbf{H}_{d} Hd和 H p \textbf{H}_{p} Hp是药物和蛋白质的表示。在step1中,bilinear attention map矩阵 I \textbf{I} I由变换矩阵 U \textbf{U} U和 V \textbf{V} V建立低秩相互作用模型,测量子结构相互作用的强度。然后利用 I \textbf{I} I通过共享变换矩阵 U \textbf{U} U和 V \textbf{V} V的双线性池化生成step2中的联合表示 f \textbf{f} f。
- 图1c:CDAN是一种域自适应技术,用于减少不同数据分布之间的领域偏移。使用CDAN将源域和目标域的联合表示 f \textbf{f} f和softmax logits g \textbf{g} g嵌入到判别器的联合条件表示中,这是一个两层全连接网络,最大限度地减少了域分类错误,以区分目标域和源域。
双线性注意力网络
BAN最初用于视觉问答VQA。VQA的任务是给定一个图像和相关的自然语言问题,系统给出与图像和问题相关的自然语言回答。VQA问题可以看作是一个多模态学习任务,与DTI相似。双线性注意力网络(BAN)考虑每对多模态输入通道,即"图像局部区域-提问句子的局部词语pair",以学习交互表示。在DTI中就是学习药物中的原子和蛋白质子结构pair之间的交互表示。
得到蛋白质表达 H p = { h p 1 , . . . , h p M } \textbf{H}_{p}=\left\{h_{p}^{1},...,h_{p}^{M}\right\} Hp={hp1,...,hpM}和药物表达 H d = { h d 1 , . . . , h d N } \textbf{H}_{d}=\left\{h_{d}^{1},...,h_{d}^{N}\right\} Hd={hd1,...,hdN},其中 M M M和 N N N分别表示蛋白质的子结构数量和药物的原子数量。双线性交互映射可以获得成对交互 I ∈ R N × M \textbf{I}\in R^{N\times M} I∈RN×M。在图1b中, U ∈ R D d × K \textbf{U}\in R^{D_{d}\times K} U∈RDd×K和 V ∈ R D p × K \textbf{V}\in R^{D_{p}\times K} V∈RDp×K表示药物和蛋白质的可学习权重矩阵。 q ∈ R K q\in R^{K} q∈RK是可学习的权重向量。SumPool是一个1D无重叠的sum pooling操作。
在学习时,DTI为二分类任务,设样本pair i i i的标签为 y i y_{i} yi,损失函数为: L = − ∑ i ( y i l o g ( p i ) + ( 1 − y i ) l o g ( 1 − p i ) ) L=-\sum_{i}(y_{i}log(p_{i})+(1-y_{i})log(1-p_{i})) L=−i∑(yilog(pi)+(1−yi)log(1−pi))
领域自适应
cross-domain自适应是为了更好的泛化。机器学习模型往往会在属于同一个分布的相似数据上得到很好的效果,但是在与训练集不同的分数数据上会表现得不太好。作者使用了CDAN来使得模型可以在没有标签的目标域数据上表现良好。
CDAN包含3个组件:特征提取 F ( ⋅ ) F(\cdot) F(⋅),解码器 G ( ⋅ ) G(\cdot) G(⋅),领域判别器 D ( ⋅ ) D(\cdot) D(⋅),使用 F ( ⋅ ) F(\cdot) F(⋅)生成输入数据的联合表达,即 f s = F ( x s ) f^{s}=F(x^{s}) fs=F(xs)和 f t = F ( x t ) f^{t}=F(x^{t}) ft=F(xt),然后使用解码器 G ( ⋅ ) G(\cdot) G(⋅)得到分类预测结果 g s = G ( f s ) ∈ R 2 g^{s}=G(f^{s})\in R^{2} gs=G(fs)∈R2和 g t = G ( f t ) ∈ R 2 g^{t}=G(f^{t})\in R^{2} gt=G(ft)∈R2,然后由多线性映射将 f f f和 g g g映射为一个联合表示 h h h。
判别器 D ( ⋅ ) D(\cdot) D(⋅)用于区分联合条件表示属于源域还是目标域。通过判别器的学习,可以使得解码器能够在无标签领域上进行泛化预测。
可解释性可视化

- 图2a:co-crystalized配体的可解释性。每个panel的左侧显示了配体的2D结构,其中突出显示的原子(橙色)被预测为有助于蛋白质结合。所有结构使用RDKit可视化。每个panel的右侧提供了配体对应的"配体-蛋白质"相互作用。
- 图2b:结合口袋上的可解释性。配体-蛋白质结合口袋的3D呈现,突出显示了围绕相应配体(青色)的正确预测的氨基酸残基(橙色)。剩余的氨基酸残基、二级结构元件和表面图用灰色表示。所有配体-蛋白质相互作用图和x射线结构的3D表示都使用Molecular Operating Environment(MOE)软件进行可视化。









![[软件工程导论(第六版)]第9章 面向对象方法学引论(课后习题详解)](https://img-blog.csdnimg.cn/24130b2be8bc4504839a8311d7409838.png)
![[软件工程导论(第六版)]第8章 维护(复习笔记)](https://img-blog.csdnimg.cn/a01f9d6c37aa473b9a57605a33c02a8a.png)