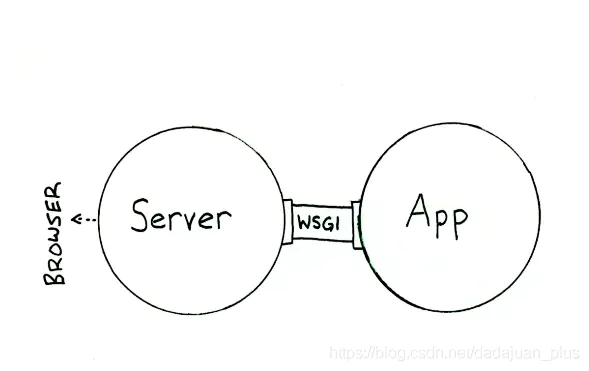
背景概括:
kaggle最近举办了一场医学乳腺癌检测的比赛(图像分类)
比赛官网地址
给的数据是dcm的专业的医学格式,自己通过DICOM库转为png后,发现该图像胸部不同的患者乳腺大小不一,简言之乳腺的CT有效图在转换成的png里面只占据了一小部分,剩下的绝大部分都是使用黑边进行了填充,于是考虑使用yolo获取图像的ROI,然后对图像进行切割来提升图像的信息密度,于是开始了踩坑之旅
(ROI:region of interest)
- 如图所示为转换为的PNG图片的例子

1获取全部转换后图像的ROI坐标信息
yolo采用的是kaggle开源的权重kaggle discussion链接地址
对图像进行目标检测之后,发现有一部分并不能检测出来存在乳腺,可以使用glob函数查看具体的没有检测出来的图片,后续单独存储一下用个低阈值的yolo做推断或者是使用opencv进行切割ROI
第一次YOLO使用的推断的阈值为0.5,随便查看了几个切割出来的txt文件发现还比较正常,只有一行,如下图所示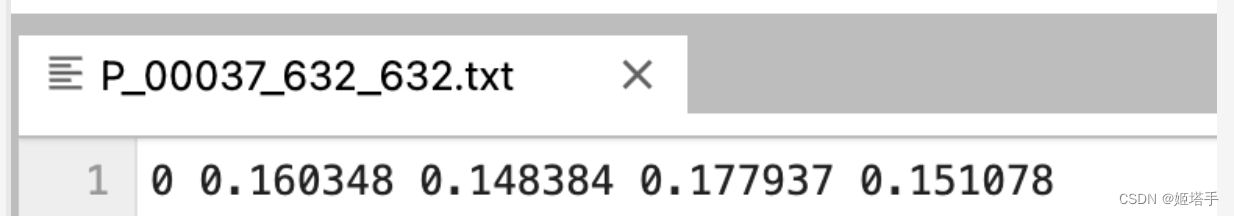
yolo检测出来的txt文件中每一行的含义分别为
label x y w h
坑一:
对每个txt文件进行读取,然后索引1,2,3,4分别获取x,y,w,h,的数值用于在原图中进行切割
def resize_to_square(label_path):
with open(label_path, "r") as f: # 打开文件
data_lxywh = f.read() .split(' ') # 读取文件
x = eval(data_lxywh[1])
y = eval(data_lxywh[2])
w = eval(data_lxywh[3])
h = eval(data_lxywh[4])
l1 = [x,y,w,h]
return l1
但是发现总共全量50000张图片每次进行到40000多张的时候总是会报错,一个隐晦的错误,搜了半天也没找到,起初是排查内存问题,硬盘问题,排查了好久,才发现是因为有的图片是检测出来了两个坐标框,坐标框凭借眼睛看是两行,但实际存储的是(以下图为例)第一行的最后一个数+/n0 (0是第二行的label),也就是说两行的时候读取txt的第五个元素实际上是第一行的最后一个元素加换行符加第二行的第一个元素
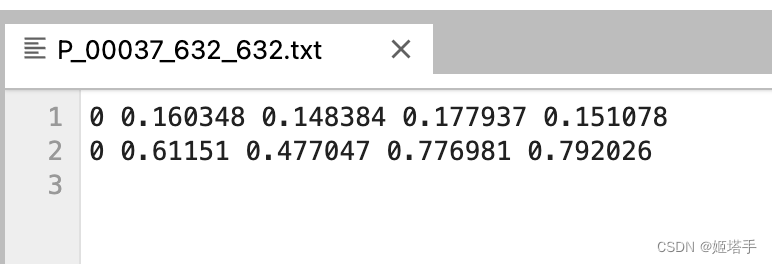
于是对txt文件读取的函数改成了这样
def resize_to_square(label_path):
with open(label_path, "r") as f: # 打开文件
data_lxywh = f.read() .split(' ') # 读取文件
x = eval(data_lxywh[1])
y = eval(data_lxywh[2])
w = eval(data_lxywh[3])
if data_lxywh[4]==data_lxywh[-1]:#判断总共有几行
h = eval(data_lxywh[4])
else:
h = eval(data_lxywh[4][:-2])## 切除换行符与多余的label
l1 = [x,y,w,h]
return l1
本以为这样就万事大吉了
踩坑二
后来处理扩增的正样本数据,发现切割出来的图片有好多杂质(源文件转换为png后,除了图片中心的核心的乳腺之外,边角部分会出现部分零散字母)
原图如下所示

部分切割出来的ROI杂质如下所示
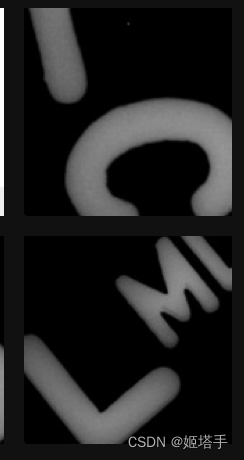
然后就想办法消除杂质,首先是考虑将原图切割一下边框,大体估计了下长宽切掉300像素就差不多,后来发现很难清除干净,该方案放弃
于是考虑从txt文件中下手,yolo获取的txt文件都是相对坐标尺度,乳腺数据应当宽高是最大的,问题就变成了挑选出最大的框,经过分析发现,yolo切割出来的txt文件中,是按照目标大小进行排序的,也就是小目标在上,大目标在下,而且本次乳腺都是大目标
于是进行了更改变成了如下所示(读取最后一行的用于ROI切割)
def resize_to_square(label_path):
with open(label_path, "r") as f: # 打开文件
data_lxywh = f.read().split(' ') # 读取文件
x = eval(data_lxywh[-4])
y = eval(data_lxywh[-3])
w = eval(data_lxywh[-2])
h = eval(data_lxywh[-1][:-1])
l1 = [x,y,w,h]
return l1
对坐标框坐标的处理与选择算是到此结束
有关图像转换,不变形切割,使用DALI加速,训练推断等等代码,比赛结束后更新