 原文标题:SMILES-BERT: Large Scale Unsupervised Pre-Training for Molecular Property Prediction
原文标题:SMILES-BERT: Large Scale Unsupervised Pre-Training for Molecular Property Prediction
链接:https://doi.org/10.1145/3307339.3342186
一、问题提出
分子指纹:可用于分子性质分类、回归或生成新分子等各种应用。传统的分子指纹不是计算基本性质,而是提供分子结构的特定部分的描述。然而,传统的分子指纹需要密集的手工特征工程和强大的领域知识。此外,这种指纹具有很强的任务依赖性,对于其他属性预测任务来说还不够通用。
SMILES:支持基于RNN的方法在多个gpu和多个设备上进行并行训练并非易事,它需要不同的训练技巧,如梯度裁剪和早期停止,以确保模型收敛;
Graph:基于gcns的方法通常计算复杂度较高,限制了探索更复杂的分子性质预测方法。(附加:深度不能太深)
现有过程发现:用筛选实验获得如此规模的分子性质的成本是异常高的。无法用大量的数据来实现对数据分布的完全拟合。
二、模型方法

希望利用无限的非标记数据中的基本信息来建立一个强大的半监督模型,以提高在有限的标记数据下的预测性能。其次,模型在训练阶段是有效的:1)支持并行训练2)用于预训练的模型全部参加调优阶段,没有像Seq3seq指纹的解码器那样的脚手架部分。
1、transformer block

2、Pre-training as Masked SMILES Recovery
与自然语言建模不同的是,SMILES没有连续的关系,the consecutive sentences classification无法使用。masked language learning仍然有希望用未标记的SMILES对模型进行预训练,我们将该任务命名为the task Masked SMILES Recovery。
3、Fine-tuning for Molecular Property Prediction
在预训练期间,用token <GO>填充每个输入SMILES。在微调阶段,使用令牌对应的模型输出进行分子性质预测。
4、Model Structure
前注意层和后注意层完全连接层将输入特征嵌入到1024大小的特征空间中。对于自我注意块,SMILES-BERT使用了一个head=4的多头注意机制。因为SMILES比自然语言序列相对简单。此外,smile的词汇量远远少于自然语言的词汇量。尝试了BERT的基础结构设置对分子性质的预测,并没有提供明显的改善。然后将SMILES-BERT保持在当前的设置中,因为在实践中对模型的计算和内存需求更少更好。
使用FairSeq进行代码编写。
屏蔽token的最小数量被设置为1。因此,每个输入SMILES至少包含一个掩码令牌。通过对这些数据集的训练,所提出的SMILES-BERT的泛化能力得到了增强。
随机保留10000个样本进行验证,另外10000个样本进行评估。训练集最终为18,671,355
Adam,前4000个step中引入了warmup(lr从10−9增加到10−4),如果没有warmup,即使经过长时间的训练,模型也不会收敛。warmup结束后,学习速率从10−4开始,采用反平方根更新策略。adam 中beta为(0.9,0.999),权值衰减为0.1。batch_size设为256,dropout为0.1。训10个epoch,ZINC验证数据集上的准确率为82.85%,这意味着从未掩码部分获得的信息可以准确地恢复82.85%的掩码SMILES。
预训练性能:

下游微调:尝试了10−5、10−6、10−7等学习率,都能得到很好的预测结果
使用50个epoch对模型进行微调,并选择验证数据上的最佳模型进行最终评估
使用三个数据集,LogP数据集,PM2数据集和PCBA-686978数据集


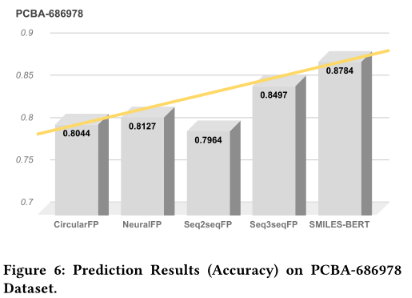
19年文章,目前也有3年(近4年了)。文章现在读起来质量偏低,内容也少。但证明了一件事,bert用于SMILES预训练后下游微调是可行。(虽然不能以现在的视角看待过去技术,但是要是放在现在......)











![已解决OSError: [Errno 22] Invalid argument](https://img-blog.csdnimg.cn/2baa3faf285449e1adb4583b8335b3b0.png)
![UnityVR一体机报错:GL_OUT_OF_MEMORY,[EGL] Unable to acquire context](https://img-blog.csdnimg.cn/4a15bed720784a51bc6d69aa41689eab.png)
