1,首先可以识别在KO前后的motif——》由CNN模型做出识别,看看这个有没有什么灵感
2,ZNF143等都可以使用来识别

3,暂时只使用单个peak文件,后期可以使用ENCODE中所有的对应的TF的peak文件

===========
1,文件解压之后:共53583个peak
narrowpeak的格式是:


参考:
https://mp.weixin.qq.com/s/Zz0DvH2-kgoU7xexfSZiQg
事实上,我可以使用ATAC-seq来作为对照,获取开放以及封闭区域的peak分别来训练CNN
当然,实际上是能够用各种组学数据来进一步缩短范围来训练的(实际上被排除掉的peak甚至可以作为负样本)
——》甚至是可以用在LL那篇文章中,看在NCR突变前后
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE261213(但是只有bw,当然可以直接由上游数据获得)


首先这个peak大致在100 bp左右

然后可以获取hg19的参考基因组


原先的peak有53583行
但是在中心flank之后都会丢失掉一些数据,可能是在chr特殊区域或者是末端


应该抽取出fa文件的第2行作为字符串存入列表中
第一性原理:对团块做个分类器(或者是聚类)
分类的解释性:——》从结果上去看有没有什么灵感
是交叉的
还是团块:clique
——》再分别对这些团块蛋白做个motif机制(有motif的)
2,整理输入数据的x:(x,y)中的x

其中fit_transform 方法是依据所有因子的字母顺序进行标记,而不是依据 Unicode 码。具体来说,它会将输入的类别按字母顺序排序,然后依次分配整数标签。
from sklearn.preprocessing import LabelEncoder, OneHotEncoder #导入 LabelEncoder 和 OneHotEncoder,用于将序列编码为整数和 one-hot 编码
# 创建一个 LabelEncoder 实例,用于将碱基序列编码为整数
integer_encoder = LabelEncoder()
integer_encoder.fit_transform(list('CTAG'))
#fit_transform 方法是依据所有因子的字母顺序进行标记,而不是依据 Unicode 码。具体来说,它会将输入的类别按字母顺序排序,然后依次分配整数标签。
#例如,对于 DNA 序列中的碱基 'A', 'C', 'G', 'T',LabelEncoder 会按字母顺序将它们编码为 0, 1, 2, 3。

所以fit_transform正好对sequences中所有序列之和的factor的水平levels(其实就是4种),按照字母顺序(其实就是unicode、ASCII)进行编码0123,不是按照出现前后顺序
参考:
https://scikit-learn.org/1.4/modules/generated/sklearn.preprocessing.LabelEncoder.html#sklearn.preprocessing.LabelEncoder.fit_transform
我们随便以1条序列作为例子:

以最后一条序列作为例子

总之对于sequences列表中的每一个peak sequence,我们已经获取了其对应的整数编码数组(序列)

然后实际上reshape是将原来的序列字符串的编码之后的一维数组,转换为2维的列向量
from sklearn.preprocessing import LabelEncoder, OneHotEncoder #导入 LabelEncoder 和 OneHotEncoder,用于将序列编码为整数和 one-hot 编码
integer_encoder = LabelEncoder()
# 创建一个 OneHotEncoder 实例,用于将整数编码转换为 one-hot 编码
one_hot_encoder = OneHotEncoder(categories='auto')
input_features = [] #初始化一个空列表,用于存储每个序列的 one-hot 编码
integer_encoded =integer_encoder.fit_transform(list('CGGGGACACGGCCCCGCCATGCGGGGGCGCTCAGGCGCGGGGCGCCGCGC'))
np.array(integer_encoded)
integer_encoded = np.array(integer_encoded).reshape(-1, 1)




然后进行独热编码:

https://scikit-learn.org/1.4/modules/generated/sklearn.preprocessing.OneHotEncoder.html
我们其实可以看到:
整数编码其实就是独热编码那个不为0而为1的位置编号
但是独热编码结果如果不转换为数组其实就很难理解
from sklearn.preprocessing import LabelEncoder, OneHotEncoder #导入 LabelEncoder 和 OneHotEncoder,用于将序列编码为整数和 one-hot 编码
integer_encoder = LabelEncoder()
# 创建一个 OneHotEncoder 实例,用于将整数编码转换为 one-hot 编码
one_hot_encoder = OneHotEncoder(categories='auto')
input_features = [] #初始化一个空列表,用于存储每个序列的 one-hot 编码
integer_encoded =integer_encoder.fit_transform(list('CGGGGACACGGCCCCGCCATGCGGGGGCGCTCAGGCGCGGGGCGCCGCGC'))
np.array(integer_encoded)
integer_encoded = np.array(integer_encoded).reshape(-1, 1)
print(integer_encoded)
one_hot_encoded = one_hot_encoder.fit_transform(integer_encoded)
one_hot_encoded
print(one_hot_encoded)


只有在toarray转换为数组之后,展现的形式我们才熟悉:



from sklearn.preprocessing import LabelEncoder, OneHotEncoder
import numpy as np
# 示例序列
sequence = 'GCTAG'
# 创建 LabelEncoder 实例
integer_encoder = LabelEncoder()
# 将序列中的每个碱基转换为整数编码
integer_encoded = integer_encoder.fit_transform(list(sequence))
# 将整数编码转换为列向量
integer_encoded = np.array(integer_encoded).reshape(-1, 1)
# 打印整数编码结果
print("整数编码结果:\n", integer_encoded)
# 创建 OneHotEncoder 实例
one_hot_encoder = OneHotEncoder(categories='auto')
# 将整数编码转换为独热编码
one_hot_encoded = one_hot_encoder.fit_transform(integer_encoded)
# 将独热编码转换为数组并打印
one_hot_encoded_array = one_hot_encoded.toarray()
print("独热编码结果:\n", one_hot_encoded_array)


所以input_features里面其实就相当于获取了很多sequence peak序列的独热编码的array,是高维array(tensor)
接下来就是对序列的拼接堆叠:也就是np.stack这一步
import numpy as np
# 示例独热编码数组
one_hot_encoded_1 = np.array([[0, 0, 1, 0],
[0, 1, 0, 0],
[0, 0, 0, 1],
[1, 0, 0, 0],
[0, 0, 1, 0]])
one_hot_encoded_2 = np.array([[1, 0, 0, 0],
[0, 1, 0, 0],
[0, 0, 1, 0],
[0, 0, 0, 1],
[1, 0, 0, 0]])
one_hot_encoded_3 = np.array([[0, 1, 0, 0],
[1, 0, 0, 0],
[0, 0, 1, 0],
[0, 0, 0, 1],
[0, 1, 0, 0]])
# 将这些独热编码数组放入一个列表
input_features = [one_hot_encoded_1, one_hot_encoded_2, one_hot_encoded_3]
# 使用 np.stack 将这些数组堆叠成一个三维数组
stacked_features = np.stack(input_features)
# 打印堆叠后的数组及其形状
print("堆叠后的数组:\n", stacked_features)
print("堆叠后的数组形状:", stacked_features.shape)




但是实际运行的时候报错了:
from sklearn.preprocessing import LabelEncoder, OneHotEncoder #导入 LabelEncoder 和 OneHotEncoder,用于将序列编码为整数和 one-hot 编码
# 创建一个 LabelEncoder 实例,用于将碱基序列编码为整数
integer_encoder = LabelEncoder()
# 创建一个 OneHotEncoder 实例,用于将整数编码转换为 one-hot 编码
one_hot_encoder = OneHotEncoder(categories='auto')
input_features = [] #初始化一个空列表,用于存储每个序列的 one-hot 编码
for sequence in sequences:
integer_encoded = integer_encoder.fit_transform(list(sequence))
#将序列中的每个碱基转换为整数编码,Fit label encoder and return encoded labels
#对每条序列sequence拆分为单碱基字符列表后,先fit,再transform,就是对ATCG进行数字编码,就是从0开始
integer_encoded = np.array(integer_encoded).reshape(-1, 1) #将整数编码转换为列向量,reshape(-1, 1)中的-1表示自动计算行数
one_hot_encoded = one_hot_encoder.fit_transform(integer_encoded) #将整数编码转换为 one-hot 编码
input_features.append(one_hot_encoded.toarray()) #将 one-hot 编码转换为数组并添加到 input_features 列表中
print(input_features)
print(input_features[0])
print(input_features[0][0])
print(input_features[0][0][0])
np.set_printoptions(threshold=40) #设置打印选项,限制输出的元素数量
input_features = np.stack(input_features) #将 input_features 列表转换为 numpy 数组
print("Example sequence\n-----------------------") #打印示例序列的标题
print('DNA Sequence #1:\n',sequences[0][:10],'...',sequences[0][-10:]) #打印第一个 DNA 序列的前 10 个和后 10 个碱基
print('One hot encoding of Sequence #1:\n',input_features[0]) #打印第一个 DNA 序列的 one-hot 编码

在stack的时候array维度不一致
要调试并找出哪些数组的形状不同,可以遍历 input_features 列表并打印每个数组的形状。
# 打印每个数组的形状
for i, feature in enumerate(input_features):
# enumerate返回index、value的元组
print(f"Array {i} shape: {feature.shape}")
# 检查是否所有数组形状一致
shapes = [feature.shape for feature in input_features]
if len(set(shapes)) != 1:
print("存在形状不一致的数组")
else:
print("所有数组形状一致")
# 如果所有数组形状一致,则进行堆叠
if len(set(shapes)) == 1:
input_features = np.stack(input_features)
print("堆叠后的数组形状:", input_features.shape)
else:
print("无法堆叠数组,因为它们的形状不一致")


其实按理来说,所有array的shape应该都是(50,4),因为有50bp长,每个bp都是一个长度为4的独热编码
问题:每个sequence只依据自身信息进行编码,是否有些序列50bp内只出现3字符,比如说某个序列只有ACG,那么独热编码的时候实际上class只有3类,所以只能3维的独热——》需要在全局统一一下


array是从0开始的,0-index,peak是1-index的

以array 53233为例,实际上就是peak 53234
果然检查了一下是只有3字母的:A/T/G

所以问题其实就出在了原本脚本中的auto这一个参数
one_hot_encoder = OneHotEncoder(categories='auto')
实际上参考scikit-learn中的
https://scikit-learn.org/1.4/modules/generated/sklearn.preprocessing.OneHotEncoder.html


所以应该指定category:
one_hot_encoder = OneHotEncoder(categories=[['A', 'C', 'G', 'T']])
当然,如果前面指定了category,后面其实就不用整数编码了,可以直接使用独热编码
from sklearn.preprocessing import LabelEncoder, OneHotEncoder #导入 LabelEncoder 和 OneHotEncoder,用于将序列编码为整数和 one-hot 编码
# 创建一个 OneHotEncoder 实例,用于将整数编码转换为 one-hot 编码
one_hot_encoder = OneHotEncoder(categories=[['A', 'C', 'G', 'T']])
input_features = [] #初始化一个空列表,用于存储每个序列的 one-hot 编码
for sequence in sequences:
# 将序列中的每个碱基转换为列向量
sequence_array = np.array(list(sequence)).reshape(-1, 1)
# 将字符序列转换为 one-hot 编码
one_hot_encoded = one_hot_encoder.fit_transform(sequence_array)
# 将 one-hot 编码转换为数组并添加到 input_features 列表中
input_features.append(one_hot_encoded.toarray())
for i, feature in enumerate(input_features):
# enumerate返回index、value的元组
print(f"Array {i} shape: {feature.shape},has value {feature}")
np.set_printoptions(threshold=40) #设置打印选项,限制输出的元素数量,表示在打印时如果数组超过40个元素,则只打印开头和结尾的部分
input_features = np.stack(input_features) #将 input_features 列表转换为 numpy 数组
print("Example sequence\n-----------------------") #打印示例序列的标题
print('DNA Sequence #1:\n',sequences[0][:10],'...',sequences[0][-10:]) #打印第一个 DNA 序列的前 10 个和后 10 个碱基
print('One hot encoding of Sequence #1:\n',input_features[0]) #打印第一个 DNA 序列的 one-hot 编码
那再测试一下:

同样,list获取字符串列表,np.array获取数组(一维)
reshape之后转换为二维向量

然后这个二维向量直接使用one-hot编码处理




在toarray之直观观察one-hot的稀疏矩阵,所以没有toarray之前其实是稀疏矩阵值为1的坐标标记
from sklearn.preprocessing import OneHotEncoder #导入OneHotEncoder即可,用于将序列编码为one-hot 编码;无需导入LabelEncoder,用于将序列编码为整数
# 创建一个 OneHotEncoder 实例,用于将整数编码转换为 one-hot 编码
one_hot_encoder = OneHotEncoder(categories=[['A', 'C', 'G', 'T']])
input_features = [] #初始化一个空列表,用于存储每个序列的 one-hot 编码
# 将序列中的每个碱基转换为列向量
sequence_array = np.array(list('CGGGGACACGGCCCCGCCATGCGGGGGCGCTCAGGCGCGGGGCGCCGCGC')).reshape(-1, 1)
print(list('CGGGGACACGGCCCCGCCATGCGGGGGCGCTCAGGCGCGGGGCGCCGCGC'))
print(np.array(list('CGGGGACACGGCCCCGCCATGCGGGGGCGCTCAGGCGCGGGGCGCCGCGC')))
print(sequence_array)
# 将字符序列转换为 one-hot 编码
one_hot_encoded = one_hot_encoder.fit_transform(sequence_array)
print(one_hot_encoded)
print(one_hot_encoded.toarray())
# 将 one-hot 编码转换为数组并添加到 input_features 列表中
input_features.append(one_hot_encoded.toarray())
for i, feature in enumerate(input_features):
# enumerate返回index、value的元组
print(f"Array {i} shape: {feature.shape},has value {feature}")
np.set_printoptions(threshold=40) #设置打印选项,限制输出的元素数量,表示在打印时如果数组超过40个元素,则只打印开头和结尾的部分
input_features = np.stack(input_features) #将 input_features 列表转换为 numpy 数组
print("Example sequence\n-----------------------") #打印示例序列的标题
print('DNA Sequence #1:\n',sequences[0][:10],'...',sequences[0][-10:]) #打印第一个 DNA 序列的前 10 个和后 10 个碱基
print('One hot encoding of Sequence #1:\n',input_features[0]) #打印第一个 DNA 序列的 one-hot 编码
验证之后没有问题:

3,整理输入数据中的y:(x,y)中的y

首先原始数据中给出的label标签其实正样本以及负样本都包括的,所以对应的标签也就有1以及0;
然后前面我所处理的数据中其实给出的基本上都是CTCF chip-nexus的peak的序列信息,可以说都是正样本positive sample。
但是实际上我是需要提供对应的负样本的,所以如何产生以及评估负样本negative sample呢?
目前有2种策略:
①1种就是将上面的CTCF chip实验中获取的peak的序列信息,再使用ATAC-seq的数据筛选一遍样本,然后将在开放区域内的peak作为正样本,反之则作为负样本;
当然事实上我们前面获取的数据中其实都没有input的数据
②第2种方法就是随机生成序列,或者是随机从CTCF peak对应的周边序列的fa文件中抽取长度为50的窗口的序列
——》现在采取方法①:
但是我们手头上只有那个数据集ATAC的bw文件,如果还是走前面bed提取序列的方法的话,我们需要将bw文件转换为bed文件,

发现peak太多了,而且peak大小其实数量级和CTCF peak差不多
在用bedtools取交集之后,其实数量级没有变化,还是10M级的

这里其实还需要注意一下narrowpeak的index是不是0-index的
因为ATAC获取的交集peak是0-index的。
——》其实我们的条件更严苛,因为我使用的是CTCF peak中心的50bp窗口,拿这个去筛选开放区域交集的窗口,但是实际上并不一定,
比如说某个CTCF的peak和开放性区域有交集,但是实际上的CTCF中心的50bp(看前面分布,实际上50bp的窗口是占少数的peak,所以说大多数CTCF 50bp都能捕获到peak的区域,至少大多数是被包含在peak内的)不一定和开放性区域有交集。
总之2种策略:
①用原始的narrowpeak去和ATAC开放性区域筛选交集,有交集的区域,重新计算中心,再flank上下游25bp,然后作为正样本,其余narrowpeak作为负样本
②直接计算narrowpeak中心区域flank的50bp窗口,再看这个窗口和ATAC开放性区域有交集的作为正样本,其余作为负样本
下面以策略②为准:
但是实际处理之后发现样本基本上都在开放性区域内

=======》
总之我现在的想法就是:
只使用现有的CTCF peak样本数据,从中使用其他组学数据进一步区分正样本以及负样本,
前面使用的是开放性区域,但是发现基本上都是重合的,
所以还是使用其他的组学数据:
比如说使用Cohesin chip-seq数据,训练Cohesin和CTCF peak重合下(共定位下)的CTCF motif,然后不重合的作为负样本。
还是参考:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE236632


还是上面的那样:
①获取CTCF和Cohesinn共定位(有overlap)的CTCF peak,然后取这个peak的中心,做50bp窗口,其余没有overlap的就作为负样本
②


单看区域的数目有40965个正样本,但是有些这些区域可能是对CTCF的1个peak的多个划分交集


解释略:

总之就是使用大概4:1的正:负样本去训练
然后如果我只是使用这中心的50bp的窗口去取交集的话,实际上也差不多,
大概在40825的正样本,和前面只看中心的40921的正样本差不了多少

现在可以从头构建(x,y)数据:
对于CTCF的原始peak,和cohesin共定位的(用bedtools识别出来),可以在该peak的bed文件新增的label一列中记录1,没有bedtools intersect识别出来的,就标记为0;
然后对于CTCF在处理之后的peak的bed数据,只保留chr+start+end+label,
然后再和之前一样使用取start、end中心再上下延伸50bp窗口。
#!/bin/bash
# 定义文件路径
CTCF_FILE="/mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/GSM7566824_WT_ChIP-nexus_CTCF_peaks.narrowPeak"
RAD21_FILE="/mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/GSM7566825_WT_ChIP-nexus_RAD21_peaks.narrowPeak"
OUTPUT_FILE="/mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/GSM7566824_WT_ChIP-nexus_CTCF_peaks_with_labels.narrowPeak"
# 使用 bedtools intersect 找出共定位的峰值,并将结果保存到临时文件
bedtools intersect -a "$CTCF_FILE" -b "$RAD21_FILE" -wa | cut -f 1-3 | sort | uniq > intersected_peaks.tmp
# 使用 awk 处理 CTCF 文件,新增标签列
awk 'BEGIN {FS=OFS="\t"}
NR==FNR {intersect[$1,$2,$3]=1; next}
{label = ($1,$2,$3) in intersect ? 1 : 0; print $0, label}' intersected_peaks.tmp "$CTCF_FILE" > "$OUTPUT_FILE"
# 删除临时文件
rm intersected_peaks.tmp
echo "处理完成,结果已保存到 $OUTPUT_FILE"
使用 bedtools intersect 找出 CTCF 和 Rad21 共定位的峰值,并将结果保存到临时文件 intersected_peaks.tmp。
使用 awk 处理 CTCF 文件,检查每个峰值是否在临时文件中出现。如果出现,则新增标签列为 1,否则为 0。
将处理后的结果保存到新的文件 GSM7566824_WT_ChIP-nexus_CTCF_peaks_with_labels.narrowPeak。
删除临时文件。


获得的新文件:

行数peak不变,我们只看前面3行+最后1行。
因为我知道前面粗略取50bp窗口的话有些peak取不到,所以这次我会一起保留label列
#这是对原始的没有label的文件
awk '{mid=int(($2+$3)/2); start=mid-25; end=mid+25; print $1"\t"start"\t"end}' GSM7566824_WT_ChIP-nexus_CTCF_peaks.narrowPeak > GSM7566824_WT_ChIP-nexus_CTCF_peaks_50.bed
bedtools getfasta -fi /mnt/sdb/zht/ref_genome/ucsc_hg19/hg19_new.fa -bed /mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/GSM7566824_WT_ChIP-nexus_CTCF_peaks_50.bed -fo /mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/GSM7566824_WT_ChIP-nexus_CTCF_peaks_50.fa
#这是对原始的没有label的文件
awk '{mid=int(($2+$3)/2); start=mid-25; end=mid+25; print $1"\t"start"\t"end"\t"$11}' /mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/GSM7566824_WT_ChIP-nexus_CTCF_peaks_with_labels.narrowPeak > GSM7566824_WT_ChIP-nexus_CTCF_peaks_with_labels_50.bed
bedtools getfasta -fi /mnt/sdb/zht/ref_genome/ucsc_hg19/hg19_new.fa -bed /mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/GSM7566824_WT_ChIP-nexus_CTCF_peaks_50.bed -fo /mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/GSM7566824_WT_ChIP-nexus_CTCF_peaks_50.fa
正好是第11列

正好发现了原始数据并没有处理好数据,简直是mess:
因为我的参考fa文件是没有异常染色体的,我一般都会在处理前将这些异常染色体除去

修改之后为:
awk '{mid=int(($2+$3)/2); start=mid-25; end=mid+25; print $1"\t"start"\t"end"\t"$11}' /mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/GSM7566824_WT_ChIP-nexus_CTCF_peaks_with_labels.narrowPeak | awk 'BEGIN {FS=OFS="\t"} $1 ~ /^chr([1-9]$|1[0-9]$|2[0-2]$|X$|Y$)/' > GSM7566824_WT_ChIP-nexus_CTCF_peaks_with_labels_50.bed
bedtools getfasta -fi /mnt/sdb/zht/ref_genome/ucsc_hg19/hg19_new.fa -bed GSM7566824_WT_ChIP-nexus_CTCF_peaks_with_labels_50.bed -fo /mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/GSM7566824_WT_ChIP-nexus_CTCF_peaks_with_labels_50.fa

这会发现才应该是没有缺失的,所以理论上每个peak中获取中心50bp窗口,前面缺失了一部分不是因为有些peak短于50bp且在末端chr上,而是因为index中有异常chr在

所以正负样本混合之后是53234个peak。
总之目前统一一下我们的数据:
awk 'BEGIN {positive=0; negative=0} {if ($4==1) positive++; else negative++} END {print "Positive:", positive; print "Negative:", negative}' /mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/GSM7566824_WT_ChIP-nexus_CTCF_peaks_with_labels_50.bed

Positive: 40657
Negative: 12577
同样获得的fa文件作为数据集,和前面一样处理:
import numpy as np #导入 NumPy 库,并将其命名为 np。NumPy 是一个用于科学计算的库,提供了支持多维数组和矩阵运算的功能
import pandas as pd #导入 Pandas 库,并将其命名为 pd。Pandas 是一个用于数据操作和分析的库,提供了高效的数据结构和数据分析工具
import matplotlib.pyplot as plt #导入 Matplotlib 库中的 pyplot 模块,并将其命名为 plt。Matplotlib 是一个用于数据可视化的库,pyplot 模块提供了类似于 MATLAB 的绘图 API
import requests #导入 Requests 库。Requests 是一个用于发送 HTTP 请求的库,简化了与 Web 服务的交互。
sequences = []
with open('/mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/GSM7566824_WT_ChIP-nexus_CTCF_peaks_with_labels_50.fa', 'r') as file:
sequence = ''
for line in file:
if line.startswith('>'):
continue # 跳过以'>'开头的行
sequence = line.strip().upper() # 去除两端空白并转换为大写
sequences.append(sequence)
sequences = list(filter(None, sequences)) # 过滤掉空序列,最外层还是要套一层list,否则不是list类型,是map类型
# 先看看数据集长什么样
pd.DataFrame(sequences, index=np.arange(1, len(sequences)+1),
columns=['Sequences']).head()
然后关键就是处理数据中的label,
# 提取最后一列并保存到 labels.txt 文件中
# cut -f 4 GSM7566824_WT_ChIP-nexus_CTCF_peaks_with_labels_50.bed > labels.txt
# 读取 labels.txt 文件
with open('/mnt/sdb/zht/project/uniprot/dl_model/Deep_Learning_in_Genomics_Primer/labels.txt', 'r') as file:
labels = file.read().splitlines()
#读取文件的所有内容,并按行分割成一个列表。splitlines() 方法会移除每行末尾的换行符
# 移除空行
labels = list(filter(None, labels))
# 创建一个 OneHotEncoder 实例
one_hot_encoder = OneHotEncoder(categories='auto')
# 将标签转换为 NumPy 数组并进行独热编码
labels = np.array(labels).reshape(-1, 1)
input_labels = one_hot_encoder.fit_transform(labels).toarray()
print('Labels:\n', labels)
print('One-hot encoded labels:\n', input_labels)

对应上:


然后是过滤空行:







可以看到,这里其实就是一个array,并不是前面sequences中对于sequence的循环。所以这里的category指定是auto,而不是手动选择0-1。



其实我们可以发现,最终的label中编码,其实就是针对levels水平而言的,然后因子的水平levels之间需要注意顺序,一般的顺序就是按照ASCII码,所以是0-1,当然这里也可以在前面指定类别是:
one_hot_encoder = OneHotEncoder(categories=[['A', 'C', 'G', 'T']])
one_hot_encoder = OneHotEncoder(categories=[['0', '1']])

这个时候我们注意到,确实在指定了category为0-1的独热编码之后,0就被指定为二维向量第一位的独热编码了;1被指定为二维向量第2位的独热编码。
哪怕后面array给出1-0,实际上也是看水平的顺序的。
然后在处理好了训练集以及测试集之后,再对其进行划分

首先这里的*array允许多个输入,
test/train_size是互补的,然后random_state实际上就是随机数的种子

from sklearn.model_selection import train_test_split #将数据集拆分为训练集和测试集
train_features, test_features, train_labels, test_labels = train_test_split(
input_features, input_labels, test_size=0.25, random_state=2025)
#(features,labels)实际上就是(x,y)
print ("x_train shape: ", train_features.shape)
#print(train_features)
print ("y_train shape: ", train_labels.shape)
#print(train_labels)
print("x_test shape: ", test_features.shape)
#print(test_features)
print ("y_test shape: ", test_labels.shape)
#

几维度的tensor,多少样本,多少feature(sequence字符),每个feature的表征。
53234——》可以发现即使我当初划分训练集与测试集的比例是4:1,也就是给出0.25的比例,虽然不能整除,但是实际上train和test还是int划分出来了。
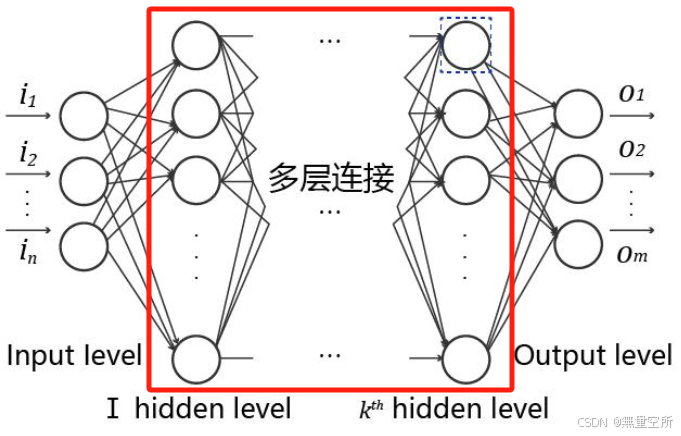
4,选择网络架构并进行训练:如果是使用tensorflow框架
# 导入所需的层和模型
# 导入了TensorFlow Keras库中的一些常用层和模型类:
# Conv1D:一维卷积层,用于处理序列数据。
# Dense:全连接层。
# MaxPooling1D:一维最大池化层。
# Flatten:将多维输入展平为一维。
# Sequential:线性堆叠模型,每一层的输出自动成为下一层的输入
from tensorflow.keras.layers import Conv1D, Dense, MaxPooling1D, Flatten
from tensorflow.keras.models import Sequential
# 初始化Sequential模型,这是一种线性堆叠的模型,每一层的输出自动成为下一层的输入
# 初始化了一个Sequential模型对象,表示模型的层是按顺序堆叠的
model = Sequential()
# 添加一维卷积层Conv1D
# filters=32表示有32个卷积核,积核的数量决定了输出特征图的深度。32个卷积核可以提取32种不同的特征。kernel_size=12表示每个卷积核的大小是12,卷积核的大小决定了每次卷积操作覆盖的输入区域。12个碱基的窗口大小可以捕捉到局部的模式。
# input_shape=(train_features.shape[1], 4)指定输入数据的形状。每个输入序列是50个碱基,4维度的one-hot编码(A, C, G, T)
# 其中train_features.shape[1]是特征的数量,4是特征的维度
model.add(Conv1D(filters=32, kernel_size=12,
input_shape=(train_features.shape[1], 4)))
# 添加一维最大池化层MaxPooling1D
# pool_size=4表示池化窗口的大小是4,即每次取4个连续的数据进行池化操作。池化操作通过取窗口内的最大值来减少特征图的尺寸,从而减少计算量和防止过拟合。
model.add(MaxPooling1D(pool_size=4))
# 添加Flatten层,将多维的输出展平成一维,以便输入到全连接层。卷积层和池化层的输出是多维的,需要展平成一维才能输入到全连接层。
model.add(Flatten())
# 添加全连接层Dense
# units=16表示该层有16个神经元,全连接层的神经元数量决定了该层的输出维度。16个神经元可以学习到16种不同的特征组合。activation='relu'表示使用ReLU激活函数,ReLU激活函数可以引入非线性,使模型能够学习到更复杂的模式。
model.add(Dense(16, activation='relu'))
# 添加输出层,也是一个全连接层
# units=2表示该层有2个神经元,输出层的神经元数量决定了模型的输出维度,2个神经元对应二分类问题的两个类别。activation='softmax'表示使用softmax激活函数,用于二分类问题,softmax激活函数将输出转换为概率分布,用于多分类或二分类问题。
model.add(Dense(2, activation='softmax'))
# 编译模型,配置其学习过程
# loss='binary_crossentropy'指定损失函数为二元交叉熵,适用于二分类问题
# optimizer='adam'指定优化器为Adam,这是一种自适应学习率的优化算法
# metrics=['binary_accuracy']指定评估模型性能的指标为二元准确率
model.compile(loss='binary_crossentropy', optimizer='adam',
metrics=['binary_accuracy'])
# 打印模型的概述,包括每层的输出形状和参数数量
model.summary()






模型框架如下:




然后是增加早停机制:
from tensorflow.keras.callbacks import EarlyStopping #Keras中的一个模块,包含了许多回调函数,用于在训练过程中执行特定的操作。EarlyStopping是一个回调函数,用于在监控的指标不再改善时提前停止训练,以防止过拟合
early_stop = EarlyStopping(monitor='val_loss',patience=10)
# monitor='val_loss':monitor:指定要监控的指标。在这里,'val_loss'表示监控验证集的损失。'val_loss':验证集的损失值。选择监控验证集的损失是因为它可以反映模型在未见过的数据上的表现,从而帮助防止过拟合。
# patience=10:patience:指定在监控的指标不再改善时,允许训练继续的轮数。在这里,10表示如果验证集的损失在连续2个训练轮次中没有改善,则停止训练。2:具体的耐心值,表示在验证集损失不再改善的情况下,允许训练继续的最大



import numpy as np
from tensorflow.keras.layers import Conv1D
from tensorflow.keras.models import Sequential
# 创建一个简单的序列数据
data = np.random.rand(10, 100, 4) # 10个样本,每个样本100个时间步,每个时间步4个特征
# 创建模型
model = Sequential()
model.add(Conv1D(filters=32, kernel_size=3, input_shape=(100, 4)))
# 查看模型结构
model.summary()


from tensorflow.keras.layers import Dense
from tensorflow.keras.models import Sequential
# 创建模型
model = Sequential()
model.add(Dense(16, activation='relu', input_shape=(10,)))
# 查看模型结构
model.summary()


import torch # torch:PyTorch 主库
import torch.nn as nn # torch.nn:包含神经网络模块和层
import torch.optim as optim # torch.optim:包含优化器模块
from torch.utils.data import DataLoader, TensorDataset # torch.utils.data:包含数据加载和处理模块
import numpy as np # numpy:用于科学计算的库
# train_features, train_labels, test_features, test_labels就是前面已经定义的(x_train, y_train, x_test, y_test)
# 将训练和测试数据转换为 PyTorch 张量,并指定数据类型为 float32
train_features_tensor = torch.tensor(train_features, dtype=torch.float32) #x_train
train_labels_tensor = torch.tensor(train_labels, dtype=torch.float32) #y_train
test_features_tensor = torch.tensor(test_features, dtype=torch.float32) #x_test
test_labels_tensor = torch.tensor(test_labels, dtype=torch.float32) #y_test

首先是将前面划分处理好的训练集以及测试集中的array数据转换为tensor数据

将numpy中的ndarray转换为pytorch支持的tensor数据类型函数,可以参考:
https://pytorch.org/docs/stable/generated/torch.tensor.html#torch-tensor

然后就是
# 创建数据加载器
train_dataset = TensorDataset(train_features_tensor, train_labels_tensor) #创建 TensorDataset,将特征和标签组合在一起,主要作用:将特征张量和标签张量组合成一个数据集对象
train_loader = DataLoader(train_dataset, batch_size=32, shuffle=True) #作用:将数据集分成小批量(batch),并支持随机打乱(shuffle)和多线程加载
test_dataset = TensorDataset(test_features_tensor, test_labels_tensor)
test_loader = DataLoader(test_dataset, batch_size=32, shuffle=False)
# 创建 DataLoader,用于批量加载数据:
# batch_size=32:每个批次包含 32 个样本。
# shuffle=True:在每个 epoch 开始时打乱训练数据,适用于train训练集,test测试集不需要打乱
主要涉及到两个函数:

前者组合feature以及label数据成为一个数据集,后者主要是用于batch获取
https://pytorch.org/docs/stable/data.html#torch.utils.data.TensorDataset

https://pytorch.org/docs/stable/data.html#torch.utils.data.DataLoader


主要关注的参数就是batch_size,以及shuffle
因为我的样本数据维度是:

train的dataset大概在4w左右,test的dataset大概在1w左右;
然后一个epoch,如果batch_size大概在32左右的话,那么大概会有1w(数量级)/30≈300个batch左右。
问题就在于这里的batch size,如果数据量不能被batch size整除,最后一个批次的数据可能会导致形状不匹配问题,也就是train以及test中最后一个batch的数据会不足。
为解决此问题,可以在PyTorch的DataLoader中设置drop_last=True,这样可以自动丢弃最后一个不足batch size的数据,确保每个epoch内数据的完整性和批处理的一致性。这是一个简单而有效的方法,尤其适用于大量数据的情况



可以看到,其实就是数据类型变化了而已,int变为float,整体是tensor
#示例
features = torch.tensor([[1, 2], [3, 4], [5, 6], [7, 8]],dtype=torch.float32)
labels = torch.tensor([0, 1, 0, 1],dtype=torch.float32)
print(features,labels)
dataset = TensorDataset(features, labels)
print(dataset)
loader = DataLoader(dataset, batch_size=2, shuffle=True)
print(loader)
for batch_features, batch_labels in loader:
print(batch_features, batch_labels)

然后就是合并tensor,再按照两个sample为一个batch进行划分。

确实最后一个batch需要注意一下,所以需要将batch size相关的参数修改一下。
修改之后为:
# 创建数据加载器
train_dataset = TensorDataset(train_features_tensor, train_labels_tensor) #创建 TensorDataset,将特征和标签组合在一起,主要作用:将特征张量和标签张量组合成一个数据集对象
train_loader = DataLoader(train_dataset, batch_size=32, shuffle=True,drop_last=True) #作用:将数据集分成小批量(batch),并支持随机打乱(shuffle)和多线程加载,batch_size如果无法整除整个数据集,可以丢弃最后一个batch
test_dataset = TensorDataset(test_features_tensor, test_labels_tensor)
test_loader = DataLoader(test_dataset, batch_size=32, shuffle=False,drop_last=True)
还是测验:

如果设置了drop_last之后:




效果非常差:

from tensorflow.keras.layers import Conv1D, Dense, MaxPooling1D, Flatten, Dropout
model = Sequential()
model.add(Conv1D(filters=32, kernel_size=12, input_shape=(train_features.shape[1], 4)))
model.add(MaxPooling1D(pool_size=4))
model.add(Dropout(0.5)) # 添加Dropout层,丢弃50%的神经元
model.add(Flatten())
model.add(Dense(16, activation='relu'))
model.add(Dropout(0.5)) # 添加Dropout层,丢弃50%的神经元
model.add(Dense(2, activation='softmax'))
model.compile(loss='binary_crossentropy', optimizer='adam', metrics=['binary_accuracy'])
model.summary()

from tensorflow.keras.layers import Conv1D, Dense, MaxPooling1D, Flatten
from tensorflow.keras.regularizers import l2
model = Sequential()
model.add(Conv1D(filters=32, kernel_size=12, input_shape=(train_features.shape[1], 4), kernel_regularizer=l2(0.01)))
model.add(MaxPooling1D(pool_size=4))
model.add(Flatten())
model.add(Dense(16, activation='relu', kernel_regularizer=l2(0.01)))
model.add(Dense(2, activation='softmax'))
model.compile(loss='binary_crossentropy', optimizer='adam', metrics=['binary_accuracy'])
model.summary()
model.compile(loss='binary_crossentropy', optimizer=tf.keras.optimizers.Adam(learning_rate=0.001), metrics=['binary_accuracy'])
history = model.fit(train_features, train_labels, epochs=50, batch_size=64, verbose=1, validation_split=0.25)



再改:



# 导入所需的层和模型
# 导入了TensorFlow Keras库中的一些常用层和模型类:
# Conv1D:一维卷积层,用于处理序列数据。
# Dense:全连接层。
# MaxPooling1D:一维最大池化层。
# Flatten:将多维输入展平为一维。
# Sequential:线性堆叠模型,每一层的输出自动成为下一层的输入
from tensorflow.keras.layers import Conv1D, Dense, MaxPooling1D, Flatten, Dropout, Input
from tensorflow.keras.models import Sequential
from tensorflow.keras.regularizers import l2
# 初始化Sequential模型,这是一种线性堆叠的模型,每一层的输出自动成为下一层的输入
# 初始化了一个Sequential模型对象,表示模型的层是按顺序堆叠的
model = Sequential()
# 添加一维卷积层Conv1D
# filters=32表示有32个卷积核,积核的数量决定了输出特征图的深度。32个卷积核可以提取32种不同的特征。kernel_size=12表示每个卷积核的大小是12,卷积核的大小决定了每次卷积操作覆盖的输入区域。12个碱基的窗口大小可以捕捉到局部的模式。
# input_shape=(train_features.shape[1], 4)指定输入数据的形状。每个输入序列是50个碱基,4维度的one-hot编码(A, C, G, T)
# 其中train_features.shape[1]是特征的数量,4是特征的维度
model.add(Input(shape=(train_features.shape[1], 4)))
model.add(Conv1D(filters=32, kernel_size=12, kernel_regularizer=l2(0.01)))
# 添加一维最大池化层MaxPooling1D
# pool_size=4表示池化窗口的大小是4,即每次取4个连续的数据进行池化操作。池化操作通过取窗口内的最大值来减少特征图的尺寸,从而减少计算量和防止过拟合。
model.add(MaxPooling1D(pool_size=4))
model.add(Dropout(0.5)) # 添加Dropout层,丢弃50%的神经元!!!!
# 再加一层
model.add(Conv1D(filters=64, kernel_size=6, kernel_regularizer=l2(0.01))) # 增加一层卷积层
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.5))
# 添加Flatten层,将多维的输出展平成一维,以便输入到全连接层。卷积层和池化层的输出是多维的,需要展平成一维才能输入到全连接层。
model.add(Flatten())
# 添加全连接层Dense
# units=16表示该层有16个神经元,全连接层的神经元数量决定了该层的输出维度。16个神经元可以学习到16种不同的特征组合。activation='relu'表示使用ReLU激活函数,ReLU激活函数可以引入非线性,使模型能够学习到更复杂的模式。
model.add(Dense(32, activation='relu', kernel_regularizer=l2(0.01)))
# 添加输出层,也是一个全连接层
# units=2表示该层有2个神经元,输出层的神经元数量决定了模型的输出维度,2个神经元对应二分类问题的两个类别。activation='softmax'表示使用softmax激活函数,用于二分类问题,softmax激活函数将输出转换为概率分布,用于多分类或二分类问题。
model.add(Dense(2, activation='softmax'))
# 编译模型,配置其学习过程
# loss='binary_crossentropy'指定损失函数为二元交叉熵,适用于二分类问题
# optimizer='adam'指定优化器为Adam,这是一种自适应学习率的优化算法
# metrics=['binary_accuracy']指定评估模型性能的指标为二元准确率
model.compile(loss='binary_crossentropy', optimizer=tf.keras.optimizers.Adam(learning_rate=0.005), metrics=['binary_accuracy']) #learning_rate=0.001
# 打印模型的概述,包括每层的输出形状和参数数量
model.summary()
from tensorflow.keras.callbacks import EarlyStopping #Keras中的一个模块,包含了许多回调函数,用于在训练过程中执行特定的操作。EarlyStopping是一个回调函数,用于在监控的指标不再改善时提前停止训练,以防止过拟合
early_stop = EarlyStopping(monitor='val_loss',patience=5)
# monitor='val_loss':monitor:指定要监控的指标。在这里,'val_loss'表示监控验证集的损失。'val_loss':验证集的损失值。选择监控验证集的损失是因为它可以反映模型在未见过的数据上的表现,从而帮助防止过拟合。
# patience=10:patience:指定在监控的指标不再改善时,允许训练继续的轮数。在这里,10表示如果验证集的损失在连续2个训练轮次中没有改善,则停止训练。2:具体的耐心值,表示在验证集损失不再改善的情况下,允许训练继续的最大
history = model.fit(train_features, train_labels,
epochs=100, batch_size=32,verbose=1, validation_split=0.25) #原先epochs是50
# model.fit:Keras中用于训练模型的函数
# epochs=50:指定训练的轮次数。每个轮次表示整个训练数据集被用来更新模型参数一次。建议:可以根据模型的训练情况调整这个值。如果模型在较少的轮次内就达到了较好的性能,可以减少这个值,例如 epochs=30
# verbose=0:指定训练过程的日志显示模式。0表示不输出训练过程的日志,1表示输出进度条,2表示每个轮次输出一行日志。建议:在调试和观察训练过程时,可以设置为 verbose=1,以便查看训练进度
再练:



再练:崩了




7,模型优化:

调整类权重
在model.fit中添加class_weight参数:
class_weight = {0: 1., 1: 3.} # 根据数据集的类别分布调整权重
history = model.fit(train_features, train_labels, epochs=50, batch_size=64, verbose=1, validation_split=0.25, class_weight=class_weight)
增加模型复杂度
增加卷积层和全连接层的数量:
from tensorflow.keras.layers import Conv1D, Dense, MaxPooling1D, Flatten, Dropout, Input
from tensorflow.keras.models import Sequential
from tensorflow.keras.regularizers import l2
model = Sequential()
model.add(Input(shape=(train_features.shape[1], 4)))
model.add(Conv1D(filters=32, kernel_size=12, kernel_regularizer=l2(0.01)))
model.add(MaxPooling1D(pool_size=4))
model.add(Dropout(0.5))
model.add(Conv1D(filters=64, kernel_size=6, kernel_regularizer=l2(0.01))) # 增加一层卷积层
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.5))
model.add(Flatten())
model.add(Dense(32, activation='relu', kernel_regularizer=l2(0.01))) # 增加全连接层的神经元数量
model.add(Dropout(0.5))
model.add(Dense(2, activation='softmax'))
model.compile(loss='binary_crossentropy', optimizer='adam', metrics=['binary_accuracy'])
model.summary()
使用学习率调度器
使用学习率调度器动态调整学习率:
from tensorflow.keras.callbacks import ReduceLROnPlateau
reduce_lr = ReduceLROnPlateau(monitor='val_loss', factor=0.2, patience=5, min_lr=0.001)
history = model.fit(train_features, train_labels, epochs=50, batch_size=64, verbose=1, validation_split=0.25, class_weight=class_weight, callbacks=[reduce_lr])
调整之后大致:
from tensorflow.keras.layers import Conv1D, Dense, MaxPooling1D, Flatten, Dropout, Input, BatchNormalization
from tensorflow.keras.models import Sequential
from tensorflow.keras.regularizers import l2
from tensorflow.keras.callbacks import ReduceLROnPlateau
# 初始化Sequential模型
model = Sequential()
# 添加输入层和第一层卷积层
model.add(Input(shape=(train_features.shape[1], 4)))
model.add(Conv1D(filters=64, kernel_size=5, kernel_regularizer=l2(0.01), activation='relu'))
model.add(BatchNormalization())
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.5))
# 添加第二层卷积层
model.add(Conv1D(filters=128, kernel_size=3, kernel_regularizer=l2(0.01), activation='relu'))
model.add(BatchNormalization())
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.5))
# 添加第三层卷积层
model.add(Conv1D(filters=256, kernel_size=3, kernel_regularizer=l2(0.01), activation='relu'))
model.add(BatchNormalization())
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.5))
# 添加Flatten层
model.add(Flatten())
# 添加全连接层
model.add(Dense(128, activation='relu', kernel_regularizer=l2(0.01)))
model.add(Dropout(0.5))
# 添加输出层
model.add(Dense(2, activation='softmax'))
# 编译模型
model.compile(loss='binary_crossentropy', optimizer=tf.keras.optimizers.Adam(learning_rate=0.001), metrics=['binary_accuracy'])
# 打印模型的概述
model.summary()
# 使用学习率调度器
reduce_lr = ReduceLROnPlateau(monitor='val_loss', factor=0.2, patience=5, min_lr=0.0001)
# 训练模型
history = model.fit(train_features, train_labels, epochs=500, batch_size=64, verbose=1, validation_split=0.25, callbacks=[reduce_lr])

调整类权重
在model.fit中添加class_weight参数:
class_weight = {0: 1., 1: 3.} # 根据数据集的类别分布调整权重
history = model.fit(train_features, train_labels, epochs=500, batch_size=64, verbose=1, validation_split=0.25, class_weight=class_weight, callbacks=[reduce_lr])
增加数据增强
使用数据增强技术生成更多的训练数据:
from tensorflow.keras.preprocessing.image import ImageDataGenerator
datagen = ImageDataGenerator(
rotation_range=20,
width_shift_range=0.2,
height_shift_range=0.2,
horizontal_flip=True)
# 使用数据增强生成训练数据
history = model.fit(datagen.flow(train_features, train_labels, batch_size=64),
epochs=500, verbose=1, validation_split=0.25, class_weight=class_weight, callbacks=[reduce_lr])
使用更复杂的模型架构
尝试使用更复杂的模型架构,如ResNet:
from tensorflow.keras.applications import ResNet50
base_model = ResNet50(weights=None, include_top=False, input_shape=(train_features.shape[1], 4, 1))
model = Sequential()
model.add(base_model)
model.add(Flatten())
model.add(Dense(128, activation='relu', kernel_regularizer=l2(0.01)))
model.add(Dropout(0.5))
model.add(Dense(2, activation='softmax'))
model.compile(loss='binary_crossentropy', optimizer=tf.keras.optimizers.Adam(learning_rate=0.001), metrics=['binary_accuracy'])
model.summary()
history = model.fit(train_features, train_labels, epochs=500, batch_size=64, verbose=1, validation_split=0.25, class_weight=class_weight, callbacks=[reduce_lr])
调整超参数
尝试不同的学习率、批量大小和正则化参数:
model.compile(loss='binary_crossentropy', optimizer=tf.keras.optimizers.Adam(learning_rate=0.0005), metrics=['binary_accuracy'])
history = model.fit(train_features, train_labels, epochs=500, batch_size=32, verbose=1, validation_split=0.25, class_weight=class_weight, callbacks=[reduce_lr])
使用交叉验证
使用交叉验证来评估模型的性能:
from sklearn.model_selection import KFold
kf = KFold(n_splits=5)
for train_index, val_index in kf.split(train_features):
X_train, X_val = train_features[train_index], train_features[val_index]
y_train, y_val = train_labels[train_index], train_labels[val_index]
history = model.fit(X_train, y_train, epochs=500, batch_size=64, verbose=1, validation_data=(X_val, y_val), class_weight=class_weight, callbacks=[reduce_lr])
learning_rate参数用于设置初始学习率,而ReduceLROnPlateau回调函数用于在训练过程中动态调整学习率。
具体来说,learning_rate=0.001设置了优化器的初始学习率为0.001,而ReduceLROnPlateau回调函数会在验证损失不再改善时,将学习率乘以factor(即0.2),直到达到min_lr(即0.0001)。——》两者不会冲突
修改后如下:
···
from tensorflow.keras.layers import Conv1D, Dense, MaxPooling1D, Flatten, Dropout, Input, BatchNormalization
from tensorflow.keras.models import Sequential
from tensorflow.keras.regularizers import l2
from tensorflow.keras.callbacks import ReduceLROnPlateau
# 初始化Sequential模型
model = Sequential()
# 添加输入层和第一层卷积层
model.add(Input(shape=(train_features.shape[1], 4)))
model.add(Conv1D(filters=64, kernel_size=5, kernel_regularizer=l2(0.01), activation='relu'))
model.add(BatchNormalization())
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.5))
# 添加第二层卷积层
model.add(Conv1D(filters=128, kernel_size=3, kernel_regularizer=l2(0.01), activation='relu'))
model.add(BatchNormalization())
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.5))
# 添加第三层卷积层
model.add(Conv1D(filters=256, kernel_size=3, kernel_regularizer=l2(0.01), activation='relu'))
model.add(BatchNormalization())
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.5))
# 添加Flatten层
model.add(Flatten())
# 添加全连接层
model.add(Dense(128, activation='relu', kernel_regularizer=l2(0.01)))
model.add(Dropout(0.5))
# 添加输出层
model.add(Dense(2, activation='softmax'))
# 编译模型
model.compile(loss='binary_crossentropy', optimizer=tf.keras.optimizers.Adam(learning_rate=0.001), metrics=['binary_accuracy'])
# 打印模型的概述
model.summary()
# 使用学习率调度器
reduce_lr = ReduceLROnPlateau(monitor='val_loss', factor=0.2, patience=5, min_lr=0.0001)
# 训练模型
history = model.fit(train_features, train_labels, epochs=500, batch_size=64, verbose=1, validation_split=0.25, callbacks=[reduce_lr])
初始学习率设置为0.001,当验证损失在连续5个epoch中没有改善时,学习率将乘以0.2,直到达到最小学习率0.0001。这样可以确保模型在训练过程中动态调整学习率,以便更好地收敛。




from tensorflow.keras.layers import Conv1D, Dense, MaxPooling1D, Flatten, BatchNormalization, Dropout, Input
from tensorflow.keras.models import Sequential
from tensorflow.keras.regularizers import l2
from tensorflow.keras.callbacks import ReduceLROnPlateau
# 初始化Sequential模型
model = Sequential()
# 添加输入层和第一层卷积层
model.add(Input(shape=(train_features.shape[1], 4)))
model.add(Conv1D(filters=64, kernel_size=5, kernel_regularizer=l2(0.01), activation='relu'))
model.add(BatchNormalization())
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.5)) # 增加Dropout率
# 添加第二层卷积层
model.add(Conv1D(filters=128, kernel_size=3, kernel_regularizer=l2(0.01), activation='relu'))
model.add(BatchNormalization())
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.5)) # 增加Dropout率
# 添加第三层卷积层
model.add(Conv1D(filters=256, kernel_size=3, kernel_regularizer=l2(0.01), activation='relu'))
model.add(BatchNormalization())
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.5)) # 增加Dropout率
# 添加Flatten层
model.add(Flatten())
# 添加全连接层
model.add(Dense(128, activation='relu', kernel_regularizer=l2(0.01)))
model.add(Dropout(0.5)) # 增加Dropout率
# 添加输出层
model.add(Dense(2, activation='softmax'))
# 编译模型
model.compile(loss='binary_crossentropy', optimizer=tf.keras.optimizers.Adam(learning_rate=0.001), metrics=['binary_accuracy'])
# 打印模型的概述
model.summary()
# 使用学习率调度器
reduce_lr = ReduceLROnPlateau(monitor='val_loss', factor=0.2, patience=5, min_lr=0.0001) # 调整min_lr
# 训练模型
history = model.fit(train_features, train_labels, epochs=100, batch_size=32, verbose=1, validation_split=0.25, callbacks=[reduce_lr])
使用类权重
如果数据集不平衡,可以通过调整类权重来平衡损失函数对不同类别的关注:
class_weight = {0: 1., 1: 3.} # 根据数据集的类别分布调整权重
history = model.fit(train_features, train_labels, epochs=100, batch_size=32, verbose=1, validation_split=0.25, class_weight=class_weight, callbacks=[reduce_lr])


class_weight = {0: 1., 1: 3.23} # 根据类别比例调整权重
history = model.fit(train_features, train_labels, epochs=100, batch_size=32, verbose=1, validation_split=0.25, class_weight=class_weight, callbacks=[reduce_lr])




实际上class weight需要修改如下;
https://stackoverflow.com/questions/44716150/how-can-i-assign-a-class-weight-in-keras-in-a-simple-way
 修改权重之后的class是:
修改权重之后的class是:
但是需要修改初始学习率:

从原来的0.001,感觉loss下降得有点慢,还是提高初始的学习率
其中调参比较好的一次代码保存在:

大致效果如下:



8,正常分析:
前面都是入学者的瞎炼,现在进行正规分析:
from tensorflow.keras.layers import Conv1D, Dense, MaxPooling1D, Flatten, BatchNormalization, Input
from tensorflow.keras.models import Sequential
from tensorflow.keras.regularizers import l2
model = Sequential()
model.add(Input(shape=(train_features.shape[1], 4)))
model.add(Conv1D(filters=32, kernel_size=20, kernel_regularizer=l2(0.01),activation='relu'))
model.add(BatchNormalization())
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.1))
model.add(Conv1D(filters=128, kernel_size=3, kernel_regularizer=l2(0.01),activation='relu'))
model.add(BatchNormalization())
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.1))
model.add(Conv1D(filters=256, kernel_size=3, kernel_regularizer=l2(0.01), activation='relu'))
model.add(BatchNormalization())
model.add(MaxPooling1D(pool_size=2))
model.add(Dropout(0.1))
model.add(Flatten())
model.add(Dense(128, activation='relu', kernel_regularizer=l2(0.01)))
model.add(Dropout(0.5))
model.add(Dense(2, activation='softmax'))
model.compile(loss='binary_crossentropy', optimizer=tf.keras.optimizers.Adam(learning_rate=0.001), metrics=['binary_accuracy']) #learning_rate=0.001,初始学习率,因为后期的loss下降的太慢
model.summary()

首先我的数据是获取cohesin共定位(overlap)附近的CTCF peak中心然后窗口大小为100bp的序列;
为什么又改为100bp了呢?
之前使用的不是50bp吗?
之前设置的50bp大概是考虑motif大小大概不会超过
(1)首先我们的每一个样本都是50bp的DNA序列(实际上是特定蛋白共定位下的CTCF的peak),然后每个字符都用ACGT的one-hot编码,
所以实际上我们输入的是一维的序列数据,而且channel是4(对应ACGT)



所以我们import的模块都是1D的卷积模块
(2)输入层只给出每个样本的shape,无需整个数据集

model.add(Input(shape=(train_features.shape[1], 4)))
(3)对于一维卷积层:
我们需要注意的架构超参数就那么几个:

参考:
https://keras.io/api/layers/convolution_layers/convolution1d/
https://www.tensorflow.org/api_docs/python/tf/keras/layers/Conv1D


kernel_size:卷积核filter的大小(2D一般是方块nxn,1D就是窗口大小)————然后一般的motif长度大概在10~30bp左右范围内,我们取20bp,卷积核可以直接覆盖一个完整的motif,从而有效地提取与该motif相关的特征;
但是保险起见为了看到更大的范围以及规律,我这里使用40bp,后面还是改回了20bp。

filter卷积核的数目:我们输入的DNA序列是4 channel的,而依据CNN架构,不同的卷积核会专注于处理不同的特征信息,1个卷积核对应1个feature map,卷积核的数目对应了这一层输出数据的channel数目————个人建议是随着网络逐层加深之后数目也加深,2的幂次为好——暂时取32
步长stride:控制卷积核移动的步长,1D默认为1
填充padding:1D不需要,为valid

卷积核正则化器:l2正则化,防止过拟合
参考:https://keras-zh.readthedocs.io/regularizers/
model.add(Conv1D(filters=32, kernel_size=20, kernel_regularizer=l2(0.01),activation='relu'))
(5)卷积之后是批归一化:
但是问题就在于批归一化放在激活函数前还是后,最原始的论文是放在前面,但是现在反正没有定数,
按照实际效果好坏来处理前后位置
参考:https://www.tensorflow.org/api_docs/python/tf/keras/layers/BatchNormalization
https://keras-cn.readthedocs.io/en/latest/legacy/layers/normalization_layer/
 BatchNormalization:添加批量归一化层,用于加速训练和稳定模型。
BatchNormalization:添加批量归一化层,用于加速训练和稳定模型。
model.add(BatchNormalization()) #暂时放在激活函数后面
至于batch normalization与pooling的位置,以pooling为先
https://stackoverflow.com/questions/42015156/the-order-of-pooling-and-normalization-layer-in-convnet

(4)卷积之后就是池化pooling:
池化窗口的大小是4,即每次取4个连续的数据进行池化操作
model.add(MaxPooling1D(pool_size=2)) #原先是4,后面该为2

然后注意这里的strides是none,默认是pool_size,意味着每个pool其实分析的元素不会有重叠
所以如果我pool size设置为4的话,那么最后获得的pooling之后的shape为11-4+1/4=2,也就是只有2个长度的feature map,这简直就没法接着往下卷积了,因为序列数据已经被霍霍完了,没必要将网络做深了。

(其实当我不知道网络结构变成什么样,下一步要提供什么样的参数的时候,可以用model_summary看一下网络输出结构是怎么样的,再然后考虑是否要进一步再叠一层CNN)
(6)随机丢弃一些神经元,防止过拟合:
model.add(Dropout(0.3))

反正放在哪里还是没有定论,放在全连接层前面比较多,但是因为是随机正则化机制,所以放在哪里都行。
卷积层也可以放,一般建议是卷积层丢drop少一点,在全连接层丢多一点。
总之input——》conv——》激活——》pooling——》batchnorm——》dropout——》全连接层(激活——》dropout)


只要是绘制logomarker:





详细解释其中的每一行代码、函数、参数,并对所使用的函数举1-2个简单例子来解释说明
训练:


对于非常小或非常不平衡的数据集,随机分割很有可能完全从其中一个分割中消除一个类。
——》
另外有:




https://stackoverflow.com/questions/34842405/parameter-stratify-from-method-train-test-split-scikit-learn
# 将数据集拆分为训练集和测试集,使用 stratify 参数保持比例不变
train_features, test_features, train_labels, test_labels = train_test_split(
input_features, input_labels, test_size=0.25, random_state=2025, stratify=input_labels)
——》
2025.1.22更新:
上述代码实际上使用的是tensorflow框架,我本来是想用pytorch的,所以暂时先做个新手测验,所以代码写的就这样;
另外前面写的只是草稿部分,现在来看有很多缺漏以及说错了的地方:
比如说weight就是train以及test的怎么平衡,其实不是一行代码就行的;
另外网络架构上稍微变了变,然后数据上也稍微变化了一下,就是一些超参数;
输入数据的编码也变化了;
然后对于卷积核的理解我是这样理解的:
虽然本质上说我们用ChIP的数据来训练,按理来说有了这个数据我们早就知道哪里是蛋白质结合哪里不是了,那为什么我们还要训练1个CNN呢,当然也可以使用其他架构,我认为原因在于:
1,首先是能运用到train之外的数据集上:
同个细胞系就算了,因为正如前面所言有了数据;
不同细胞系呢,可以参考,当然现在学了生物不敢说那么绝对,但是可以迁移学习;
当然理论上来说不同细胞系基因组难道不是一样吗,所以如果只是给个DNA序列,我们又不做DNA-seq也就是WGS、WES,会有什么区别吗,值得思考
2,我们可以调整前提条件:
比如说多个组学数据筛选下的正负样本,这样学到的东西就不是简单的TFBS二分类问题了,
比如说我用ATAC数据去筛选,我用TAD边界去筛选,我考虑链的正反方向性等等,或者共定位,或者多个motif异二聚体异多聚体等等,当然这就不是模型的作用了,重点应该是上游数据的处理上
3,理论上,训练完毕之后的CNN,我提取出第一层卷积层(这里我捉摸着可能是因为第一层可视化之后最有生物学意义,当然大家都可以自己去可视化不同的卷积层),然后选取权重最大的那几个kernel卷积核,就是best kernel,然后我再对这个核在正样本上做一个cross correlation,然后相关性最高的位点,取这些正样本的子序列做一个平均,类似于motif效果;但是我的窗口可以开很大,所以40bp我就看40bp motif,多少就多少。
整理修改之后的CNN在PR之后仓库